INTRODUCCIÓN
Campylobacter spp., es una de las principales bacterias causantes de gastroenteritis en los países industrializados. En Paraguay, según un estudio realizado en el año 2019 por Huber et al. 1, se lo reporta como el segundo enteropatógeno bacteriano asociado a síndrome diarreico agudo después del grupo de E.coli diarreogénicas entre las cuales se destacan E. coli entero patogénica, E. coli enteroagregativa entre otras, presentando una prevalencia del 13.4%.
La detección de este patógeno en nuestro país, se encuentra muy limitada, debido a la necesidad de equipamiento alternativo e insumos costosos, esto último podría generar un gran sesgo en el momento de conocer la prevalencia e implicancia real de este patógeno en salud pública 1,2.
La infección bacteriana causada por Campylobacter spp., se denomina campylobacteriosis y se presenta generalmente en pacientes pediátricos, pero afecta a todo el rango etario 3. Las especies comúnmente involucradas en estas patologías son Campylobacter jejuni y Campylobacter coli. Varios estudios han demostrado que Campylobacter jejuni es el principal causante de la infección en humanos en un 80% al 95% seguido de Campylobacter coli4-6. Se adquiere normalmente por el consumo de carne de aves de corral mal cocida en su mayor proporción, así como de leche no pasteurizada y agua contaminada 3-5,7.
El síndrome diarreico causado por Campylobacter spp., es autolimitado, sin embargo, existen drogas antimicrobianas de elección para el tratamiento como macrólidos y fluoroquinolonas que se utilizan en caso de que el paciente se encuentre con alguna inmunosupresión o la infección se prolongue o complique 8-11. Además se han registrado estudios que hablan de síndromes extraintestinales relacionados con este patógeno como ser el Síndrome de Guillain Barre, artritis reactiva, bacteriemias, entre otros 12-15.
Este patógeno presenta una serie de factores de virulencia responsables del proceso de fisiopatogenia como ser los genes flaA, cadF, racR y dnaJ responsables de la expresión de la adherencia y colonización; los genes virB, ciaB y pldA que participan en la invasión, los genes ctdB, ctdC, citotoxinas de distensión, que evitan en las células eucariotas que entren en mitosis, conduciendo a la muerte celular y wlaN, indicado como el gen involucrado en la expresión de mimics de gangliosidos en el síndrome de Guillain Barre 16-19.
Tradicionalmente, el diagnóstico y confirmación de las especies de Campylobacter se ha realizado mediante cultivo bacteriológico y pruebas bioquímicas 20,21; sin embargo, en los últimos años, se han implementado técnicas moleculares como la reacción en cadena de la polimerasa (PCR) para la detección directa del patógeno a partir de muestras diarreicas, así como diferenciación de especies, detección de factores de virulencia 22,23 y técnicas de genotipificación 24-26.
Este estudio, tiene como objetivo caracterizar el perfil de virulencia y la resistencia antimicrobiana en cepas de Campylobacter spp., aislados de pacientes con síndrome diarreico agudo en el Laboratorio Central de Salud Pública, periodo 2020 - 2021.
MATERIALES Y MÉTODOS
El tipo de estudio utilizado fue retrospectivo de corte transversal, tomando como periodo de estudio los años 2020 y 2021. El muestreo realizado fue no probabilístico de casos consecutivos, como criterio de inclusión se consideró todas las cepas aisladas de muestras que se remitieron con la ficha epidemiológica para el estudio de Enfermedades Diarreicas Agudas (EDAS) en el periodo de estudio de 2020-2021. Se sometieron a estudio 168 cepas de Campylobacter spp.
La recolección de datos fue realizada mediante un cuestionario donde fueron registrados los resultados laboratoriales obtenidos de la caracterización del patógeno como especie de Campylobacter spp., los genes que codifican los perfiles de virulencia (virB, ciaB, pldA, flaA, cadF, racR, dnaJ, wlaN, ctdB, ctdC) y resistencia antimicrobiana del estudio de las cepas aisladas.
Las muestras remitidas fueron sembradas directamente en un agar específico Skirrow modificado preparado a partir de agar brucella (Oxoid Ltd., Ogdensburg, NY), suplemento FBP (sulfato ferroso, metasulfito de sodio, piruvato de sodio y sangre) y una mezcla antibiótica (vancomicina, trimetoprim, cefalotina y polimixina B), y posteriormente incubadas por 48 horas en un ambiente de microaerofilia (10% O2, 5% de CO2 y N2 para balance) a una temperatura de 42°C.
Las colonias sospechosas fueron identificadas de acuerdo a la característica de las colonias, tinción de gram pruebas fenotípicas como oxidasa, indoxil acetato e hidrolisis de hipurato. Las especies de Campylobacter jejuni y Campylobacter coli, fueron confirmadas mediante la Reacción en Cadena de la Polimerasa (PCR) de punto final según el protocolo de Farace et al. y Persson et al 27,28 (Tabla 1).
Detección del perfil de virulencia. Para la detección de los genes de virulencia se investigaron los genes, ctdC, ctdB, cadF, virB, flaA, pldA, racR, wlaN, ciaB y dnaJ, mediante la Reacción en Cadena de la Polimerasa (PCR) de punto final según el protocolo de Talukder et al 2008 29. (Tabla 1).
Resistencia antimicrobiana. Se determinó por el método de concentración inhibitoria mínima (CIM) en agar, utilizando tiras de test de CIM marca LIOFILCHEM Diagnostic, de origen italiano para los siguientes antimicrobianos: eritromicina, ciprofloxacina y tetraciclina y agar Mueller-Hinton enriquecido con 5% de sangre ovina. La interpretación de los resultados se realizó según los puntos de cortes de manual del Instituto de Estandarización de Laboratorios Clínicos (CLSI) M45-A2 30.
Tabla 1 Secuencias de primers utilizados.
| GEN | PARTIDORES | SECUENCIAS 5 3 | PESO (pb) |
|---|---|---|---|
| jun | jun1 | CATCTTCCCTAGTCAAGCCT | 773 |
| jun2 | AAGATATGGCACTAGCAAGAC | ||
| col | col1 | AGGCAAGGGAGCCTTTAATC | 364 |
| col2 | TATCCCTATCTACAAATTCGC | ||
| flaA | flaA 664 | AATAAAAATGCTCATAAAAACAGGTG | 855 |
| flaA 1494 | TACCGAACCAATGTCTGCTCTGATT | ||
| cdtB | cdtB-113 | CAGAAAGCAAATGGAGTGTT | 370 |
| cdtB-713 | AGCTAAAAGCGGTGGAGTAT | ||
| cdtC | cdtC-192 | CGATGAGTTAAAACAAAAAGATA | 620 |
| cdtC-351 | TTGGCATTATAGAAAATACAGTT | ||
| racR | racR-25 | GATGATCCTGACTTTG | 182 |
| racR-593 | TCTCCTATTTTTACCC | ||
| dnaJ | dnaJ-299 | AAGGCTTTGGCTCATC | 584 |
| dnaJ-1003 | CTTTTTGTTCATCGTT | ||
| virB11 | virB-232 | TCTTGTGAGTTGCCTTACCCCTTTT | 720 |
| virB-701 | CCTGCGTGTCCTGTGTTATTTACCC | ||
| ciaB | ciaB-403 | TTTTTATCAGTCCTTA | 494 |
| ciaB-1373 | TTTCGGTATCATTAGC | ||
| pldA | pldA-84 | AAGCTTATGCGTTTTT | 986 |
| pldA-981 | TATAAGGCTTTCTCCA | ||
| wlaN | wlaN-DL 39 | TTAAGAGCAAGATATGAAGGTG | 913 |
| wlaN-DL 41 | CCATTTGAATTGATATTTTTG | ||
| cadF | cadF-F2B | TTGAAGGTAATTTAGATATG | 400 |
| cadF-R1B | CTAATACCTAAAGTTGAAAC |
Analisis estadístico. El análisis fue realizado utilizando el programa estadistico EpiInfo version 7.7, para analizar tanto los datos sociodemográficos y resultados laboratoriales. Las variables continuas se expresaron como promedios y desviación estandar, y las variables categóricas se consignaron como frecuencias y porcentajes (%).
El trabajo estuvo sujeto a todas las cuestiones éticas de trabajos en poblaciones y sujeto a las normas del Comité de Ética del Laboratorio de Salud Pública del Ministerio de Salud Pública y Bienestar Social.
RESULTADOS
De las 168 cepas de Campylobacter spp., sometidas a estudio, se observó un predominio de cepas de la especie Campylobacter jejuni con un porcentaje de 95% (160/168 ) y Campylobacter coli con un 5 % ( 8/168 ).
Respecto a los factores de virulencia estudiados se obtuvo un mayor porcentaje de flaA (96%) y ctdC (94%), seguido de cadF (86.9%), ctdB (85%), dnaJ (79.7%), racC (71%) y en menor porcentaje se detectó pldA (49%), ciaB (21%), wlaN (13%) y virB (2%) (Tabla 2).
Tabla 2 Factores de virulencia en cepas de Campylobacter spp. Estudiadas.
| Factores de virulencia | Frecuencia | Porcentaje | IC 95% |
| cadF | 146 | 86.90% | 80.85% - 91.61% |
| ciaB | 36 | 21.43% | 15.48% - 28.41% |
| ctdB | 143 | 85.12% | 78.82% - 90.13% |
| ctdC | 158 | 94.05% | 89.33% - 97.11% |
| dnaJ | 134 | 79.76% | 72.88% - 85.56% |
| flaA | 161 | 95.83% | 90.02% - 97.41% |
| pldA | 83 | 49.40% | 41.62% - 57.21% |
| racC | 120 | 71.43% | 63.96% - 78.12% |
| virB | 4 | 2.38% | 0.65% - 5.98% |
| wlaN | 22 | 13.10% | 8.39% - 19.15% |
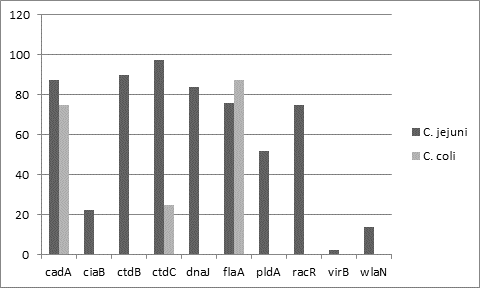
En cuanto al perfil de virulencia con relacion a las especies de Campylobacter estudiadas (Gráfico 1), se pudo observar que las cepas de Campylobacter jejuni poseen mayor diversidad en la distribucion de factores de virulencia con respecto a Campylobacter coli. Las cepas de Campylobacter jejuni presentaron diferentes perfiles de presentacion de los factores de virulencia estudiados. El perfil de factores mas observado en las cepas en este estudio fueron las que presentaron el perfil flaA, ctdC, cadF, ctdB, dnaJ, racR. En las cepas de Campylobacter coli se pudo detectar solo la presencia de los genes flaA, cadF y ctdC.
En cuanto a la resistencia antimicrobiana observada en cepas de Campylobacter spp., en este estudio se pudo observar una sensibilidad del 100% a la droga antimicrobiana eritromicina, 51 % de resistencia a ciprofloxacina y un 6 % de resistencia a tetraciclina (Tabla 3).
Tabla 3 Resistencia antimicrobiana para Campylobacter spp.
| Antimicrobiano | Rango CIM (mg/L) | Puntos de corte CIM (mg/L)* | Frecuencia (%) | ||||
| S | I | R | S | I | R | ||
| Eritromicina | 0.016 - 256 | ≤ 8 | 16 | ≥ 32 | 168 (100) | - | 0 (0) |
| Tetraciclina | 0.016 - 256 | ≤ 4 | 8 | ≥ 16 | 162 (96.43) | - | 6 (3.57) |
| Ciprofloxacina | 0.002 - 32 | ≤ 1 | 2 | ≥ 4 | 82 (48.8) | - | 86 (51.2) |
*Valores de punto de corte de CLSI CIM. CLSI, Instituto de Estándares Clínicos y de Laboratorio. CIM, concentración inhibitoria mínima; S, sensible; I, intermedio; R, resistente.
DISCUSIÓN
Las cepas analizadas en este estudio, fueron revividas en agar sangre directamente de cepas conservadas a -80ºC. No se pudo analizar cepas de años anteriores ya que no estaban viables en el momento de recuperación, por lo tanto, se procedió a trabajar con cepas de los últimos años. Sería importante revisar los procedimientos de conservación de cepas para este tipo de bacterias. Por otro lado, todas las detecciones realizadas en este estudio fueron realizadas por la técnica de reacción en cadena de la polimerasa, lo cual genera un proceso tedioso, ya que la mayoría de los genes detectados tenían temperaturas de alineamiento diferente. La implementación de PCR tiempo real para la detección de estos genes u otra técnica molecular como secuenciación de genoma completo presentarían una mayor sensibilidad, especificidad, rapidez y una mayor información genética.
Las cepas de Campylobacter spp., estudiadas en su mayoría correspondieron a la especie Campylobacter jejuni (95%). La frecuencia alta de Campylobacter jejuni encontrada respecto a Campylobacter coli, concuerda con estudios realizados en el país como el de Huber et al.1, Orrego et al.2. Asi mismo, es lo reportado en la mayoría de otros estudios como Nejabat et al.31, Notario et al.32, Gonzalez Hein et al.33, Ghorbanalizadgan et al. (34, entre otros.
Se pudo observar, una diversidad de genes de virulencia en cepas de las especies de Campylobacter estudiadas, mayor presentación de dichos genes en las cepas de Campylobacter jejuni, con diferentes perfiles de genes de virulencia entre cepas, no asi, en las cepas de Campylobacter coli, donde, se pudo detectar la presencia solamente de 3 genes de virulencia, como, el gen flaA y los genes cadF y ctdC.
La frecuencia de los factores de virulencia estudiados, destacaron, una mayor proporción detectada de genes de la toxina de distención citolítica (CDT) entre estos, los genes cdtB y cdtC, los cuales están involucrados en la unión con la célula huesped e ingresó al interior de la misma hasta el compartimiento del nucleo activando una actividad DNASA llevando a la apoptosis de la célula. El rol crucial de estas toxinas dentro de la patología intestinal dando lugar a una infección gastrointestinal prolongada y con grave inflamación de la mucosa 35. La alta frecuencia de estos factores de virulencia también se destaca en el estudio de González Hein et al. 33, Bang et al. 36; Data et al. 37; Rozne et al. 38; Den et al. 39, no así en el estudio realizado por Rizal et al. 40, donde reportó un 28% de cdtB.
Se detectó entre un 80% y 70% de los genes dnaJ y racR , siendo estos, genes de proteinas que juegan un papel importante en la termoregulación y termotolerancia. Estos genes no fueron detectados en cepas de Campylobacter coli. Lo mismo fue reflejado por otros estudios como el de Tarkut et al.41, Bardon et al.48.
El gen flaA que codifica la proteina flagelina, factor de virulencia importante dentro del estudio de este patógeno y el gen cadF, ambos involucrados en la adhesión celular. Se pudieron detectar estos genes en ambas especies de Campylobacter estudiadas, en mayor proporción el gen flaA seguido de el gen cadF. El gen ciaB involucrado en la translocación celular se detectó en menor proporción que los genes citados anteriormente. Ghorbanalizadgan et al.34, en su estudio realizado detectó el 100% de los genes flaA, cadF y ciaB.
En menor frecuencia fueron detectados los genes involucrados en la invasión celular como pldA, responsable de la síntesis de la fosfolipasa de membrana importante en la colonización y virB, codifica al plasmido pVIR que ayuda en la penetración e invasión. La baja proporción de detección del gen virB, tambien fue reportado por González Hein et al.33.
En este estudio, fue detectado un 13% del gen wlaN involucrado con otros genes en la producción de B-1,3 galactosyltransferasa, responsable de la biosíntesis de GM1 , asociados con el riesgo de desarrollar Sindrome de Guillain Barre (GBS). Se observó reporte similar en el estudio de Kordinas et al.42, otros estudios presentaron frecuencias mas altas de detección del gen como el de Casabonne et al.43 y Datta et al.37 con un 20 a 25 %. En cambio, un estudio realizado por Nejabat et al.31, reportó una frecuencia de 4,6% en un estudio realizado.
Es importante destacar la detección de este gen, ya que, el gen wlaN solo fue detectado en cepas de Campylobacter jejuni. Teniendo en cuenta que es la especie aislada con mayor frecuencia en el pais y la relevancia de la presencia del gen como factor de riesgo para el desarrollo de ciertas patologías, podría resultar interesante a nivel epidemiológico y clínico.
La resistencia antimicrobiana observada en este estudio, destacó, una resistencia del 51% a ciprofloxacina, 6% a tetraciclina y no presentó resistencia a eritromicina. En nuestro país, según estudios realizados años anteriores, la resistencia a quinolonas se mantiene. Como lo reportado por Orrego et al.2, en el año 2014, con un 49% de resistencias a quinolonas y Huber et al.1, en el 2019, reportó un 48% de resistencia a ciprofloxacina. Estudios como el de García et al.44 y otros destacan que la resistencia a ciprofloxacina está presente en cepas aisladas principalmente de pacientes pediátricos en los cuales no se utilizan quinolonas como tratamiento empírico de diarrea, por tanto se podría suponer que la infección ha sido provocada por bacterias previamente resistentes asociado el aumento de la resistencia a las quinolonas con la introducción de estos fármacos en la industria avícola, principalmente enrofloxacina, cuyo metabolito activo es ciprofloxacina45-47. Sería importante el reporte y la vigilancia a nivel de la industria avícola en nuestro país. Tambien se reportaron resistencia a tetraciclina en un 28%. Hasta el momento no se han reportado en el país datos importantes de resistencia a eritromicina que es la droga de elección al tratamiento. Resistencias similares a ciprofloxacina presentan estudios como el de Notario et al.32, Bardon et al.48.
CONCLUSION
La identificación de los factores de virulencia nos permite reconocer la importancia de los mismos para la mejor comprensión de la patogénesis en ciertos procesos infecciosos extraintestinales que se presentan o se podrían presentar después de un cuadro diarreico agudo por este patógeno.
El conocimiento de la resistencia antimicrobiana es la clave para el desarrollo de terapias para el tratamiento de las infecciones, así como mejorar nuestra comprensión de la patogénesis. Dado al patrón de resistencia antimicrobiana de Campylobacter spp., que ha presentado en este estudio, es importante realizar la vigilancia de su susceptibilidad in vitro, especialmente para aquellos antimicrobianos utilizados de manera empírica en el tratamiento de la enterocolitis bacteriana y en aquellas cepas aisladas de pacientes con enfermedades invasoras o inmunocomprometidos.