INTRODUCCIÓN
La evolución de la resistencia a los antimicrobianos ha puesto en riesgo el valor de las drogas de amplio espectro disponibles para el tratamiento de las enfermedades infecciosas. Desde la aparición de cepas productoras de carbapenemasas1, que por lo general van acompañadas de otros mecanismos de resistencia, se describen las bacterias resistentes a múltiples antibióticos. A esta problemática se suma la pobre producción farmacéutica de nuevos antibióticos, haciendo cada vez más difícil lograr el éxito terapéutico contra las infecciones.
En nuestro país, las primeras cepas portadoras de carbapenemasa del tipo KPC fueron confirmadas en Enterobacterales en el año 2009 y desde ese entonces se han registrado brotes en distintos centros asistenciales de todo el país2. Las carbapenemasas del tipo metalobetalactamasa genotipo NDM, fueron confirmadas en Asunción y Central en Acinetobacter pitti en el año 20123, siendo Paraguay el primer país en confirmar la presencia de este mecanismo de resistencia en Acinetobacter sp. en la región4 y actualmente son las carbapenemasas más frecuentes en Enterobacterales.
Toda esta problemática de carácter mundial, ha promovido la consideración de la utilización de drogas en desuso como las polimixinas para el tratamiento terapéutico, las que por muchos años han sido relegadas por su toxicidad para el uso clínico5.
Actualmente, sólo dos polimixinas, polimixina E (colistina) y polimixina B, están disponibles comercialmente. La colistina ha resurgido como tratamiento de última línea a mediados de la década de los 90 para el tratamiento de infecciones severas ocasionadas por bacilos gramnegativos resistentes a múltiples antimicrobianos5.
Desafortunadamente, el aumento de la utilización de este antibiótico trajo como consecuencia el incremento de la resistencia global a la colistina.
Los mecanismos de resistencia a esta droga son de varios tipos: intrínsecos, que son propios de los microorganismos; y los adquiridos, que pueden ser cromosómicos y plasmídicos5.
Desde la publicación del mecanismo plasmídico de resistencia a polimixinas en Enterobacterales, en el año 2015, cuyo gen codificante es el mcr-1 (Mobile Colistin Resistance), se llevaron a cabo estudios retrospectivos en animales, alimentos, humanos y medioambiente, los cuales arrojaron innumerables resultados, entre ellos, que el mismo se halla ampliamente diseminado en varios países del mundo, en muestras de diversos orígenes y en diferentes especies de Enterobacterales6.
Estudios detallados del gen, han demostrado la gran cantidad de variantes y alelos de mcr-1 circulantes7.
En Paraguay, la confirmación de mcr-1 fue realizada y publicada en agosto de 2016 en cepas de Klebsiella pneumoniae aisladas de muestras urinarias de pacientes ambulatorios de sexo femenino; luego fue confirmado en otros miembros de Enterobacterales aislados tanto de pacientes hospitalizados como de la comunidad. Además, en los aislamientos se observó la presencia de resistencia acompañante, incluso a carbapenémicos por producción de carbapenemasas8.
El gran desafío sin duda representa el análisis de los aislamientos bacterianos a fin de obtener el perfil de susceptibilidad a la colistina.
Los laboratorios de microbiología tropiezan con serios problemas a la hora de llevar a cabo las pruebas de sensibilidad, debido a la dificultad que las mismas representan, puesto que no todos los laboratorios cuentan con los insumos ni la complejidad para realizar las metodologías de referencia, como pruebas de dilución en caldo.
Varios investigadores han publicado resultados de diferentes metodologías de evaluación de la sensibilidad a colistina comparando con las de dilución en caldo; y los resultados obtenidos fueron muy variables, sin embargo, la mayoría de ellos coinciden en que los datos proporcionados por pruebas que no sean de referencia pueden conducir a una terapia inadecuada con colistina, lo cual puede conllevar al fracaso terapéutico en el tratamiento de la enfermedad infecciosa9-12.
JUSTIFICACION
La resistencia a los antimicrobianos genera grandes preocupaciones a nivel mundial. Los tratamientos de las enfermedades infecciosas se ven dificultados por la diseminación de gérmenes resistentes a múltiples drogas. En el caso de los Enterobacterales, resistentes a los antibióticos de amplio espectro como los carbapenemes, han suscitado la necesidad de recurrir a opciones antiguas de tratamiento, como las polimixinas.
Dentro de esta familia, la colistina es la más utilizada en el tratamiento de infecciones ocasionadas por bacilos gramnegativos resistentes a múltiples antibióticos
Por lo antes expuesto, el objetivo de esta revisión consiste en proporcionar información acerca de los mecanismos de acción de la droga y mecanismos de resistencia de los Enterobacterales a la colistina; así como las metodologías utilizadas para las pruebas de susceptibilidad a este antimicrobiano y las recomendaciones de los distintos centros de referencia sobre la utilización y los criterios de interpretación de cada una de ellas.
MARCO TEÓRICO
Las polimixinas, que incluyen seis compuestos (A, B, C, D, E y M), son un grupo de antimicrobianos bactericidas con características estructurales particulares, con estructuras de oligopéptidos cíclicos no ribosómicos, de los cuales solo polimixina B y colistina (polimixina E) son los únicos disponibles en el mercado5.
El descubrimiento de la colistina fue en el año 1947 por un grupo de investigadores japoneses (Koyama y cols), y el nombre fue otorgado por los mismos, por tratarse de un metabolito secundario de la bacteria grampositiva del suelo Paenibacillus polymyxa subsp. colistinus13.
Históricamente, la colistina se utilizó por primera vez en la década de 1950 como formulación intravenosa. En 1959, la Food and Drug Administration de los EE. UU. aprobó la colistina como agente antimicrobiano contra bacilos gramnegativos por su actividad bactericida para el tratamiento de varios tipos de infecciones, incluida la diarrea infecciosa y las infecciones del tracto urinario. Además, las polimixinas se han administrado durante varias décadas en formulaciones tópicas para infecciones oculares y de oído como así también para la descontaminación intestinal selectiva. También se utilizaron para combatir infecciones causadas por gramnegativos multirresistentes14,15. En medicina veterinaria, colistina y polimixina B han sido utilizados durante décadas con fines profilácticos y terapéuticos16.
La colistina es un agente activo contra patógenos gramnegativos aerobios, que frecuentemente representan la fuente principal de infecciones potencialmente mortales, como Pseudomonas aeruginosa resistente a carbapenémicos, Acinetobacter baumannii, Klebsiella pneumoniae, Escherichia coli y otras enterobacterias. Sin embargo, algunas especies bacterianas patógenas o potencialmente patógenas, como: Serratia marcescens, Proteus spp., Providencia spp., Morganella morganii, Vibrio cholerae, Brucella sp., Campylobacter sp., Legionella sp, Chromobacterium sp., Neisseria spp., Edwardsiella spp., algunas especies del género Aeromonas, además de Burkholderia cepacia y cocos gramnegativos anaeróbicos, poseen una resistencia intrínseca a la colistina(15, 16).
En medicina humana, para el tratamiento de infecciones causadas por bacilos gramnegativos, se encuentran disponibles dos formas de colistina: sulfato de colistina (SC), de uso oral y tópico; y metanosulfonato de colistina (CMS) o colistimetato de sodio (CMS), que es una sal de sodio de colistina cargada negativamente, es un profármaco inactivo utilizado para formulaciones parenterales y de nebulización, ya que es menos tóxico que el sulfato de colistina. Entre las dos formas disponibles, el sulfato de colistina es la única aprobada para su uso en la producción porcina en algunos países para el control de infecciones causadas por enterobacterias, en particular Escherichia coli y Salmonella spp.(17, 18).
Con relación a la polimixina B, aunque presenta un amplio espectro de actividad, principalmente contra bacilos gramnegativos, cocos grampositivos como Staphylococcus aureus19, Streptococcus gordonii, Streptococcus agalactiae, así como contra bacterias anaerobias facultativas como Listeria monocytogenes20, su uso clínico fue abandonado a mediados de la década de 1970 debido a los efectos adversos notificados, principalmente nefrotoxicidad y neurotoxicidad, sumado a esto el descubrimiento y la aprobación de nuevos y eficaces antimicrobianos. Sin embargo, permanecieron en la práctica clínica para pacientes que sufrían de fibrosis quística (FQ) debido a infecciones pulmonares por Pseudomonas spp. y en soluciones tópicas con otros agentes antimicrobianos para el tratamiento de infecciones del oído u ojo13,16,17.
A mediados de la década de 1990, las polimixinas habían resurgido como un tratamiento de último recurso contra bacilos gramnegativos multirresistentes, debido a la aparición de superbacterias gramnegativas multirresistentes, en particular Pseudomonas aeruginosa, Acinetobacter baumannii y Klebsiella pneumoniae, que son resistentes a todos los demás antibióticos disponibles, además de la falta de nuevos antimicrobianos disponibles para tratar estas bacterias aisladas de infecciones15,21.
Desafortunadamente, el uso excesivo e incorrecto de este antimicrobiano en humanos y animales, ha llevado a la aparición global de patógenos resistentes a la colistina. Sin embargo, el desarrollo de bacterias resistentes también puede ocurrir sin acompañamiento de ninguna exposición a la droga, dejando a los médicos con las manos vacías para tratar pacientes con infecciones por bacterias multirresistentes22,23. De hecho, las polimixinas ahora juegan un papel fundamental principalmente contra las infecciones por bacterias gramnegativas potencialmente mortales, ya que son una de las pocas, y en ocasiones únicas, opciones de tratamiento y que tienen actividad contra bacilos gramnegativos multirresistentes15.
Mecanismo de acción antimicrobiana de las polimixinas
Como resultado de la similitud estructural entre la colistina y la polimixina B, se ha sugerido que comparten los mismos mecanismos de acción23,24. El mecanismo antibacteriano exacto por el cual la colistina ejerce su acción bactericida aún no ha sido bien dilucidado15. Es principalmente activa contra bacilos gramnegativos debido a la presencia en éstos de lipopolisacáridos (LPS) en la pared celular25. Por lo tanto, comprender la arquitectura de la membrana externa de los bacilos gramnegativos es crucial para descifrar el mecanismo o los mecanismos de acción de la colistina.
Una de las principales funciones de la membrana externa es ser una barrera de permeabilidad frente a diversos agentes nocivos, incluyendo diferentes antimicrobianos26. El papel protector de la membrana externa se relaciona principalmente con la presencia de LPS en la superficie de la célula que dificulta la penetración de antibióticos hidrófobos y/o antibióticos de moléculas grandes27. La estructura de los LPS comprende tres dominios: la cadena de antígeno O, un polisacárido central y un lípido A conservado que actúa como ancla hidrofóbica en la membrana externa16. El blanco principal de la colistina es el LPS de la membrana externa donde ejerce su acción antibacteriana a través de la interacción directa con el componente lípido A del LPS18,26.
Las cadenas de hidrocarburos saturados del lípido A son encerradas juntas dentro de la membrana por fuerzas de Van der Waals, mientras que el magnesio divalente (Mg+2) y cationes de calcio (Ca+2) asociados con el lípido A funcionan como puentes adyacentes a las moléculas de LPS, estabilizando así las moléculas. La función de la membrana externa se acentúa aún más a través de la presencia de la alta carga negativa transportada por el lípido A, restos de éster fosfórico, además del fosfato y grupos carboxilato dentro del núcleo y azúcares del antígeno O26.
Acción de la colistina contra bacterias gramnegativas
El mecanismo de acción más importante de la colistina sobre las bacterias, es el de ejercer su acción bactericida al alterar las membranas externas e internas a través de una vía, denominada la “vía de adopción autopromocionada", considerando su naturaleza anfipática y que es fundamental para la absorción de la molécula de colistina a través de la membrana externa15. En este modelo, la fusión inicial de la colistina con la membrana bacteriana se produce a través de interacciones electrostáticas entre el diaminobutírico catiónico, residuos de ácidos de colistina y grupos de fosfato aniónico del LPS de la membrana externa de la bacteria. Así, la colistina desplaza competitivamente los cationes divalentes Mg+2 y Ca+2, de los grupos fosfatos cargados negativamente, desestabilizando las moléculas del LPS y debilitando la membrana, permitiendo así la captación de colistina. Después de eso, la colistina se adhiere al componente lípido A del LPS, que conduce a un trastorno de la membrana externa de la bacteria28,29.
Cabe destacar que la afinidad de la colistina por los LPS es al menos tres veces mayor que su afinidad por los cationes divalentes30. Esto conduce a un mecanismo de acción del tipo detergente que implica un aumento en la permeabilidad de la envoltura celular, seguida de la fuga de contenido de la célula y, posteriormente, la colistina inserta sus regiones hidrofóbicas (cola de ácido graso y aminoácidos en las posiciones 6 y 7) a través de estas grietas en la membrana externa, dando como resultado la captación auto-promovida18, que conduce a una lisis de la membrana, fuga de contenido periplásmico y citoplasmático y, en última instancia, muerte celular. Este proceso es independiente de la captación de colistina en la célula15,20,21,27,28,29.
Los cinco (Figura 1)mecanismos diferentes de actividad antibacteriana de la colistina son18:
Actividad de colistina antibacteriana directa: la fusión inicial de colistina con la membrana bacteriana se produce a través de interacciones electrostáticas entre el ácido diaminobutírico catiónico, los residuos de colistina y el resto de fosfato aniónico del LPS en la membrana, por lo tanto, los grupos de fosfato aniónico en la membrana externa. Así se logra interrumpir las membranas externas e internas bacterianas conduciendo a la lisis celular;
Actividad de la colistina como anti-endotoxina: la porción del lípido A de LPS representa una endotoxina en bacterias gramnegativas. Así, la colistina inhibe la actividad endotoxina del lípido. Al unirse a las moléculas de LPS y neutralizarlas, de este modo suprime la inducción de choque mediante la liberación de citocinas como el factor de necrosis tumoral alfa (TNF-α) y la interleucina 8 (IL-8);
Vía de contacto vesícula-vesícula: la colistina se une a vesículas de fosfolípidos aniónicos después de atravesar la membrana externa, y conduce a la fusión de la hoja interna de la membrana externa con la hoja externa de la membrana citoplásmica, lo que lleva a la pérdida de fosfolípidos y muerte celular;
Vía de muerte del radical hidroxilo: la colistina actúa a través de la producción de especies reactivas de oxígeno (ROS), esto se conoce como reacción de Fenton, causando daño al ADN, lípidos y proteínas, y termina con la muerte celular; y
Inhibición de las enzimas respiratorias: la actividad antibacteriana de la colistina se produce mediante la inhibición de las enzimas respiratorias vitales.
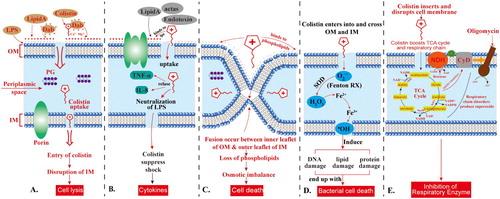
Fuente: El Gawad et al (2020) (5)
Figura 1 Acción de la colistina sobre la membrana de las bacterias gramnegativas.
Acción de la colistina contra bacterias grampositivas
Generalmente, las polimixinas muestran una actividad reducida contra las bacterias grampositivas ya que no se adhieren favorablemente al ácido lipoteicoico que se encuentra en la membrana citoplasmática. Sin embargo, como se mencionó anteriormente, la polimixina B tiene un amplio espectro de actividad, principalmente contra gramnegativos, pero también se ha demostrado que es eficaz contra bacterias grampositivas19,22.
De hecho, el mecanismo de acción de la polimixina B es no basado en un efecto detergente o lítico sobre la membrana bacteriana, como se ha informado previamente para los gramnegativos. Se ha demostrado que la polimixina B induce la aposición de vesículas aniónicas, además de la formación de contactos funcionales vesícula-vesícula que permiten un intercambio rápido y selectivo de fosfolípidos, particularmente entre las monocapas externas de las vesículas31. Cabe destacar que la inserción de grupos funcionales hidrofóbicos a la estructura de la polimixina B a través de la acilación de la amina de la cadena lateral de Dab1 (ácido diaminobutírico) con diferentes ácidos grasos pueden aumentar su capacidad para atravesar las membranas y conferir interacciones favorables con el ácido lipoteicoico de las membranas bacterianas grampositivas que conduce a una mayor actividad antibacteriana32.
Muy recientemente, Yu y colaboradores20 han revelado que la colistina puede inducir la acumulación de ROS en bacterias grampositivas, tales como Paenibacillus polymyxa C12, Bacillus subtilis WB800 y Paenibacillus polymyxa ATCC842, que llevan al estrés oxidativo independientemente de la lisis de la membrana celular, lo que resulta en muerte celular. La generación del estrés oxidativo está relacionada con la estimulación secuenciada del ciclo del ácido tricarboxílico (TCA) y la cadena respiratoria, seguida por el agotamiento transitorio de NADH. De hecho, el mecanismo detallado de formación de estrés oxidativo por la colistina aún no está completamente aclarado.
A fin de tratar de comprender mejor el complejo mecanismo de acción de estos antimicrobianos, es importante conocer la estructura química de la droga.
Estructura química de las polimixinas
Ambas polimixinas utilizadas en tratamiento (polimixina B y colistina) poseen la misma estructura básica, bastante compleja y de gran tamaño. Las únicas diferencias entre ellas están en los aminoácidos componentes (Figura 2).
La característica fundamental de estas drogas es la de ser anfipáticas (un extremo hidrofílico y otro hidrofóbico), capaces de distribuirse bien tanto en ambientes polares como no polares; o sea, tanto en agua como a través de las membranas lipídicas de los eucariotas32.
La colistina contiene una mezcla de D y L-aminoácidos dispuestos como un anillo de heptapéptido cíclico con una cadena lateral de tripéptido. La cadena lateral está unida covalentemente a un ácido graso a través de un grupo acilo32.
Al hablar del tratamiento con colistina, la droga de uso endovenoso utilizada es en realidad la colistina metanosulfonato de sodio. Los tratamientos locales (colirios y nebulizaciones) son realizados con sulfato de colistina.
La colistina metanosulfonato de sodio es una pro-droga inactiva, se prepara a partir de colistina por reacción del grupo amino de los residuos Dab con formaldehído seguido de bisulfito de sodio (para uso endovenoso)33.
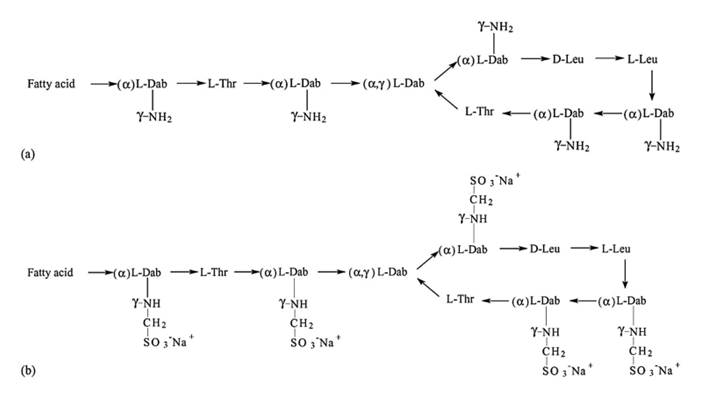
Fuente: Li J. et al. 2005.34
Figura 2 Estructura química de colistina (a), y colistina metanosulfonato de sodio (b).
En cuanto a la estabilidad, la colistina es resistente a la pepsina, eripsina y tripsina, pancreática, no así a la lipasa. En un ambiente seco, la colistina sulfato puede permanecer estable a temperatura ambiente hasta 12 meses.
La colistina metanosulfonato de sodio (pro-droga) se hidroliza en medios acuosos, y forma una mezcla compleja de derivados, con el potencial de producir hasta 32 productos diferentes, entre ellos a la colistina34.
Vías de administración y presentaciones de colistina
El sulfato de colistina se puede administrar por vía oral en forma de tabletas y jarabe para la descontaminación selectiva del tracto digestivo (sin absorción) y tópicamente para el tratamiento de infecciones bacterianas de la piel28.
El colistinmetanosulfonato de sodio es un profármaco, menos tóxico que el sulfato de colistina. Tiene diferentes vías de administración, parenteral (incluida la intravenosa) e intramuscular, pero también es posible la administración intratecal o intraventricular28. La inyección intramuscular se utiliza raramente en la práctica clínica porque puede ser muy dolorosa localmente y también porque su absorción es variable. Tanto el sulfato de colistina como la colistina metasulfonato de sodio pueden administrarse mediante inhalación en terapia con aerosol, pero hay una mayor frecuencia de broncoconstricción con el sulfato de colistina35.
La administración de colistina metasulfonato de sodio por inhalación y por las rutas intratecal e intraventricular permite concentraciones mucho más altas en el líquido pulmonar y el líquido cefalorraquídeo, respectivamente, que las observadas con la administración sistémica. Además, esas vías de administración conducen a una exposición plasmática insignificante y son menos tóxicas (en particular, menos nefrotóxicas)36.
Mecanismos de resistencia
Los mecanismos de resistencia a las polimixinas en los bacilos gramnegativos son complejos y hasta la fecha no se han logrado entender completamente. En general, estos microorganismos pueden desarrollar resistencia a las polimixinas a través de mecanismos intrínsecos, de mutación o de adaptación, además de la resistencia adquirida horizontalmente mediada por el gen mcr-1 y sus variantes6,37.
Los principales mecanismos de resistencia a las polimixinas se pueden resumir como sigue29:
Modificaciones del resto de LPS mediante la adición de grupos catiónicos al LPS;
Mutaciones que conducen a la pérdida del LPS;
Mutaciones de la porina y sobreexpresión de sistemas de bombeo de flujo;
Sobreproducción de polisacárido capsular (CPS) en algunos bacilos gramnegativos que ocultan los sitios de unión de polimixinas y la liberación de polimixinas que atrapan CPS; y
Inactivación enzimática de colistina.
Resistencia a la colistina codificada cromosómicamente
Algunos investigadores han publicado la resistencia cruzada entre colistina y polimixina B28.
Si bien los mecanismos subyacentes de resistencia son comunes entre los bacilos gramnegativos, los mismos pueden diferir entre diferentes especies. La siguiente tabla 1 expone algunos ejemplos de las principales características de mecanismos de resistencia y modificaciones asociados a las polimixinas29.
Tabla 1 Ejemplos de características de resistencia y modificaciones asociados a polimixinas.
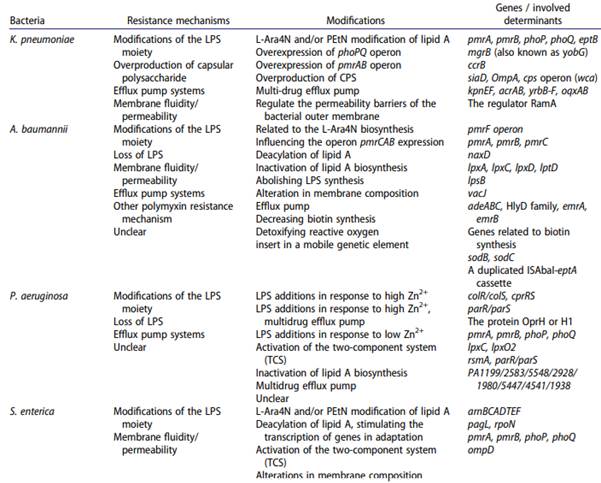
Fuente: El-Sayed A. et al. (2020) (5).
Ejemplo de resistencia a colistina codificada cromosómicamente en Enterobacterales resistente a múltiples drogas.
La resistencia cromosómica a esta droga en Klebsiella pneumoniae, no siempre está dada por el mismo mecanismo, sino puede estar mediada por una o varias estrategias combinadas. A continuación, se describen los más frecuentemente observados:
La modificación del lípido A mediante mutaciones en los genes pmrA, pmrB o phoQ, que a su vez, regulan positivamente los sistemas PhoP / PhoQ y PmrA / PmrB (38), lo que lleva a la adición de L-Ara4N o PEtN al LPS15;
Las mutaciones en el gen mgrB (un regulador de retroalimentación negativa del sistema PhoPQ) que codifica la proteína MgrB (proteína transmembrana reguladora compuesta por 47 aminoácidos. El gen mgrB se regula al alza tras la activación del sistema PhoP), prototipo observado con mayor frecuencia en cepas de Klebsiella pneumoniae portador de carbapenemasa tipo KPC (Klebsiella pneumoniae carbapenemasa)39;
Las mutaciones del gen mgrB por inserción de secuencias (tipo IS5, IS1F, ISKpn13, ISKpn14, IS10R) o mutaciones puntuales representan la causa principal de resistencia a la polimixina en las cepas clínicas de Klebsiella pneumoniae39,40.
Las mutaciones en el operón ccrB (regulación de la resistencia a colistina). Este operón codifica dos proteínas, a saber; la proteína reguladora CrrA y la proteína cinasa sensorial CrrB. En Klebsiella pneumoniae, se ha reportado que la inactivación del gen crrB conduce a la sobreexpresión del operón pmrAB que a su vez, conduce a la activación del operón pmrHFIJKLM, genes pmrC y pmrE, que termina con la adición de L -Ara4N y pEtN al lípido A de LPS40.
La sobreproducción de los polisacáridos capsulares aniónicos de superficie (CPS) (29) que representan una barrera protectora contra las polimixinas, donde la regulación positiva de los genes de biosíntesis capsular, a saber; siaD, OmpA y cpsoperon (wca), dificulta la unión de polimixinas con lípido A22 al atrapar polimixinas.
La acilación del lípido A en Klebsiella pneumoniae podría estar regulada por lpxM (msbB o waaN), donde su inactivación puede conducir a una falta de modificación de L-Ara4N41.
La heterorresistencia
Este término se emplea para referirse a las poblaciones bacterianas que contienen una mayoría de bacterias inhibidas en concentraciones por debajo del punto de corte de sensibilidad. Las CIM de las polimixinas en estas bacterias se encuentran ´por debajo de 2 ug/mL; sin embargo, las subpoblaciones (en torno a 1 por cada 103 -107) pueden sobrevivir en presencia de polimixinas a una concentración mayor a 2 ug/mL, lo que a su vez da como resultado la amplificación de las subpoblaciones resistentes en presencia de polimixina sola y, en consecuencia, desarrolla resistencia a la polimixina. Sin embargo, hasta la fecha no se cuenta con un método estandarizado para la detección de este mecanismo de resistencia. Otros autores publicaron que la detección de heterorresistencia se puede realizar mediante ensayos de microdilución, en los que las bacterias heterorresistentes muestran el fenómeno de pocillos salteados (pocillos sin crecimiento, aunque el crecimiento todavía se produce a concentraciones más altas)42.
En este contexto el antimicrobiano actuaría como agente de selección, eliminando la población más sensible y permitiendo la supervivencia de la fracción mutante, lo que podría deberse también a un fenómeno de persistencia. La exposición a concentraciones del fármaco insuficientes podría representar un factor de riesgo para el enriquecimiento in vivo de la población resistente, y esto podría relacionarse con fracaso terapéutico. Los aislamientos de Acinetobacter baumannii de pacientes tratados previamente con colistina presentan un mayor grado de heterorresistencia. Esta es en parte la base teórica para el uso en terapia combinada de colistina, en particular en infecciones graves15.
Algunos investigadores sugirieron que el fenómeno de heterorresistencia a polimixinas se debía a mutaciones en genes cromosómicos, como los genes de biosíntesis del lípido A (lpxA, lpxC, lpxD) o la adición de L-ara4N, que a su vez, afectan al regulador de respuesta PhoP. Además de ello, han demostrado que el mecanismo de heterorresistencia confiere un alto nivel de resistencia (CMI> 128 mg/L)43,44. Sin embargo, otros investigadores publicaron que a un pH ligeramente ácido5,8, tiene lugar una fuerte inducción de la adición de L-Ara4N y PEtN, que a su vez, contribuye a la resistencia a la polimixina45; además, que la resistencia a la polimixina se atribuye a las condiciones ácidas de crecimiento de las bacterias a través de la actividad transcripcional de varios genes, que incluyen; yjdB, pmrC, pagB y pmrF22.
Bombas de eflujo
El sistema de bomba de eflujo también puede estar involucrado en el mecanismo de resistencia a las polimixinas46. Comúnmente, la activación de estas bombas de eflujo da como resultado un aumento en la resistencia a diferentes antibióticos al mismo tiempo, incluida la colistina. En diversas especies bacterianas, se han descrito diferentes bombas de eflujo, como las proteínas Sap (péptidos antimicrobianos sensibles), BrlR, el complejo AcrAB-TolC o KpnEF. Las proteínas Sap están compuestas por cinco proteínas diferentes codificadas por el operón sapABCDF38. En varios organismos, las bombas de eflujo AcrAB-TolC, KpnEF, MtrC-MtrD-MtrE, VexAB, RosAB y NorM se han designado para dar tolerancia a la polimixina B.
En Klebsiella pneumoniae, también fue descrita la resistencia a la polimixina mediante la activación de las bombas de flujo AcrAB29.
Resistencia a polimixinas mediadas por plásmidos
El ultimo mecanismo de resistencia a polimixinas, es el mediado por transferencia horizontal de un gen mcr (resistencia móvil a colistina) transmitido por plásmidos, y se ha convertido en una causa importante de la diseminación de la resistencia a las polimixinas entre varios bacilos gramnegativos5,17,42.
Este mecanismo plasmídico de resistencia, publicado recién a finales del año 2015, pudo ser rastreado mediante estudios retrospectivos realizados con bacilos gramnegativos en los años 80 en China y 2005 en Francia, aislados de aves de corral y terneros, respectivamente47.
Los primeros reportes de portación de mcr-1 fueron dados en colonias de Escherichia coli aislados de animales6, y desde entonces fue reportada la diseminación en otros miembros de Enterobacterales: Klebsiella pneumoniae, Enterobacter cloacae, Klebsiella aerogenes (antiguamente Enterobacter aerogenes), Cronobacter sakazakii, Salmonella enterica, Raoultella ornithinolytica, Citrobacter freundii, Citrobacter braakii, Shigella sonnei, Kluyvera ascorbata, y Moraxella spp.42,48 en más de 30 países de los 5 continentes49; a partir de muestras de varios orígenes: animales, humanos, alimentos, medios acuáticos, aguas residuales de hospitales50.
Ante la detección de este mecanismo de resistencia en varios países de la Región de las Américas51-54, en aislados tanto de animales como de humanos, la Organización Panamericana de la Salud (OPS) / Organización Mundial de la Salud (OMS) ha publicado la Alerta Epidemiologia en fecha 10 de junio de 2016, con el fin de que los Estados Miembros se ocupen en implementar y mantener la capacidad para detectar, prevenir y controlar la transmisión de microorganismos con resistencia transferible a colistina. Además de llamarlos a tomar medidas que conduzcan a prohibir el uso indiscriminado de estos antimicrobianos para profilaxis y como promotores de crecimiento en animales destinados al consumo humano 55.
En nuestro país las primeras confirmaciones de mcr-1 se dieron en agosto de 2016, en cepas de Klebsiella pneumoniae aisladas de muestras urinarias de pacientes ambulatorios (56). Luego, fue detectado mcr-1 en otros miembros de Enterobacterales, aislados de pacientes ambulatorios y hospitalizados, y asociado con otros genes de resistencia como beta-lactamasa de espectro extendido, resistencia a fluorquinolonas mediada por plásmidos y portación de carbapenemasas8.
Básicamente mcr-1 es una enzima transferasa de lípido A de fosfoetanolamina, que pertenece a la superfamilia de fosfatasa alcalina “YhjW / YjdB / YijP”; y el mecanismo por el cual puede mediar la resistencia a la colistina es el siguiente: mcr-1 codifica una PEtN (fosfoetanolamina) transferasa que conduce a la adición de un resto PEtN al lípido A de LPS, ello hace que aumente las cargas catiónicas en el LPS y, en consecuencia, disminuye la unión de colistina a LPS 49,57. Esta acción se atribuye a la estructura química de la PEtN transferasa. La región N-terminal de la PEtN transferasa se inserta en la membrana interna, mientras que el dominio de sulfatasa catalítica C-terminal se encuentra en el espacio periplásmico. Este último proceso es responsable de la transferencia de un resto pEtN de su donante fisiológico fosfatidiletanolamina de LPS42.
En la actualidad se conocen 22 variantes genéticas funcionales de mcr-1: mcr-1.1, mcr-1.2, mcr-1.3, mcr-1.4, mcr-1.5, mcr-1.6, mcr-1.7, mcr-1.8, mcr-1.9, mcr-1.10, mcr-1.11, mcr-1.12, mcr-1.13, mcr-1.14 y mcr-1.15, mientras que las otras variantes genéticas de mcr-1.16, mcr-1.17, mcr-1.18, mcr-1.19, mcr-1.20, mcr-1.21 y mcr-1.22 Estas variantes se diferencian de mcr-1 en uno o algunos aminoácidos. Por tanto, todos comparten una identidad elevada de nucleótidos y aminoácidos (∼99%) y, por tanto, confieren un efecto similar sobre la resistencia a la colistina50,58,59.
Los determinantes de la resistencia transferible a la colistina se han extendido, y hoy se conocen varios alelos nuevos de mcr-1. Hasta ahora, se han reportado nueve alelos mcr incluyendo mcr-1 a saber; mcr-2 (1617 pb), mcr-3 (1626 pb), mcr-4 (1626 pb), mcr-5 (1644 pb), mcr-6 (1617 pb), mcr-7 (1620 pb), mcr-8 (1698 pb), y el mcr-9 detectado más recientemente (2661 pb)59-65.
Métodos para las pruebas de susceptibilidad
A pesar del uso clínico prolongado (décadas), el método óptimo para las pruebas de susceptibilidad a la polimixina aún permanece sin definir. Sin embargo, con el creciente aumento de bacilos gramnegativos resistentes a múltiples antimicrobianos, con el consecuente aumento del uso de colistina, impulsaron a la comunidad científica a desarrollar varios métodos para determinar la susceptibilidad a los mismos. El éxito terapéutico depende de los resultados de pruebas de sensibilidad confiables, por lo que los laboratorios de microbiología enfrentan a diario este gran desafío9-12.
Las dificultades en la realización de las pruebas de susceptibilidad a estas drogas son diversas, entre las que se pueden citar16:
La mala difusión de las polimixinas en el agar por el gran tamaño de sus moléculas,
Las propiedades catiónicas inherentes de las polimixinas,
La aparición de heterorresistencia a las polimixinas en muchas especies,
La falta de un método de referencia confiable que pueda permitir comparaciones reales de los valores comerciales,
La compleja farmacocinética (PK) y farmacodinámica (PD), y,
La escasez de datos que correlacionen los datos de Concentración Inhibitoria mínima (MIC), la concentración del fármaco y los resultados clínicos, que han dificultado el establecimiento de puntos de corte clínicos66,67.
Las guías y los criterios de interpretación de las pruebas de susceptibilidad a las polimixinas han sufrido varias modificaciones a lo largo de los años, como por ejemplo, cambios sucesivos en los puntos de corte interpretativos, cambios en las composiciones de los drogas y componentes utilizados, etc.
Las pruebas de difusión de discos no están recomendadas por ninguna de las guías más utilizadas para la realización de las pruebas, y con igual criterio, el método de epsilométrico o de tira de gradiente68.
En el año 2016, tanto Clinical and Laboratory Standards Institute (CLSI) como European Commitee on Antimicrobial Susceptibility testing (EUCAST) han recomendado que la metodología de referencia para pruebas de sensibilidad a las polimixinas sería la microdilución en caldo69.
Ya para el año 2017, CLSI ha publicado los valores de corte epidemiológicos (ECV) para un grupo de Enterobacterales: Escherichia coli, Klebsiella pneumoniae, Raoutella ornithinolytica, Enterobacter cloacae y Enterobacter aerogenes, pero sin categorizar a estos microorganismos como Sensible, Intermedio y Resistente sino como salvajes o no salvajes70.
Tabla 2 Criterios de interpretación de Valor de Corte Epidemiológico (ECV) de colistina en Enterobacterias.
| ECV Colistina CIM (ug/mL) | Aplica para: | |
|---|---|---|
| Salvaje | No salvaje | |
| ≤ 2 | ≥ 4 | Klebsiella aerogenes, Enterobacter cloacae, Escherichia coli, Klebsiella pneumoniae y Raoultella ornithinolytica |
Fuente: Clinical and Laboratory Standards Institute (2017) (70).
Sin embargo, EUCAST permitía categorizar (tabla 3)a los Enterobacterales, por lo que el Laboratorio de Referencia Regional (LRR) ha recomendado la utilización de esta guía para la interpretación de los puntos de corte71.
Tabla 3 Criterios de interpretación de Puntos de Corte de colistina en Enterobacterales.
| Sensible | Resistente | |
| Enterobacterales | ≤ 2 ug/mL | ≥ 4 ug/mL |
Fuente: EUCAST (2021)72.
La determinación de la CIM a colistina está asociada con diversos problemas metodológicos que conllevan a errores no aceptables respecto al método de referencia, por lo que el grupo de trabajo CLSI-EUCAST en el año 2016 acuerdan que método de referencia para Enterobacteriaceae, Pseudomonas aeruginosa y Acinetobacter spp. es el de microdilución en caldo (único método para evaluar la sensibilidad a colistin), según estándar ISO (20776-1) llevado a cabo bajo las siguientes condiciones73:
Usar caldo Mueller-Hinton ajustado a cationes.
No se deben incluir aditivos en ninguna parte del proceso de ensayo (en particular, polisorbato-80 u otro tensioactivo).
Las policubetas deben ser de poliestireno sin tratamiento.
Se debe utilizar sulfato de polimixina (no el derivado metanosulfonato de colistina que es la pro-droga inactiva).
En el año 2020 el CLSI introdujo varias novedades en la tabla 2A del M100, 30 th ed.; de donde se eliminaron los valores de corte epidemiológicos y se reemplazaron por puntos de corte para Enterobacterales con los criterios de interpretación para colistina y polimixina B (I ≤ 2 y R ≥ 4)74, los cuales siguen vigentes en la tabla 2A del M100, 31 th ed del año 2021 (75). Además, se incluyeron informaciones iimportantes sobre la dosificación de las drogas74,75:
- Colistina (metanosulfonato) debe administrarse con una dosis de carga y dosis máximas ajustadas por funcionalidad renal.
- Polimixina-B debe administrarse con una dosis de carga y dosis máximas recomendadas.
- Cuando colistina o polimixina-B se administran por vía sistémica, es probable que ninguna sea efectiva para neumonía.
- La microdilución en caldo, la elución de discos y la placa de tamizaje con colistina son métodos aceptados para evaluar la sensibilidad de colistina.
- Para polimixina-B el único método aceptado es la microdilución en caldo.
- La difusión con discos y las tiras de gradiente no deben ser utilizados, por los errores graves que arrojan.
Sin embargo, el EUCAST no ha introducido cambios en los criterios de interpretación de los puntos de corte para las polimixinas. El Laboratorio de Referencia Regional (LRR), del Instituto Nacional de Enfermedades Infecciosas (INEI-ANLIS) Dr. Carlos Malbrán de Argentina; adoptó estos criterios para el informe (Tabla 4) de Enterobacterales y las polimixinas (S ≤ 2 ug/mL, R ≥ 4ug/mL), mencionado más arriba72.
La Tabla 4 expone las diferencias entre ambas guías, en relación a las polimixinas y Enterobacterales (72, 75).
Tabla 4 Diferencia entre los Puntos de Corte para colistina en Enterobacterales. Año 2021.
| Puntos de Corte (2021) | CLSI | EUCAST | |||||
| S | I | R | S | I | R | ||
| Enterobacterales | - | ≤ 2 | ≥ 4 | ≤ 2 | - | ≥ 4 | |
Además, el LRR ha estandarizado para los laboratorios de Microbiología de la región otras alternativas para llevar a cabo las pruebas de susceptibilidad a colistina76.
Tabla 5 Metodologías para evaluar la sensibilidad a colistina en Enterobacterales.
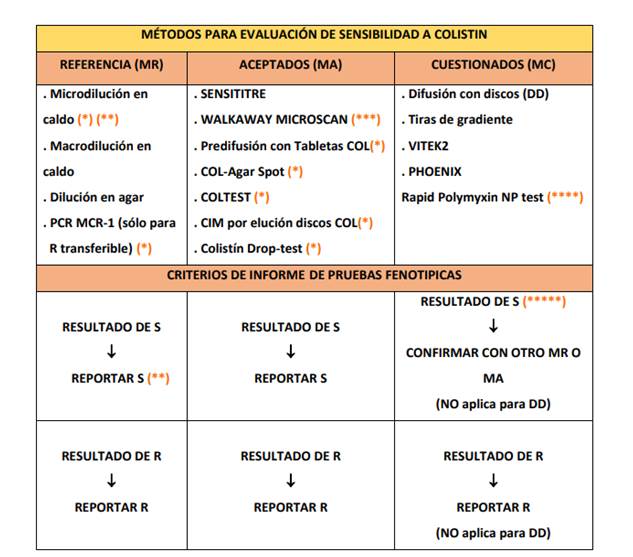
Fuente: INEI-ANLIS. Servicio Antimicrobianos (2020)76.
De esta manera los laboratorios de Microbiología Clínica poseen más opciones para evaluar la sensibilidad a colistina, que en el caso de pacientes internados, suele ser imperioso contar con este antibiótico.
Según las recomendaciones del LRR, los métodos MR (tabla 5) son los propuestos como de referencia para los Laboratorios Nacionales de Referencia; y los MA (tabla 5) son los que están disponibles comercialmente y pueden ser puestos a punto por los laboratorios de Microbiología. Ambos pueden ser considerados seguros a la hora de predecir la sensibilidad de colistina76.
En el caso de contar con los métodos cuestionados, los resultados resistentes pueden ser informados como tal, pero los sensibles deben ser corroborados por los métodos recomendados, tales como aceptados o de referencia.
La prueba colorimétrica rápida (Figura 3), diseñada por Nordmann y colaboradores en el año 201677, representa una alternativa económica y rápida para los laboratorios de Microbiología. La misma consiste en evaluar el crecimiento en una solución glucosada con el agregado de colistina a concentración final de 3,75 µg/mL. La interpretación se basa en el cambio de color del indicador, a consecuencia del cambio de pH, siendo, además, de fácil interpretación. Sin embargo, el LRR aconseja su uso solo para predecir resistencia a colistina en Enterobacterales, en caso de arrojar resultado de sensible, recomienda corroborar el resultado con pruebas más confiables.
Otra metodología, totalmente desaconsejada es la de difusión de discos, sin embargo, existen laboratorios que aún la siguen realizando. Cabe destacar que, por esta metodología, los puntos de corte e interpretación para colistina y Enterobacterales, fueron retirados en el año 198678.

Fuente: Rodríguez C. et al. (2017)79.
Figura 3 Resultados de prueba rápida de detección de resistencia a colistina en Enterobacterias.
CONCLUSIONES
La resistencia a los antimicrobianos representa en la actualidad uno de los principales problemas de salud pública a nivel mundial, tanto en pacientes hospitalizados por infecciones graves, como también pacientes de la comunidad.
El tratamiento de las enfermedades infecciosas es hoy día un reto para los médicos, debido a la gran propagación de la resistencia bacteriana, incluso a drogas de amplio espectro, y a la cada vez menor disponibilidad de nuevas drogas eficaces disponibles.
Las polimixinas se han utilizado durante varias décadas como agentes bactericidas contra los bacilos gramnegativos de difícil tratamiento. Como resultado de sus efectos tóxicos adversos, su uso se ha limitado o incluso detenido. Sin embargo, se han reintroducido en la práctica clínica como último recurso contra los bacilos gramnegativos resistentes a múltiples antimicrobianos. Los mecanismos de acción de estos antimicrobianos guardan estrecha relación con sus particularidades estructurales, ya que se trata de moléculas de gran tamaño y de características anfipáticas, las mismas actúan alterando las membranas bacterianas externas e internas, lo que da como resultado final la muerte bacteriana.
De igual modo, el mecanismo de resistencia bacteriana a estos antimicrobianos es variado y complejo, pudiendo ser cromosómico y extracromosómico o plasmídico.
El mecanismo cromosómico de resistencia principal es el de modificación de la membrana externa de la bacteria, con la consecuente acción del tipo detergente, ocasionando el aumento en la permeabilidad de la envoltura externa, seguido de fuga del contenido de la célula, resultando así en la muerte bacteriana.
Además, la heterorresistencia a las polimixinas es una amenaza emergente atribuida a la exposición bacteriana a dosis subóptimas de la droga, y representa una fuente potencial de selección de resistencia a la colistina.
El ultimo mecanismo de resistencia identificado; mecanismo plasmídico mediado por el gen mcr-1; se ha identificado en miembros de Enterobacterales. Esto ha puesto en relieve la posibilidad de perder la eficacia a la colistina, por la rápida propagación que implica este fenotipo. Hasta el momento, se han identificado 22 nuevas variantes genéticas de mcr-1 en diferentes países del mundo, lo que indica la posibilidad de una evolución continua. Además, se han reportado nuevas variantes alélicas de mcr-1: mcr-2, mcr-3, mcr-4, mcr-5, mcr-6, mcr-7, mcr-8 y el más reciente detectado mcr-9.
La compleja estructura de la molécula de colistina, y los diversos mecanismos involucrados en la resistencia bacteriana, se reflejan en el gran desafío que deben enfrentar a diario los laboratorios de Microbiología Clínica a la hora de realizar las pruebas de susceptibilidad y detección de la resistencia a esta droga.
El LRR ha puesto a consideración las metodologías:
De referencia: Las que deben ser efectuadas en los Laboratorios de Referencia Nacionales; y son las pruebas de dilución (microdilución en caldo, macrodilución en caldo, dilución en agar) y la detección plasmídica del gen mcr.
Aceptadas: Que pueden ser estandarizadas en los laboratorios de Microbiología Clínica, a fin de otorgar resultados confiables para el tratamiento de pacientes con enfermedades infecciosas. Estas pruebas son: Elución de discos de colistin de 10 ug, COL-agar-spot, Colistin Drop Test, predifusión con tabletas de colistina, COLTEST, y los automatizados como Sensititre y Walkaway.
Cuestionados: cuya predicción de los resultados resistentes son confiables, sin embargo, los resultados sensibles deben ser confirmados por una de las 2 metodologías anteriores.
Entre las pruebas tenemos a: NP-Test (método colorimétrico rápido), automatizados: Vitek y Pheonix.
Los métodos que impliquen difusión en agar, no deben ser considerados a la hora de estudio de la sensibilidad a esta droga.















