INTRODUCCIÓN
El procesamiento de extracción de ADN consiste en su almacenamiento y purificación del resto de los componentes celulares y se fundamenta en las características fisicoquímicas de este ácido nucleico 1. En ese contexto, el uso de apropiados métodos de muestreo, tipo de tejido y la utilización de protocolos viables de extracción del ADN son aspectos críticos en estudios basados en PCR. Los métodos de extracción de ADN deben ser simples, reproducibles, económicos y no contaminantes 2.
A inicios de 1990, la limitante del tiempo de los métodos de extracción se vio zanjada al introducir al mercado combos o kits de extracción que utilizan el mismo principio de matrices orgánicas a pequeña escala o matrices inorgánicas compactas cargadas positivamente con capacidad de retener ADN (varios microgramos) y separarlo del resto de las biomoléculas, obteniendo un extracto libre de inhibidores 3. Sin embargo, tanto los métodos de extracción de ADN tradicionales y comerciales (combos o kits) incluyen tres etapas principales: lisis celular, eliminación de proteínas y/o lípidos, y precipitación y redisolución del ADN. Para tal fin, es imprescindible contar con muestras que permitan aislar ADN en gran cantidad y calidad, siendo escogidos por excelencia para esta labor la sangre o el tejido4.
En los estudios moleculares se necesario estandarizar los métodos de extracción de ADN que garanticen su sencillez y eficacia. Dentro de este contexto, el presente trabajo evaluó dos métodos de extracción de ADN genómico (ADNg) en base a los protocolos Chelex y de Proteinasa K a partir de sangre fresca, sangre impregnada en papel filtro y pelos procedentes de animales de la especie ovina con el propósito de determinar el método que reúne las mejores condiciones de aplicabilidad y eficiencia para la cuantificación de ácidos nucleicos y su amplificación posterior utilizando la reacción en cadena de la polimerasa (PCR).
MATERIALES Y MÉTODOS
Animales
Se seleccionaron cinco ovinos de una granja dedicada a la cría de animales para autoconsumo, localizada en la ciudad de Asunción, departamento Central, Paraguay (-25.25180 latitud sur, -57.54914 longitud oeste). De cada animal se obtuvieron muestras de sangre fresca, pelo y sangre depositada en papel filtro, dando un total de 30 unidades observacionales5. La muestra de sangre fue extraída mediante punción de la vena yugular, y depositada en tubos con EDTA, inmediatamente se realizó la refrigeración del mismo para su posterior envío al laboratorio. Las muestras de pelo se obtuvieron de manera aleatoria de la cara, procurando extraer el bulbo piloso, aproximadamente 50 pelos por ovino. Por último, la sangre fue extraída mediante la punción en una cantidad mínima suficiente para ser depositada como una gota en el papel filtro S&S 903 que permite colectar las muestras en campo y transportarlas a temperatura ambiente a largas distancias.
Extracción de ADN
El ADNg, fue extraído de muestras de sangre impregnada en papel filtro, pelo y sangre fresca con EDTA (previa descongelación de esta última muestra).
Método resina quelante Chelex(r) 100
Se utilizó la técnica para muestras en papel filtro descripta por Del Puerto6 impregnando la gota de sangre en papel filtro S&S 903. Se corta el trozo de papel que contiene la sangre y se coloca en un tubo de 1.5 ml con 1 ml de agua bidestilada, dejando reposar por 10 min a temperatura ambiente; se centrifuga por 3 min a 13 000 rpm, se descarta el sobrenadante, y se añade 150 µl de Chelex(r) 100 al 5% de la alícuota de stock. Luego se incuba a 56 °C por 30 min, agitando con vortex por 10 s, hirviendo 10 min. Se agita nuevamente por 10 s y se centrifuga por 3 min a 14 000 rpm. El sobrenadante conteniendo el ADN estuvo listo como plantilla para la PCR, almacenándose a -20 °C.
En el caso de la muestra con sangre fresca, se utilizó la técnica descripta por Ascencio5. Se mezcla 50 µl de sangre con 500 µl de TE (10 mM tris, 1 mM EDTA, pH 8), se centrifuga a 11 000 rpm durante 10 s y se descarta el sobrenadante. Se repiten estos tres pasos hasta que el pellet quedo limpio. Luego se agrega 100 µl de la preparación obtenida a partir de 1000 µl agua molecular, 50 mg de Chelex(r) 100 y 1 mg de proteinasa K. Posteriormente se incuba a 56 °C durante 50 min y a 95°C durante 10 min.
La técnica adaptada para muestras de pelo es la descrita por Walsh7. Se colocaron tres pelos en un microtubo. Para ello se hace un corte al pelo a 3-4 cm de la raíz (con el bulbo) y se mezcla con 1000 µl de agua destilada. Luego se coloca en el bloque térmico a 70 °C durante 3 min, y se centrifugan a 14 000 rpm durante 3 min. Se descarta el sobrenadante dejando cerca de 30 µl del volumen y se agrega 200 µl de Chelex(r) 100 al 5% y se incuba durante 30 min a 56 °C. Las muestras se mezclan mediante el vortex por 5 s, y finalmente el tubo se coloca en agua hirviendo durante 8 min.
Método proteinasa K
Se adaptó la técnica descrita por Martínez 8 para muestras de sangre. Se mezcla 50 (l de sangre total con 500 (l de TE, se centrifuga a 11 000 rpm durante 10 s, se retira el sobrenadante y el precipitado se resuspende en 500 (l de TE. Luego, se centrifuga a 11000 rpm durante 10 s. Estos pasos se repiten dos veces. Se descarta el sobrenadante del último lavado y el precipitado se resuspende en 100 (l de tampón. Se incuba a 56 ºC durante 45 min; luego se eleva la temperatura a 95 ºC durante 10 min. Finalmente, las muestras se diluyen 1:2 con TE y se conservaron a -20 ºC.
La técnica adaptada para muestras de sangre en papel filtro y pelo descripta por Martínez 8, se inició, transfiriendo tanto pelos como cerca de 1 cm2 de papel filtro impregnado de sangre a cada tubo de 1.5 ml, seguidamente en cada uno de ellos se añadió 100 (l de tampón K, incubando a 56 ºC durante 45 minutos y luego a 95 ºC durante 10 minutos. Las muestras fueron conservadas a -20 ºC
Integridad y Cuantificación del ADN
Se utilizó el NanoDrop ND-1000 para cuantificar las 30 muestras evaluadas por triplicado y se determinó el valor promedio de la concentración y pureza de ADN de cada una de las muestras.
PCR Convencional
Mediante la amplificación por PCR se verificó la integridad del ADN. Se utilizó el gen 12S, específico para ovinos y los siguientes pares de cebadores: forward 5´-AGCCTGTTCTATAATCGATA-3´ y reverse 5´-GTCTCCTCTCGTGTGGTTGA-3´, los que generan una banda de 364 pb. La PCR fue llevada a cabo en un termociclador 1000 Thermal Cycler, Biorad(r), mediante un programa térmico, conllevando una primera etapa a 94 °C durante 1 min., seguido de 25 ciclos que comprendían, una primera fase de desnaturalización a 94 °C durante 1 min, una fase de hibridación a 54 °C durante 1 min y una fase de extensión de la cadena a 72 °C durante 1 min. El programa térmico finalizó con una extensión a 72 °C durante 5 min.
Electroforesis
Los productos de la PCR fueron posteriormente detectados mediante geles electroforéticos de agarosa al 1.5%, utilizando bromuro de etidio como agente intercalante del ADN. Es importante destacar que cada etapa del procedimiento (extracción de ADN, amplificación, carga de ADN y electroforesis) fue realizada en salas separadas.
Diseño y Análisis Estadísticos
Los datos se analizaron mediante el programa estadístico Infostat v. 2018e9. Se realizó estadística descriptiva y la distribución teórica de las observaciones fue verificada con la prueba de Kolmogorov-Smirnov utilizando la corrección de Lilliefors. Una vez determinada la normalidad de la distribución, se aplicó el análisis de varianza con arreglo factorial de tratamientos (diseño bifactorial) y test de Tukey para determinar diferencias entre medias.
RESULTADOS Y DISCUSIÓN
Se logró la extracción del ADNg de los tres tipos de muestras con los dos métodos de extracción en estudio. Los resultados (Tabla 1) revelan la distribución normal de las observaciones.
Tabla 1 Resultados de la prueba de normalidad de la variable concentración de ADN, obtenidos a través del test Kolmogorov-Smirnov utilizando la corrección de Lilliefors
| Variable | Estadístico | n | p -valor |
|---|---|---|---|
| Concentración ng/µl | 0.15408 | 30 | 0.06703 |
Las concentraciones de ADN obtenidas se presentan en la Tabla 2. El método de extracción Chelex presentó un mayor promedio de concentración de ADN a partir de sangre entera (111.87 ng/µl), en tanto que la menor concentración de ADN se observó en la muestra de pelos (20.61 ng/µl). Para el caso del protocolo de extracción proteinasa K, la mayor concentración se observó para las muestras en papel filtro (151.75 ng/µl), en tanto que el valor más bajo se obtuvo con pelos (18.25 ng/µl). Resultados similares han sido reportados en otras investigaciones 10,11,12.
Si bien, algunas muestras evidenciaron valores de concentración más altas en comparación a otras, es importante acotar que se encontraron dentro de límites aceptables para amplificación, teniendo en cuenta que el promedio requerido es normalmente de 20 ng/µl 13 y en un rango de 70 a 100 ng/µl 10
Tabla 2 Concentración (ng/µl) de ADN obtenido a partir de tres matrices biológicas y procesadas mediante dos métodos de extracción.
| Método | Materiales | X | ±DE | CV | A |
|---|---|---|---|---|---|
| Chelex | Papel Filtro | 79.63 | 21.58 | 27.10 | 52.45 |
| Sangre Entera | 111.87 | 18.68 | 16.69 | 45.6 | |
| Pelo | 20.61 | 1.61 | 7.81 | 4.03 | |
| Proteinasa K | Papel Filtro | 151.75 | 34.06 | 22.44 | 71.96 |
| Sangre Entera | 42.45 | 8.69 | 20.48 | 20.22 | |
| Pelo | 18.25 | 18.25 | 16.83 | 8.3 |
±DE = desvío estándar; CV = coeficiente de variación; A= amplitud
Con relación a la pureza obtenida con ambos métodos, se evidenció una mayor cantidad de muestras contaminadas a partir del método de extracción con Chelex (Tabla 3).
Tabla 3 Pureza de ADN obtenido a partir de tres tipos de muestras y dos métodos de extracción de ADN (n=30 muestras biológicas)
| Método | Materiales Biológico | Categorías | Frecuencia | |
|---|---|---|---|---|
| N | % | |||
| Chelex | Papel Filtro | Contaminado | 5 | 17 |
| Sangre Entera | Contaminado | 5 | 17 | |
| Pelo | ARN | 1 | 3 | |
| Óptimo | 4 | 13 | ||
| Proteinasa K | Papel Filtro | Aceptable | 1 | 3 |
| Contaminado | 4 | 13 | ||
| Sangre Entera | Aceptable | 1 | 3 | |
| ARN | 1 | 3 | ||
| Óptimo | 3 | 10 | ||
| Pelo | Aceptable | 2 | 7 | |
| Contaminado | 1 | 3 | ||
| Óptimo | 2 | 7 | ||
Esto pudo deberse, quizás a un problema de contaminación al momento de la manipulación del reactivo o al hecho de que la técnica, si bien presenta un procedimiento sencillo y corto, requiere la utilización de esferas magnéticas que, al término del procedimiento, permanecen alojadas en el fondo de los microtubos, pudiendo propiciar valores de contaminación. Teniendo en cuenta esto, algunos autores 14 recomendaron utilizar el separador magnético a la solución de ADN durante 3 min a fin de eliminar las esferas magnéticas que pudiesen haber permanecido en el tubo. Por otro lado, las muestras reflejan gran cantidad de impurezas que pueden deberse a hidratos de carbono, péptidos, fenoles, sales tampón, y otros compuestos aromáticos 15.
Por su parte, el protocolo con proteinasa K mostró mayores variaciones de pureza, lo cual es consistente con los valores expresados en similares investigaciones, que señalan que la pureza promedio de muestras obtenidas por este método es A260 / 280 > 1.7 14.
Los resultados del análisis inferencial mostraron diferencias significativas al evaluar los valores de concentración obtenidos a partir de los tres tipos de muestras y los métodos de extracción (Tabla 4).
Tabla 4 Promedios de concentración de ADN utilizando tres tipos de muestras y dos métodos de extracción
| Material | Método | Promedio |
| Pelo | Proteinasa K | 18.25ª |
| Pelo | Chelex | 20.61ª |
| Sangre entera | Proteinasa K | 42.45ª |
| Papel filtro | Chelex | 79.63b |
| Sangre entera | Chelex | 111.87b |
| Papel filtro | Proteinasa K | 151.75c |
a,b,c Promedios con letras diferentes son significativamente diferentes (p<0.05)
El diseño bifactorial reveló que concentraciones de ADN obtenidas a partir de pelo y extraídas mediante el protocolo Chelex y Proteinasa K fueron estadísticamente similares y de valores más bajos. Contrariamente, la concentración alcanzada con papel filtro y método de extracción proteinasa K fue la más elevada y estadísticamente diferente a los demás valores (p<0.05). Por otra parte, las concentraciones de este trabajo fueron similares a las observadas en investigaciones 10,16 referentes a muestras de pelo.
El factor que pudo provocar diferencias entre los valores obtenidos con los protocolos de extracción Chelex y proteinasa K a partir de sangre entera, pudo estar relacionado a la degradación del ADN, vinculado a las diferencias en cuanto al tiempo entre la realización de un protocolo y otro, o que para muestras de sangre, la utilización del Chelex no es tan propicia como para otros tipos de muestras.
Con respecto a la elevada concentración del ADN extraído mediante proteinasa K a partir de papel filtro, debe mencionarse que una alta contaminación proteica pudo conducir a la sobreestimación de la concentración real del ADN 17.
En la Figura 1 se presentan los productos de amplificación de la PCR analizados en gel de agarosa al 1.5% para un fragmento de 364 pb. Los controles negativos en cada reacción se presentaron limpios (ausencia de bandas). En la hilera superior se visualizan las bandas del ADN correspondiente a sangre entera y papel filtro para el método de extracción con Chelex, las que se presentaron íntegras e intactas; sin embargo, para las muestras pelo se observan bandas intactas excepto en dos muestras (12 y 14), donde el ADN probablemente se encontró degradado. El método de proteinasa K mostró bandas intensas para el ADN extraído a partir de pelo y sangre entera; sin embargo, las correspondientes a papel filtro mostraron mayor inconveniente, en coincidencia con el exceso de concentración y de contaminación obtenidos, lo que pudo responder a una elevada contaminación proteica.
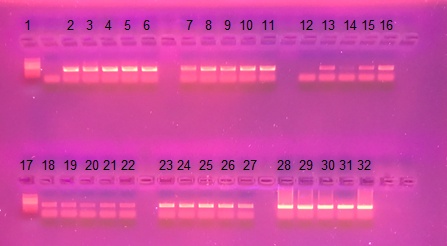
Figura 1 Electroforesis en gel de agarosa al 1.5%. Hilera superior mostrando ADN extraído con el protocolo Chelex. Carriles 2-6 muestras de ADN extraído de sangre entera; carriles 7-11 muestras de ADN extraído de papel filtro; carriles 12-16 muestras de ADN extraído de pelo. Hilera inferior mostrando ADN extraído con el protocolo Proteinasa K. Carriles 18-22 muestras de ADN extraído de papel filtro; carriles 23-27 muestras de ADN extraído de pelo; carriles 28-32 muestras de ADN extraído de sangre entera
CONCLUSIÓN
En base a los resultados obtenidos se concluye que, el método de extracción con resina Chelex resultó un método simple que permitió la extracción de ADN en mayor cantidad a partir de los tres materiales biológicos, pero con bajo índice de pureza.
Por su parte, el método de extracción con proteínasa K fue un método simple, corto; sin embargo, produjo cantidades de concentración y niveles de pureza apropiados para estudios moleculares a partir de los tres materiales biológicos.















