INTRODUCCIÓN
Las bacterias son capaces de desarrollar varios mecanismos de resistencia frente a uno o más antimicrobianos; aquellos adquiridos y transmisibles son los más significativos debido a su potencial de diseminación, consisten fundamentalmente en la producción de enzimas inactivadoras o en la aparición de modificaciones que impiden la llegada de la droga a su sitio de acción1.
Salmonella enterica es un patógeno zoonótico transmitido por alimentos de gran repercusión en el mundo con creciente aparición y diseminación de variantes resistentes a los antimicrobianos. Las quinolonas son junto a los β-lactámicos los de mayor uso, y el nivel de resistencia a los mismos ha aumentado considerablemente en los últimos años en todo el mundo2. La aparición de cepas de Salmonella resistentes a las cefalosporinas de tercera generación (C3aG), quinolonas y a colistina es motivo de alarma y representa una amenaza clínica progresiva, ya que las primeras son tratamiento de elección de salmonelosis y especialmente para las infecciones invasivas por Salmonella no tifoidea3.
La resistencia a las C3aG se ha atribuido principalmente a la gran diseminación de plásmidos portadores de genes que codifican β-lactamasas de espectro extendido (BLEE) y β-lactamasas del tipo AmpC4. Las β-lactamasas de espectro extendido hidrolizan penicilina, cefalosporinas de 1a, 2a, 3a y 4a G, y monobactam; aunque existe una gran variedad de éstas enzimas, actualmente CTX-M es la más frecuente en la mayor parte del mundo; con diferencias en la prevalencia de sus variantes en los distintos países5; siendo CTX-M-2 y CTX-M-15 las de mayor diseminación a nivel regional y mundial6-10.
A su vez β-lactamasas tipo AmpC, hidrolizan cefalosporinas de 1a, 2a, 3a G, y con muy poca eficacia a las de 4a G5,11. La producción de AmpC en algunas especies bacterianas puede ser constitutiva o inducible; en el caso de Salmonella spp., puede ser adquirida a través de un plásmido y el gen blaCMY-2 fue encontrado en cepas, principalmente asociadas a aves de corral12-14. Además es posible la presencia simultánea de diferentes mecanismos de resistencia a β-lactámicos, (BLEE/AmpC/pérdida de porinas)5,15, lo que podría dificultar la interpretación fenotípica y se debe recurrir a los métodos moleculares.
En cuanto a las quinolonas se ha venido observando en enterobacterales el paulatino incremento de la resistencia a esta familia de antimicrobianos16. Una de ellas, la ciprofloxacina, es utilizada en el tratamiento de enfermedades infecciosas, principalmente en diarreas graves, como así también es de uso frecuente en el ámbito veterinario17, hechos que han favorecido el aumento de su resistencia en diferentes patógenos.
Entre los principales mecanismos adquiridos de resistencia, se destacan los PMQR (plasmid-mediated mechanisms of resistance to quinolones), que involucran proteínas citoplasmáticas conocidas como qnr (quinolone resistance) las que confieren protección a las topoisomerasas (ADN-girasa y topoisomerasa IV) dificultando la unión a quinolonas; y a la enzima inactivante acetiltransferasa aac (6’)-Ib-cr 18,19.
Se han descrito varios tipos de qnr (qnrA, qnrS, qnrB, qnrC, qnrD, qnrVC y qnrE) cada uno con sus variantes alélicas; estas proteínas suelen estar asociadas con BLEE, AmpC, carbapenemasas y son transportadas en los mismos plásmidos conjugativos que confieren multirresistencia20.
La colistina es otro antimicrobiano que requiere vigilancia, debido a su uso en el sector ganadero como promotor de crecimiento en animales de consumo21. Su reintroducción en esquemas de tratamiento se debe a las infecciones humanas causadas por bacterias gramnegativas multirresistentes productoras de carbapenemasas y a la falta de disponibilidad de nuevos medicamentos; sin embargo, su uso está restringido debido a su nefrotoxicidad22. Uno de los determinantes de resistencia a la colistina está mediado por el gen mcr (mobile colistin resistance), cuya transferencia es plasmídica, este gen se ha diseminado rápidamente en diferentes géneros de bacterias gramnegativas en todo el mundo, afectando el medio ambiente, los alimentos, los animales y los seres humanos23,24.
OBJETIVO
El objetivo de este trabajo fue determinar la resistencia a los antimicrobianos y caracterizar genes de resistencia plasmídicos a quinolonas, β-lactámicos y colistina en aislados de Salmonella provenientes de la Vigilancia Integrada de Enteropatógenos entre los años 2020 y 2021.
MATERIALES Y MÉTODOS
Fueron estudiadas 501 cepas de Salmonella spp. como parte de la vigilancia de patógenos entéricos y resistencia a los antimicrobianos, colectadas entre los años 2020 y 2021 por los Laboratorios de la Red de Enteropatógenos del Laboratorio Central de Salud Pública.
Se investigó principalmente la resistencia a las C3aG, a quinolonas y a colistina en aislamientos recuperados de humanos, alimentos, animales de consumo, y ambiente.
La identificación de los aislados se llevó a cabo por metodología de bioquímica convencional. La serovariedad se determinó con antisueros somáticos y flagelares utilizando el esquema de Kauffmann-Le Minor25 y por métodos moleculares como la reacción en cadena de la polimerasa PCR26.
La susceptibilidad a los siguientes antimicrobianos: ampicilina (AMP), amoxicilina/ácido clavulánico (AMC), cefotaxima (CTX), ceftazidima (CAZ), ácido nalidíxico (NAL), tetraciclina(TET), ciprofloxacina (CIP), cloranfenicol (CHL), gentamicina (GEN), nitrofurantoína (NIT), cotrimoxazol (SXT) se evaluó por el método de Kirby Bauer siguiendo las pautas del CLSI (Clinical and Laboratory Standards Institute)27.
Para la búsqueda de genes de resistencia plasmídica se utilizó el siguiente tamizaje fenotípico: presencia de resistencia o sensibilidad disminuida a NAL y CIP; resistencia a CTX y CAZ con y sin sinergia fenotípica visible para BLEE y AmpC (utilizando AMC - AFB: ácido fenilborónico - resistencia a FOX: Cefoxitina); y resistencia a colistina(COL) utilizando la técnica tamizaje de ASC (Colistina Agar Spot)28.
Las cepas seleccionadas fueron sometidas a pruebas de reacción en cadena de la polimerasa (PCR) convencional para la caracterización molecular de los genes de resistencia plasmídicos: PMQR como qnr (A, B y S) y aa-6¨-Ibr; genes de β-lactamasas (CTX-M, PER-2 y CMY); y el gen mcr-1 de resistencia a polimixinas29.
Todos los datos obtenidos fueron recopilados de la base de datos del sistema de gestión laboratorial utilizado en el Laboratorio Central de Salud Pública. Los análisis estadísticos se realizaron con el programa R versión 4.3.1, se calculó los intervalos al 95% según Newcombe30.
RESULTADOS
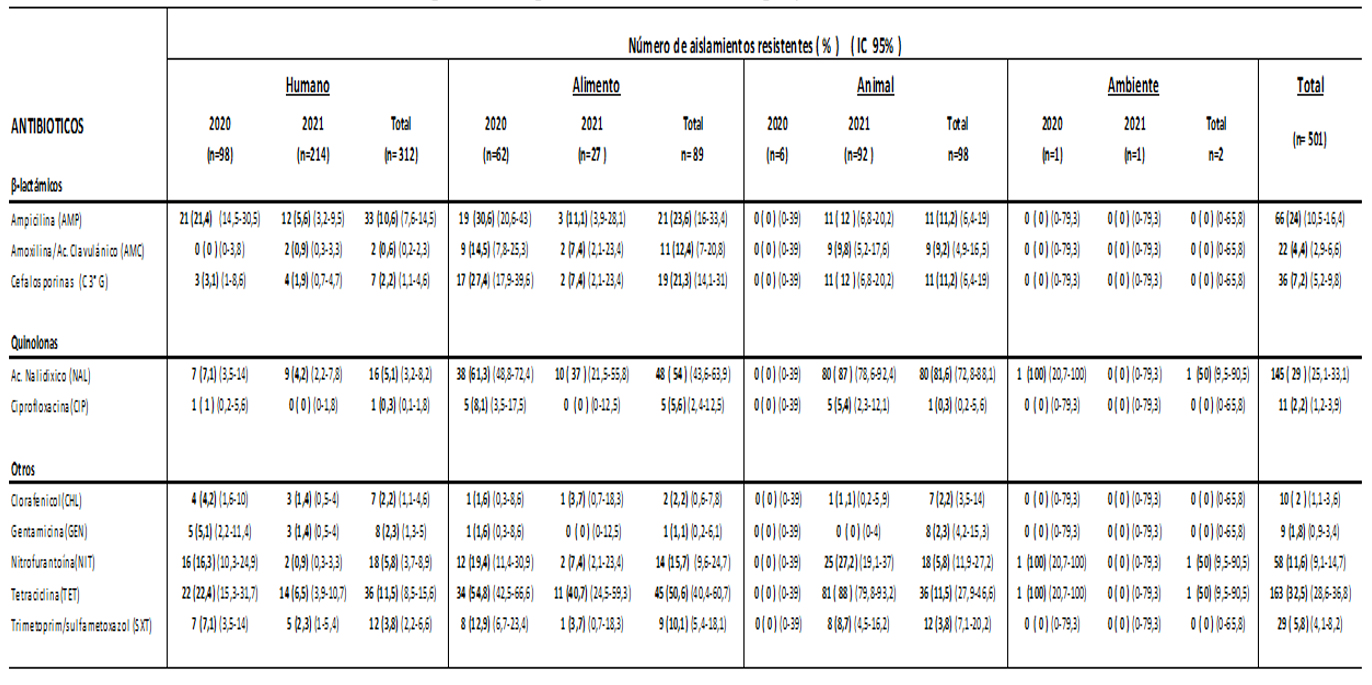
De las 501 cepas de Salmonella spp. estudiadas, 167 fueron del año 2020 y 334 del año 2021. El origen de los aislamientos se distribuyó en: muestras clínicas de humanos (n =312), muestras de alimentos (n= 89), animales de consumo (n= 98), y ambiente (n= 02). En las Tablas 1 y 2 se visualizan respectivamente el porcentaje de resistencia encontrado en los aislamientos de Salmonella a los grupos de antimicrobianos vigilados, y la resistencia sumada a la sensibilidad intermedia a quinolonas.
Tabla 2: Resistencia + sensibilidad intermedia en Salmonella a quinolonas - 2020-2021.
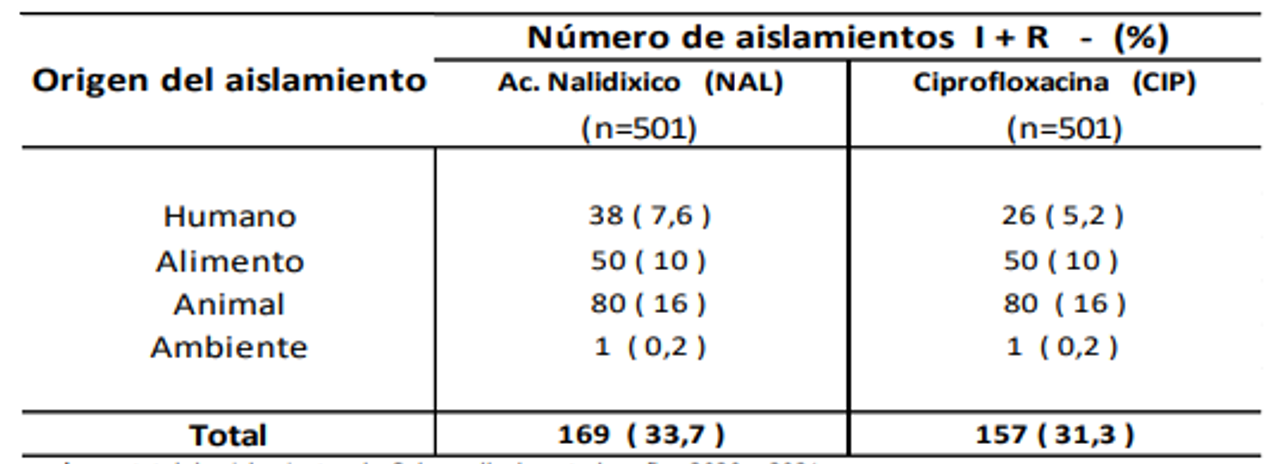
n: número total de aislamientos de Salmonella durante los años 2020 y 2021.
En cuanto a los mecanismos de resistencia plasmídicos, en las tablas siguientes se muestran todos los mecanismos detectados y se pueden visualizar los resultados de genotipo en el 2020 (Tabla 3) y en 2021 (Tabla 4).
Tabla 3: Caracterización de genes de resistencia trasmitidas por plásmidos en Salmonella - 2020 - Paraguay.
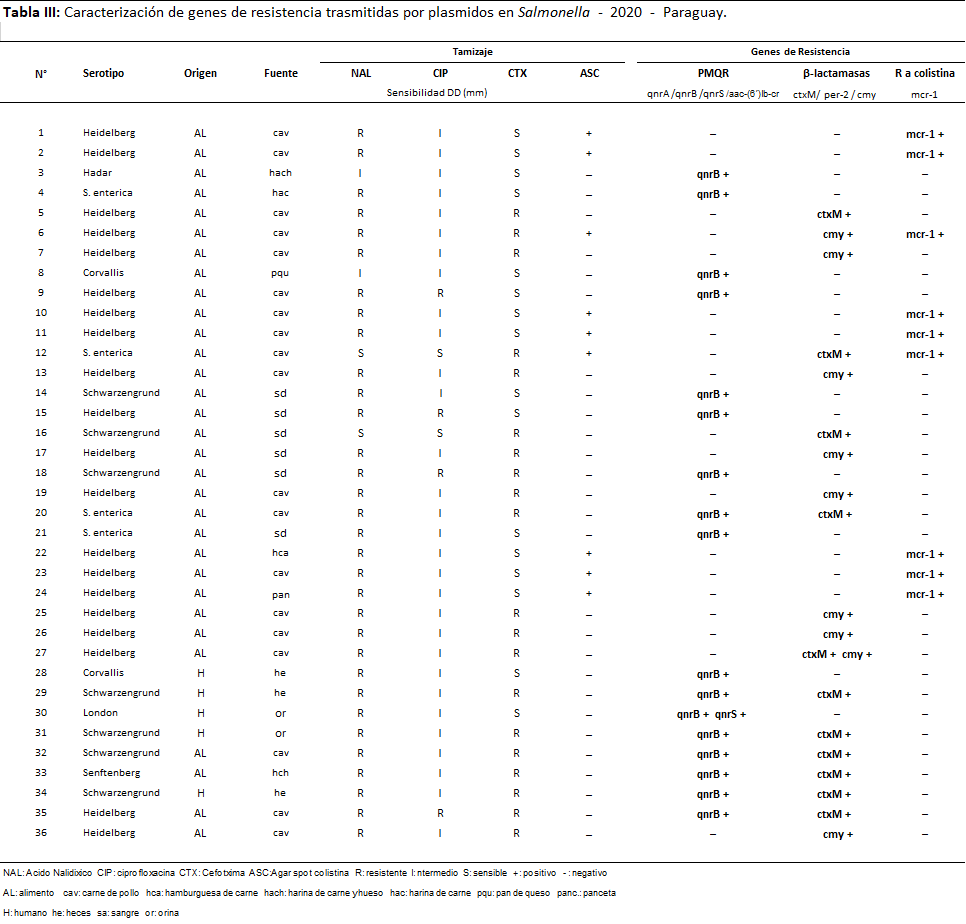
En el año 2020 el 21 % de las cepas (36/167) presentaron mecanismos de resistencia; de las cuales fueron de origen alimentario el 86% (31/36) todos provenientes de carne de aves y el 14% (5/36) de origen humano. Fueron 47 los genes de resistencia detectados, algunos de ellos coexistiendo en la misma cepa; los prevalentes fueron genes de β-lactamasas 42,6% (20/47), PMQR 38,3% (18/47) y 19.2% (09/47) de mcr-1.
Tabla 4: Caracterización de genes de resistencia trasmitidas por plasmidos en Salmonella - 2021 - Paraguay.
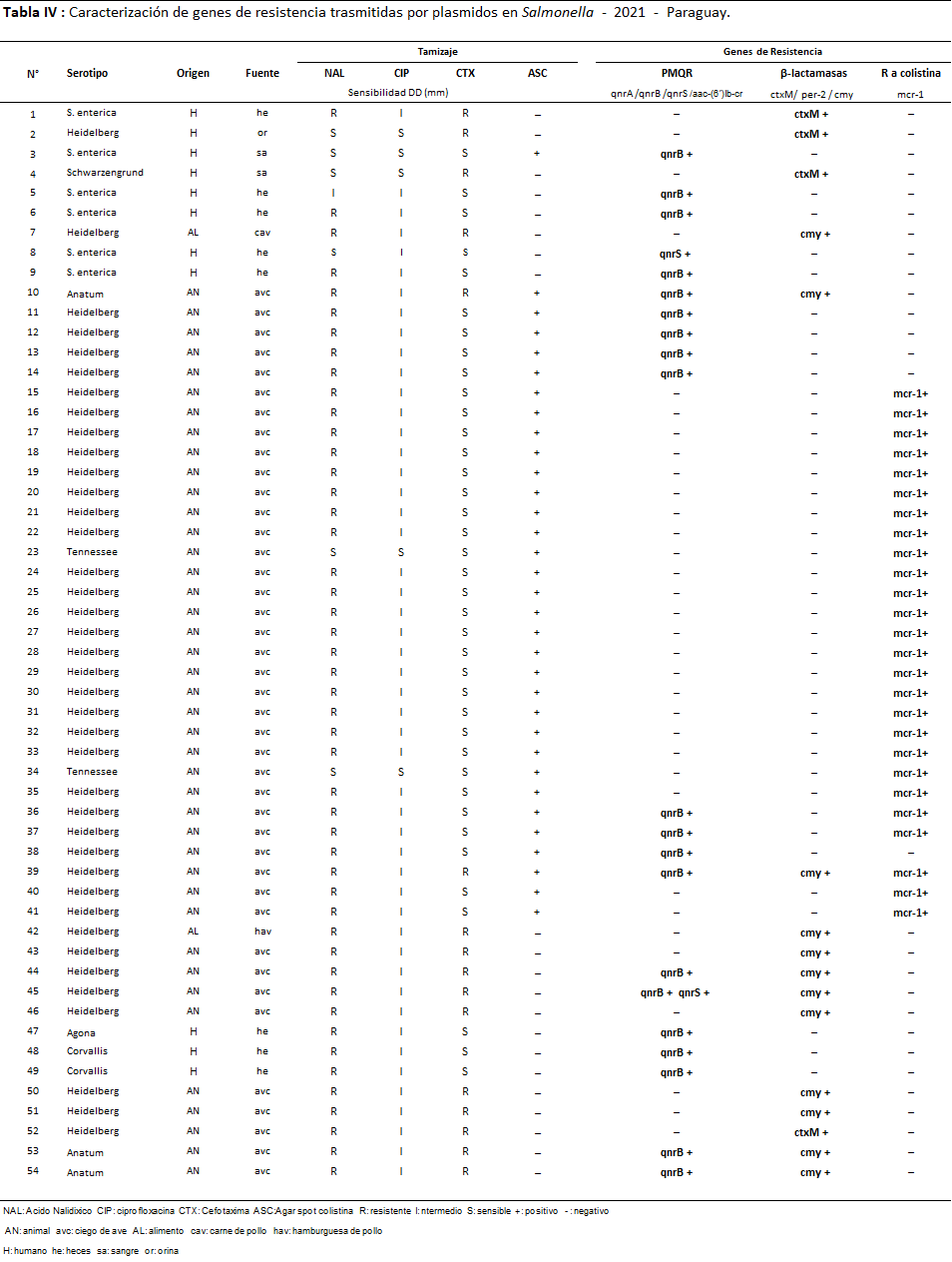
En el año 2021 el 16,2% (54/334) de las cepas presentaron mecanismos de resistencia: de origen animal 75,9% (41/54-ciego de aves), de humanos el 20,4% (11/54) y de alimentos un 3,7% (2/54 también de aves). Fueron detectados 64 genes de resistencia donde el 40,6% (26/64) fueron genes mcr-1; 34,4% (22/64) fueron PMQR y 25% (16/64) β-lactamasas.
El 18% (90/501) de las cepas de Salmonella estudiadas en los dos años presentaron al menos un mecanismo de resistencia, fueron detectados un total de 111 genes plasmídicos (71 cepas portaron un solo gen, 17 cepas dos genes y 2 cepas tres genes distintos), distribuidos de la siguiente manera Qnr B: 41,1% (37/90); mcr-1: 38,9% (35/90); CMY: 23,3% (21/90); CTX-M: 16,7% (15/90) y Qnr S: 3,3% (3/90); no fueron detectados los genes Qnr A, aa-6¨-Ibr, ni PER-2.
En cuanto a los serotipos 59 fueron identificados como Heidelberg (22 de alimentos, 36 de origen animal y 1 de humano), 08 Schwarzengrund (4 de alimentos y 4 de humanos); 4 Corvallis (3 de humanos 1 de alimento), 1 Agona y 1 London (ambos de humanos); 2 Tennessee, 3 Anatum (todos de animal), 1 Senftenberg (de alimento); 1 Hadar (de alimento) y por último 10 S. entérica (6 de humanos y 4 de alimentos).
DISCUSIÓN
En este estudio realizado en Paraguay durante un periodo de dos años, se investigó la resistencia antimicrobiana en cepas de Salmonella spp. aisladas de diversas fuentes, incluyendo alimentos, animales y muestras clínicas de humanos. Los resultados mostraron resistencia a varios antimicrobianos y la existencia de mecanismos de resistencia transferibles por plásmidos en estas cepas. Es importante mencionar que en nuestro estudio el perfil de resistencia encontrado fue más pronunciado en los aislamientos provenientes de alimentos y animales que en los de origen humano.
Las cepas analizadas en conjunto exhibieron mayor resistencia a tetraciclina, ácido nalidíxico y ampicilina, hallazgos similares fueron encontrados en estudios previos realizados en el país31 y comparable a otros realizados a nivel regional32,33; también son consistentes con un informe de la Unión Europea en indicadores humanos y bacterias zoonóticas que reveló un aumento generalizado en la resistencia de Salmonella a la ampicilina y tetraciclina entre 2019 y 202034; pero a diferencia de nuestros hallazgos la resistencia a las C3aG fue menor ( por debajo del 1% en aislamientos humanos y casi nulo en aislamientos de alimentos y animales).
Si tenemos en cuenta los antimicrobianos utilizados con mayor frecuencia para el tratamiento de salmonelosis, en nuestro estudio los aislamientos humanos mostraron resistencia a ampicilina 10,6%, cotrimoxazol 3,8%, C3aG 2,2% y ciprofloxacina 0,3% (5,2% de sensibilidad intermedia). Nuestros porcentajes son más bajos en comparación a los descritos en Brasil por Oliveira do Reis y col en el año 201935 en muestras de orina ( ampicilina 31,3%, cotrimoxazol 9,4%, C3aG y ciprofloxacina 3,1%); y Perú en estudios realizados por Quino y col.33) donde se encontraron resistencias superiores al 50% frente a la ampicilina, cotrimoxazol y cefotaxima en aislados de muestras humanas y no humanas, con la diferencia que las cepas estudiadas en dicho trabajo fueron del serotipo Infantis. Estos resultados sugieren que existe variabilidad en la resistencia antimicrobiana entre diferentes países y regiones, lo cual puede estar influenciado por varios factores como la epidemiología de las cepas circulantes.
En cuanto a las quinolonas, se observó una alta resistencia en ácido nalidíxico, en cepas aisladas de animales, alimentos y del ambiente. Con ciprofloxacina, si bien la resistencia en cepas de todos los orígenes estudiados alcanzó solamente 2,2%, al considerar la sensibilidad disminuida (categoría intermedia de sensibilidad) los valores aumentaron significativamente, dato relevante en esta droga de primera línea en tratamiento de infecciones humanas.
En relación con los mecanismos de resistencia se detectó la presencia de PMQR en las cepas con sensibilidad disminuida. El gen prevalente encontrado fue QnrB (41,1%), el cual estuvo uniformemente distribuido en la población humana, animal y en alimentos. También se detectó el gen QnrS en un 3,3% de las cepas; además se encontraron 2 aislamientos con asociación de los genes QnrB y QnrS, principalmente en humanos. Sin embargo, no se detectaron los determinantes de tipo QnrA ni aac-(6´)-Ib-cr. Estos hallazgos difieren de estudios realizados en diferentes países, por ejemplo, en Corea del Sur36 se encontró una alta prevalencia de genes QnrS (67,6%) en aislamientos humanos, además QnrB, aac-(6´)-Ib-cr, QnrA en menor porcentaje, y la presencia de genes combinados QnrS y aac-(6 ́)-Ib-cr. Un estudio realizado en Brasil37 en Salmonella aisladas de humanos y productos alimenticios reveló la prevalencia del gen aac-(6´)-Ib-cr seguido de Qnr S, Qnr B y una cepa Qnr D cuyos resultados también difieren a los nuestros.
Por otro lado, en un estudio realizado recientemente en muestras cecales de bovinos en nuestro país38 se encontró en cepas de E. coli 8,3% de resistencia a quinolonas debida a la portación de los genes QnrS, QnrB y aac-6´-Ib-cr, estos datos demuestran la circulación de genes similares en diferentes enterobacterales.
Otro aspecto importante a resaltar es que varios estudios sugieren que los animales de consumo y sus alimentos derivados son reservorios de bacterias portadoras de β-lactamasas (como BLEE o AmpC) y que podrían promover la transmisión de estos determinantes de resistencia a los humanos39-41. En nuestro trabajo se pudo observar que el 40% de la población bacteriana con mecanismos de resistencia plasmídicos fueron portadoras de enzimas β-lactamasas y los genes detectados fueron en el 23,3 % de los casos del tipo CMY casi en su totalidad provenientes de fuente aviar o sus alimentos derivados y un 16,7 % fueron genes tipo CTX-M. Estos resultados son comparables a los encontrados en Brasil por Melo R. y col.42 donde estudiaron cepas Heidelberg de pollos encontrando 80% CMY-2 y 10% CTX-M; en cambio nuestros datos son diferentes a los resultados de un estudio realizado en Corea40 en aislamientos de Salmonella provenientes de pollo, donde encontraron 11,8% de CTX-M y sólo 0,7% de CMY-2, en este punto es importante considerar que ninguno de sus aislamientos fueron Heidelberg. En el presente estudio, el serovar Heidelberg de origen aviar, fue encontrado como mayor productor de β-lactamasa del tipo AmpC (CMY), también descrito en varios trabajos14,42,43,44. Según una clasificación general45 de los 10 primeros serotipos prevalentes en muestras de aves de corral en las Américas, fueron más frecuentes Heidelberg, Kentucky, Enteritidis y Typhimurium, por otro lado en un estudio de la dinámica de Salmonella en productos avícolas46, los serovares Heidelberg y Minnesota fueron los más prevalentes en Brasil y en productos cárnicos importados de Brasil, con resistencia a betalactámicos conferida por los genes blaCMY-244.
En nuestro trabajo las cepas de Salmonella portadoras de β-lactamasas del tipo CTX-M (16,7%) estuvieron presentes tanto en humanos como en pollos (animales y alimentos derivados). Además estos genes estuvieron asociados a los serotipos Schwarzengrund, Heidelberg y Senftenberg; éstos hallazgos son similares a los de Silva y col. en 201347 donde se encontró un 14% de CTX-M en aves de corral, principalmente en los serotipos Schwarzengrund y Agona. En Brasil un estudio realizado por Fernandes y col. en 2016, que examinó 630 cepas de Salmonella aisladas de humanos y no humanos, encontró que el 7,3% de las cepas portaban β-lactamasas de espectro extendido, siendo la mayoría variantes de CTX-M como CTX-M8 y CTX-M2.48. Estos resultados respaldan la evidencia de la presencia y dispersión de cepas de Salmonella portadoras de genes CTX-M en humanos y en animales de consumo, lo que sugiere una posible transmisión de estos genes a través de la cadena alimentaria.
Es interesante observar que en los últimos años cepas de Salmonella Infantis, fueron descritas como un problema emergente en varios países del mundo, debido a la portación de β-lactamasas del tipo CTX-M (gen blaCTX-M65) en humanos y en otras fuentes49-52, sin embargo el serotipo Infantis portador de resistencia plasmídica no fue encontrado en nuestro actual trabajo.
La aparición de genes móviles de resistencia a colistina (mcr) ha generado preocupación en todo el mundo y su presencia en aislados provenientes de animales sugiere un papel importante en la transmisión alimentaria53. El gen mcr-1 ya se ha identificado en bacterias de humanos, animales, productos alimenticios animales y fuentes ambientales en diferentes países de América54, incluyendo nuestro país55. Un análisis de revisión sistemática mostró que la prevalencia del gen mcr-1 es mayor en aislamientos de animales que en alimentos y humanos en América Latina56.
Según un estudio realizado en países de altos ingresos de todos los continentes una diversidad de organismos entre los cuales se encuentra Salmonella albergan varios genes mcr y están ampliamente difundidos en la industria avícola; E. coli fue identificado como el organismo predominante en la propagación de genes mcr57,54. Luego de la alerta epidemiológica emitida por Laboratorio de Referencia Regional58 de las Américas muchos países de la región reforzaron la vigilancia logrando detectar el gen mcr-159-64; en nuestro país fue evidenciada la circulación del gen mcr-1 con un trabajo de vigilancia multicéntrico55 en el que fueron estudiadas 150 cepas de infecciones humanas: 7 aislamientos ( 3 E. coli, 3 Klebsiella pneumoniae y 1 Salmonella enterica serovar Schwarzengrund).
En nuestro estudio se identificó una prevalencia alta de mcr-1 entre los mecanismos plasmídicos detectados (38,9% de mcr-1), los aislados de Salmonella que contenían estos genes en su mayoría del serotipo Heidelberg fueron provenientes de animales y alimentos de origen aviar, y solamente un aislado fue proveniente de panceta; sin embargo no fue detectado en aislados de origen humano, lo que sugiere baja circulación de este gen en los serotipos que habitualmente producen infecciones en el hombre.
Muy diferente a lo descrito por Sia y col.65 quienes describieron la vigilancia pública de Inglaterra 52 cepas de Salmonella de con genes mcr, 32 correspondientes a mcr-1 en su mayoría humanos asociados al serovar Typhimurium monofásica; y por Fortini y col.53 que igualmente encontraron este serotipo en humanos y asociado a mcr-1 y mcr-5.
El hallazgo de una mayor cantidad de genes de resistencia en cepas provenientes de aves destinadas al consumo (pollo: en pie como en alimento) es un factor preocupante. El serovar Heidelberg ha sido identificado como el principal productor de resistencia a β-lactámicos (CMY) y colistina (mcr-1), y también portador de otros genes asociados a dicha resistencia.
Es importante destacar que en el caso de las cepas de serovar Schwarzengrund y Senftenberg encontramos β-lactamasas de espectro extendido del tipo CTX-M y resistencia a quinolonas tipo Qnr B, estos genes se observaron tanto en humanos como en animales y alimentos. Otros serotipos como Corvallis, Anatum, Tennessee, Agona, Hadar y London también fueron portadores de genes de resistencia.
La detección y caracterización molecular de genes de resistencia en cepas de Salmonella, constituye un avance importante en el conocimiento sobre la circulación de cepas que presentan resistencia, en alimentos y animales de consumo en nuestro país, y que pueden transmitirse fácilmente al ser humano. Es crucial destacar que la resistencia observada está mediada por mecanismos de transferencia de genes, lo que implica que puede ser transmitida entre cepas bacterianas y entre diferentes especies, hecho que contribuye a la propagación y diseminación de la resistencia antimicrobiana.
Esta realidad nos sitúa en posición vulnerable, representa un desafío significativo en términos de tratamiento de infecciones y resalta la importancia de continuar fortaleciendo la vigilancia multisectorial y multidisciplinaria en las áreas involucradas, para tomar decisiones cimentadas y aplicar políticas que apunten a intervenciones para mitigar este problema.
En consecuencia, es necesario continuar monitoreando la resistencia antimicrobiana en Salmonella y aplicar estrategias efectivas de control y prevención para reducir la propagación de cepas resistentes y asegurar la eficacia de los tratamientos antimicrobianos.