INTRODUCCIÓN
El agua es un recurso natural básico para la supervivencia de los seres vivos. Sin embargo, es vehículo para la transmisión de una amplia variedad de patógenos. Más de 2,5 millones de personas mueren por año por enfermedades asociadas a aguas contaminadas y condiciones sanitarias precarias, mayormente en países en desarrollo1,2. La calidad de la sanidad del agua es determinada usualmente por la concentración de coliformes fecales indicadoras y ocasionalmente por bacteriófagos3,4. Sin embargo, existen numerosos estudios que demuestran que el alcance de los estándares mínimos de coliformes fecales no predice la contaminación viral y la presencia de otras especies patógenas bacterianas en el agua3,5.
Las inundaciones son eventos hidrometereológicos extremos que constituyen uno de los factores climáticos que promueven la aparición de epidemias. Los cambios en los patrones de precipitación están aumentando en frecuencia e intensidad debido al cambio climático6-8, como consecuencia, se produce el aumento en la ocurrencia y en la magnitud de los eventos de inundación extrema, que son reconocidos como el tipo de desastre natural más frecuente y devastador en el mundo9. Estas aguas de inundación pueden irrumpir en los desagües y en la infraestructura de abastecimiento de agua10 causando la exposición de los pobladores a efectos inmediatos como: hospitalizaciones, efectos psicológicos, daños fisiológicos e incluso mortalidad11,12 así como contacto directo con el agua contaminada o a los cortes y abrasiones, promoviendo el surgimiento de brotes de una amplia gama de enfermedades infecciosas12-16.
Una de las causas de brotes de enfermedades infecciosas por contaminación de agua o alimentos son los virus. Entre ellos se encuentran virus causantes de gastroenteritis como: rotavirus (RV), adenovirus (AdV), astrovirus (AstV), norovirus (NoV) y sapovirus (SaV)17-19. Estos se transmiten vía fecal - oral, son altamente infecciosos y son excretados en las heces en altas concentraciones (105-1011 partículas virales/gramo) durante un periodo prolongado, que puede llegar a periodos mayores de 28 días2,17. Por ello, pasan fácilmente al sistema de alcantarillado, donde son altamente estables y mantienen su infectividad incluso luego de ser expuestos a procesos de desinfección, resultando en la contaminación potencial de cauces hídricos si no se posee un sistema de saneamiento adecuado18-20.
Los RV, familia Reoviridae, son la mayor causa de gastroenteritis infantil en todo el mundo. La mortalidad asociada a estos virus está estimada en aproximadamente 600.000 por año21. Se clasifican de forma binaria según sus genes VP4 (P[X]) y VP7 (GX) y poseen una enorme variabilidad genética22,23. En nuestro país se ha reportado su circulación con una diferencia en la distribución temporal de RV entre niños y adultos, produciendo en los primeros un pico de incidencia en los meses más fríos del año24.
La familia Caliciviridae incluye a los NoV y SaV y son reconocidos como la causa más importante de brotes y epidemias de gastroenteritis no bacteriana en todos los grupos de edad, que ocurren principalmente como resultado de contaminación de alimentos y agua. Los más prevalentes son los NoV, que están distribuidos en 5 genogrupos (GI-GV) de los cuales GI, GII y GIV infectan humanos y en 32 genotipos que co-circulan y con los que se ha observado, ocurre recombinación25-27. En Paraguay, se han reportado la presencia y epidemiología molecular de NoVs en casos de gastroenteritis aguda (GEA) infantil no bacteriana. Se ha demostrado la circulación y alta prevalencia de cepas pandémicas del genotipo GII.4 así como la alta prevalencia de genotipos poco comunes emergentes28.
Los AstV, familia Astroviridae, son virus de genoma de ARN, se han detectado en heces de niños, pero también se han encontrado en las heces de una amplia variedad de especies de mamíferos, así como en especies de aves de corral y aves acuáticas silvestres29,30. Según la ICTV, están reconocidas 19 especies dentro del género MAstV y 3 especies dentro del género AAstV29,31.
AdV pertenece a la familia Adenoviridae y se divide en 5 géneros. Mastadenovirus es el género específicamente asociado a infecciones y enfermedades en huéspedes mamíferos32. Más de 60 tipos de adenovirus humanos han sido reportados clasificándolos en 7 grupos (A-G). Los serotipos entéricos que se asocian sobre todo con la gastroenteritis son HAdV-40 y HAdV-41 que pertenecen a las especies F, otras especies tales como A, C, y D también se han asociado con la diarrea33,34.
Las micobacterias no tuberculosas (MNT) son responsables de una amplia gama de patologías, principalmente infecciones en heridas en los ojos, oídos, nariz, garganta u otras infecciones generalizadas en el cuerpo y son de importancia para la salud pública35. La mayoría de las MNT presenta resistencia a gran parte de las drogas empleadas en el tratamiento de M. tuberculosis36-38. Además, poseen alta resistencia a los procesos clásicos de desinfección, especialmente al clorado39,40.
Durante los meses de junio a agosto del 2014 la ciudad de Asunción, Paraguay, así como varios municipios del país, sufrió precipitaciones extremas que causaron el colapso del sistema de alcantarillado y de contención de los cursos de agua (ríos y arroyos), ocasionando una de las más graves inundaciones en la historia de la ciudad41. La Dirección de Meteorología e Hidrología de la Dirección Nacional de Aeronáutica Civil (DINAC) registró un nivel histórico de las aguas del río Paraguay a 7,38 metros de profundidad el 10 de julio del 201442. Estos eventos dejaron como resultado un número elevado de damnificados. Según el Fondo de las Naciones Unidas para la Infancia (UNICEF), alrededor de 245.000 personas debieron ser desplazadas a refugios temporales o campamentos provisionales en zonas altas de la ciudad, de las cuales, aproximadamente 180.000 eran menores de edad. Con esto, hubo un aumento de los problemas de salud y seguridad entre los afectados43.
El Departamento de Biología Molecular y Biotecnología (DBMB) del Instituto de Investigaciones en Ciencias de la Salud de la Universidad Nacional de Asunción (IICS-UNA) ha sido pionero en la introducción de técnicas moleculares aplicadas para la identificación y caracterización de micobacterias, como también la identificación y caracterización en genotipos de RV y NoV24,28,44,45.
Por lo anteriormente expuesto y teniendo en cuenta que estos agentes patógenos no pueden ser detectados por técnicas laboratoriales convencionales y que para su detección se requiere el examen de grandes volúmenes de muestra, se implementó una técnica de concentración de micobacterias y agentes víricos en muestras de agua para su posterior detección y caracterización por técnicas moleculares46.
Este trabajo presenta la identificación por técnicas moleculares de micobacterias y agentes víricos que causan brotes epidémicos transmitidos por el agua, en muestras obtenidas de las inundaciones producidas en Asunción, Paraguay, durante el periodo de junio a agosto del 2014, así como datos secundarios de los servicios de salud de barrios afectados, referentes a casos de enfermedades infecciosas registrados en la quincena del evento de inundación.
MATERIALES Y MÉTODOS
Descripción de la zona de estudio:
El río Paraguay es el cuerpo hidrográfico más importante de la ciudad de Asunción, capital de Paraguay, ya que a través de éste desarrolla el comercio fluvial y actividades turísticas. La ciudad posee dos áreas biogeográficas: la zona de la bahía del Río Paraguay y la zona interior. La zona de la bahía, ubicada a 2 km del centro de la ciudad, superficie aproximada de 375 ha., cuenta con una amplia variedad de hábitats; se encuentra nutrida por arroyos que desembocan en el río Paraguay. La bahía, además, es un sistema pulsátil que sufre grandes fluctuaciones estacionales en el nivel de las aguas del río, produciendo la variación de la profundidad y la extensión de sus márgenes a lo largo del año47.
Los arroyos más importantes afluentes en la Bahía de Asunción son: “Guazu” (Cauce Antequera), “Perú” y “Las Mercedes”; los arroyos “Pozo Colorado”, “De los Patos”, “YkuaSati” y “Jaén” corren escondidos bajo el pavimento de Asunción y la mayoría presenta problemas de contaminación ambiental como consecuencia de efluentes industriales, pluvio-cloacales y residuos sólidos urbanos, que conlleva a una severa degradación del sector. Además, sus márgenes se encuentran pobladas por algunos de los barrios más populosos de la ciudad, como son: San Jerónimo, La Encarnación, Ricardo Brugada, 3 de Febrero, Pelopincho y San Felipe; o por asentamientos precarios de poblaciones emergentes siendo uno de los mayores problemas que presentan sus áreas inundables en los márgenes, lo que provoca una serie de daños y perjuicios socioeconómicos de alto impacto año tras año48.
Muestras:
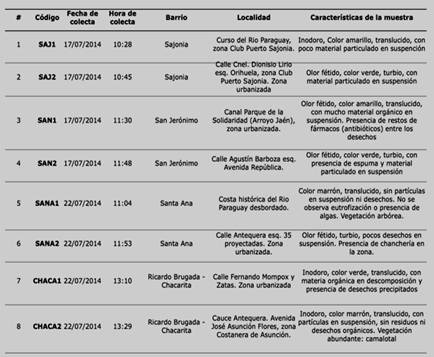
Los puntos de toma de muestra fueron elegidos por conveniencia, incluyendo las zonas más afectadas por las crecidas originadas en las lluvias intensas (Figura 1). Se colectaron muestras de agua superficial de las zonas inundadas de los Barrios San Jerónimo, Sajonia, Santa Ana y Ricardo Brugada - Chacarita (3 de Febrero); en corriente arriba de los arroyos desbordados “Jaen” (Canal de San Jerónimo) e “YkuaSati” (Cauce Antequera) y del cauce principal del Río Paraguay (Tabla 1). El arroyo “YkuaSati” es el que circula de forma subterránea bajo las calles Paraguarí y Antequera y su desembocadura en la bahía corresponde a los puntos Y= 25° 16' 34,68” S; X= 57° 37' 32,67”, cercana a la Escuela Graduada N°18 “República de Cuba” (Tabla 1). Fueron realizadas 8 colectas entre el 17 y el 22 de julio del 2014, pasados dos días del cese de las precipitaciones extremas registradas en Paraguay en ese periodo.
Técnica de colecta y medición de parámetros físico-químicos y microbiológicos: La toma de muestra fue realizada sumergiendo el contenedor de polipropileno a 30 cm de la superficie del agua, con la boca del frasco situada hacia la corriente, para evitar el ingreso de material flotante colectando un volumen final de 10 L. Se procedió a la identificación y codificación de la muestra y a la toma de datos geográficos, utilizando la aplicación de posicionamiento global de Google Maps (Google LLC, California, Estados Unidos) y se midieron in situ los parámetros físico-químicos de: temperatura (°C), salinidad (ppm), conductividad (uS), pH y sólidos disueltos totales (TDS; ppm), con un medidor multiparamétrico (Multi-parameterTestr 35 series, Oaktron, Thermo Fisher Scientific, Singapore). Las muestras luego fueron derivadas al Laboratorio del DBMB del IICS-UNA, donde se procesaron para realizar su concentración y para su posterior codificación y almacenamiento a -20ºC; una fracción de las mismas fue remitida a la plataforma comercial del Laboratorio de Calidad de Aguas de la Facultad de Ciencias Exactas y Naturales de la UNA para el conteo de coliformes totales y fecales por el método de Unidad Formadora de Colonias(UFC), estándar normalizado para el análisis de aguas potables y residuales SM9222D (49).
Detección viral a partir de muestras de inundación:
Preparación de las muestras para análisis viral: Para el estudio de virus presentes en la muestra, de los 10 L colectados en el muestreo se separaron fracciones de 2 L de cada muestra, que se procesaron junto con controles. Se utilizó como control negativo 2 L de agua bidestilada sin inóculo y como control positivo y de inhibición del medio acuoso 2 L de agua bidestilada con 50 μL de inóculos de suspensión fecal de RVA G9P [8], NoV GI.1 y NoV GII.4. Es denominado control de inhibición del medio acuoso a la fracción de la muestra de agua inoculada con suspensión fecal positiva para cada uno de los virus, a fin de detectar substancias presentes en el medio acuoso que puedan afectar los ensayos moleculares.
Concentración viral en muestras de agua: Para optimizar la detección de las partículas virales presentes en la muestra de agua, se realizó la concentración de las mismas por la metodología de floculación con leche descremada pre-Floculada (PSM) al 1% según Calgua et al. en 200846. Brevemente, se ajustó el pH a 3,5 (±0,3) con HCl [1N], mezclando vigorosamente con agitador magnético. Se adicionaron a la muestra 100 mL de leche descremada pre-Floculada (PSM) 1% (pH 3,5) y se homogeneizó levemente con agitador magnético por 8 a 10 horas para que los flóculos se formen. Luego se detuvo el movimiento para que los flóculos sedimenten por gravedad durante 8 a 10 horas y se removió el sobrenadante por medio de succión con tubo de silicona y bomba peristáltica (G-5, UlvacSinku Kiko, Japón). El sedimento con los flóculos fue trasvasado a una botella para centrífuga, con volumen de 500 mL, y llevado a centrifugación (8.000 rpm) durante 30 minutos a 4°C. Se retiró, inactivó y descartó el sobrenadante para luego resuspender el sedimento con 2 mL de PBS. El concentrado final fue homogeneizado con agitador vortex, distribuido en tubos de 5 mL. Una vez concentradas las muestras y los controles, fueron almacenados a -30°C.
Extracción de ácidos nucleicos virales: Se realizó la extracción de los ácidos nucleicos (ARN y ADN) virales a partir de 200 μL de las muestras de aguas concentradas y de los controles mediante el método de extracción con sílica e isotiocianato de guanidina, descrito por Boom et al., 1990 (50). Los ácidos nucleicos extraídos fueron eluídos en agua ultrapura (con inhibidor de ARNasas) a un volumen final de 40 μL y almacenados a -30°C.
Electroforesis en gel de poliacrilamida (PAGE): Los ácidos nucleicos extraídos fueron sometidos a electroforesis en geles de acrilamida al 7,5% en buffer TBE a 10W de potencia constante para la identificación del ARN genómico de rotavirus. Los geles fueron teñidos con nitrato de plata [0,2%] para la visualización de los resultados51.
Detección de genes virales:
La síntesis de ADN complementario (ADNc) se realizó mediante Transcripción Reversa (RT), utilizando iniciadores randómicos bajo el protocolo estandarizado previamente en el DBMB del IICS-UNA. Brevemente, se adicionaron 5 μL de dNTP [2,5 mM] y 2,5 μL de iniciador randómico [50 ng/ mL] a 15 μL ARN extraído. Se incubó a 95ºC durante 5 minutos y se colocó en baño de hielo durante 5 minutos. Luego se adicionaron 27,5 μL de la mezcla de los siguientes reactivos: 14,4 μL de H2O ultrapura, 10 μL de Buffer 5X, 2,5 μL de DTT 0,1 M y 0,6 μL RT-MMLV (Invitrogen®, Thermo Fisher, USA); los tubos se incubaron durante 90 minutos a 37ºC y hasta la inactivación de la enzima se incubó la reacción durante 5 minutos a 85ºC.
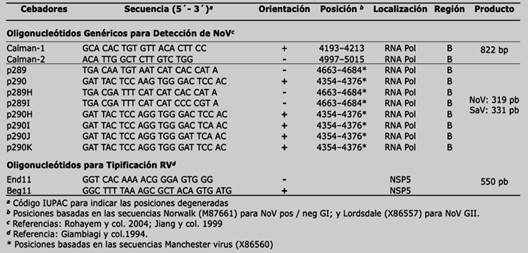
La detección de genes de rotavirus y norovirus se realizó por Reacción en Cadena de la Polimerasa previa retrotranscripción (RT-PCR) del gen NSP5 de rotavirus según el protocolo de Giambiagi et al.51, y PCR anidada para el gen de la polimerasa viral (RdRp) de norovirus, según los protocolos de Rohayem et al., 2004 y Jiang et al., 199952,53. NSP5 es un gen no estructural de los RV y RdRp es el gen que codifica para la polimerasa de los NoV. Los productos amplificados fueron sometidos a electroforesis en geles de agarosa al 2% en buffer TAE a 5 V/cm, teñidos con bromuro de etidio (5 μg/μL) por 10 minutos. y registrados utilizando el sistema de captura de imagen Canon y software Kodak Digital Science. Se consideró como resultado positivo para rotavirus la muestra que presentó el amplicón de aproximadamente 550 pb. del gen de NSP5 y se consideró con resultado positivo para la familia Calicivirus la muestra que presentó uno de los amplicones de 319 pb. (NoV), 331 pb. (SaV) y/o 822 pb. del gen viral (RdRp) mediante las técnicas de RT-PCR y electroforesis en agarosa. Fueron consideradas negativas para estos virus las muestras que presentaron amplicones diferentes a los característicos de dichos genes. En la Tabla 2 se listan las secuencias de los iniciadores, la localización en el genoma y el tamaño de los productos en pares de bases generados por la técnica utilizada para la amplificación de los genes virales.
Reacción en Cadena de la Polimerasa (RT)-PCR en Tiempo Real: Se realizó, además, la detección por amplificación de genes virales por RT-PCR en Tiempo real, en el termociclador ABI 7500 Real Time PCR System (Applied BioSystems, USA), utilizando el kit comercial FTD Viral Gastroenteritis (Fast Track Diagnostics, Luxemburgo), que posee sondas y cebadores y sondas específicos asociados a marcadores de fluorescencia (FAM, VIC y Cy5), para la amplificación de genes de astrovirus, rotavirus, adenovirus (set cebadores y sondas ARA) y, por otro lado, norovirus tipos GI y GII, los genogrupos de NoV más prevalentes en el mundo (set cebadores y sondas NORO) y sapovirus (set cebadores y sondas Sapo), en reacciones independientes; según la metodología recomendada por el fabricante. Brevemente, se colocaron 15 μL de la mezcla de reacción, conteniendo el tampón de reacción, la mezcla de enzimas, los cebadores y las sondas marcadas, en los pocillos de una placa óptica para PCR en tiempo real. Posteriormente, se agregaron en cada uno de ellos, de forma independiente, 10 uL de ácidos nucleicos extraídos de cada muestra y de los controles positivo de la reacción de PCR en tiempo real e interno (gen conservado de otro agente control utilizado para certificar la calidad de los reactivos y la reacción) y como control negativo se utilizó agua destilada, se selló la placa con film adhesivo y se sometió a centrifugación leve por 10 segundos. El programa de amplificación utilizado fue de: 42ºC por 15 minutos, 94ºC por 3 minutos y 40 ciclos de 94ºC por 8 segundos y 60ºC por 34 segundos. Se consideró positiva aquella muestra que logró generar una curva de amplificación de forma sigmoidea con valores de RFU y Ciclo umbral de detección (Ct, del inglés: Cycle Threshold) definidos, según la detección de longitud de onda emitida durante la reacción, específica para cada sonda marcada y gen viral asignado. Los resultados fueron visualizados en computador a través del programa del ABI 7500 Real Time.
Detección de micobacterias a partir de muestras de agua de inundación:
Concentración de micobacterias a partir de las muestras de agua: Se realizó la concentración de MNT por el método de filtración por absorción: Todos los elementos del sistema de concentración fueron lavados en agua desionizada y esterilizados antes de su uso. Brevemente, se separó una fracción de 1 L de los 10 L de cada muestra colectada, para luego ser filtrada primeramente utilizando un sistema de filtración con bomba de vacío y membrana de fibra de vidrio AP40 (Nihon Millipore, Tokio, Japón), de porosidad 0,70 µm, para retener y eliminar las partículas mayores. El filtrado resultante se volvió a filtrar con el mismo sistema, pero a través de una membrana de ésteres de celulosa mixta HA sin carga eléctrica, de porosidad 0,45 mm y 142 mm de diámetro (NihonMillipore, Tokio, Japón). Una fracción de las membranas recuperadas luego de la filtración se guardó en leche descremada a -20ºC, para posterior cultivo, y otra fracción se guardó en tampón TE [1x], para posterior análisis según la metodología de Van Soolingen D et al.54. Los filtros en TE [1x] fueron macerados, agitados en vortex y centrifugados a 14.000 g por 5 minutos. Posteriormente, se retiraron 400 µL del sobrenadante y fue sometido a ciclos de calor (80ºC) y frío (-20ºC) para la inactivación de la muestra, y extracción de ácidos nucleicos por el protocolo descrito por Van Soolingen et al., 199154.
Amplificación de genes de micobacterias no tuberculosas: La detección de ácidos nucleicos de MNT y su caracterización molecular se realizó a través de PCR-asociado al análisis de restricción enzimática (PRA). Se utilizaron los cebadores Tb11 y Tb12 según el protocolo descrito por Telenti et al., 1993, que amplifican un fragmento de 441 pb. del gen que codifica la proteína de choque térmico de 65 kDa (Hsp65) presente en todas las especies de micobacterias55 y los cebadores M1 y M4 descriptos por Wu et al., 2008, que amplifican una región de 463 pb. que contiene al fragmento de 441 bp. del gen Hsp65 con el objeto de aumentar la sensibilidad mediante la PCR anidada56. Los productos de PCR fueron sometidos a electroforesis en geles de agarosa al 2% en buffer TAE a 5 V/cm, teñidos con bromuro de etidio (5 μg/μL) por 10 minutos y registrados utilizando el sistema de captura de imagen Canon y software Kodak Digital Science. Se consideró positiva para MNT una muestra que presente el amplicón de 441 pb. del genHsp65 y su variabilidad mediante la digestión enzimática con HaeIII, excepto por la presencia del patrón 160/140/70 pb., analizando los patrones de restricción con los algoritmos publicados en la plataforma PRASITE, disponible en línea57.
Análisis de reportes de casos de gastroenteritis:
Se realizó una búsqueda activa retrospectiva (BAR) de casos consecutivos que cumplían la siguiente definición de casos operativa: toda persona que haya consultado en los servicios de salud seleccionados y en el periodo estipulado por diarrea y/o vomito acompañado o no de fiebre, de manifestación aguda, o que en la planilla de motivo de consulta figure GEA; que se define principalmente como: 3 o más deposiciones de diarrea acuosa en un mismo día, acompañado de episodios de vómito58.
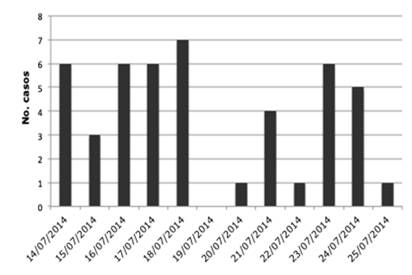
El periodo de búsqueda fue del 14 al 25 de julio de 2014, que corresponden a tres días antes y tres días después de las fechas de toma de muestras ambientales.
Los barrios trabajados fueron San Jerónimo, Sajonia, Santa Ana y Ricardo Brugada - Chacarita y se seleccionaron las Unidades de Salud de la Familia (USF) que distan a menos de 500 metros de los puntos de colecta de muestras ambientales analizadas en este estudio.
Como fuente de datos se recurrió a las planillas diarias de consulta de las USF y se registraron los siguientes datos: sexo, fecha de nacimiento, barrio, edad.
RESULTADOS
Las colectas fueron realizadas en dos jornadas distintas comprendidas del 17 al 22 de julio del 2014, que fueron seleccionadas debido a la baja probabilidad de lluvia (10%) pronosticada para la región para esas fechas. Las temperaturas medias ambientales fueron de 22°C y 28°C, respectivamente, con cielo parcialmente nublado y clima poco ventoso. La posición geográfica de los puntos de colecta pueden ser observados en la Figura 1 y en la Tabla 1 se resumen las características de las muestras colectadas.

Figura 1: Registro geográfico de los puntos de colecta. SAJ1 y SAJ2 corresponden a los puntos de toma de muestra del Barrio Sajonia, SAN1 y SAN2 a los del Barrio San Jerónimo, SANA1 y SANA2 a los del Barrio Santa Ana, CHACA1 y CHACA2 a los del Barrio Ricardo Brugada - Chacarita.
Los parámetros físico-químicos de las muestras se listan en la Tabla 2 y se puede observar que la temperatura de las aguas no superó los 25°C, con una media de 23°C. La mayor variación con respecto a la temperatura ambiental se dio en las muestras CHACA 1 y CHACA 2, donde las aguas se encontraban once y seis grados centígrados más frías. Todas las aguas colectadas presentaron un pH levemente ácido en el momento de la colecta.
La conductividad eléctrica es una medida de capacidad para conducir la electricidad, está relacionada con el contenido de sales disueltas y su típica conversión al cloruro de sodio disuelto se realiza suponiendo que 1 μS/cm es equivalente a cerca de 0,6 mg de NaCl por kg de agua59. En nuestro estudio, la conductividad varió entre un máximo de 495 μS/cm en la muestra tomada en zona urbana del barrio San Jerónimo (SAN-2) y un mínimo de 125,8 μS/cm en la muestra de agua del Río Paraguay tomada en Sajonia (SAJ-1). Realizando la conversión, se estima una concentración de 297 mg y 75,48 mg de NaCl por kg de agua, respectivamente. El agua pura no es un buen conductor de la electricidad (5,5 μS/m), debido a que la corriente eléctrica es transportada por los iones en solución, la conductividad eléctrica aumenta a medida que la concentración iónica se incrementa59. Siendo que el agua potable típica está en el rango de 5-50 mS/m (50 - 500 μS/cm), se puede inferir que todas las aguas colectadas se encuentran con una conductividad eléctrica similar a la del agua potable (Tabla 2).
La salinidad es el contenido de sales minerales disueltas en un cuerpo de agua, este contenido de cloruro de sodio (NaCl) es el que le da sabor y debido a eso a las aguas con bajo contenido se las denomina “agua dulce”. La salinidad del agua varía según la intensidad de la evaporación, es por eso las aguas superficiales son más saladas, y es más intensa en las zonas tropicales y menor en las zonas polares. El porcentaje medio de NaCl que existe en los océanos es de 3,5%, o sea 35 g/L de agua y el contenido de sal en agua potable es, por definición, menor a 0,05 % (< 0,5 g/L ó 500 mg/L). El agua es señalada como salobre a una concentración de NaCl de 0,5 - 30 g/L, o definida como salina si contiene de 3 a 5 % (3-5 g/L) de sal en volumen59. Las muestras de agua obtenidas de las inundaciones presentaron un valor máximo de 280 mg/L (SAN-2) y mínimo de 72,9 mg/L (SAJ-1). Considerando estas informaciones, las podemos calificar como de agua dulce, debido a que presentan valores de concentración de NaCl similares a los del agua potable (Tabla 2).
El total de sólidos disueltos (TDS, del inglés: Total DissolvedSolids) es una medida del contenido combinado de todas las sustancias inorgánicas y orgánicas contenidas en un líquido en forma molecular, ionizada o en forma de suspensión micro-granular (sol coloide)59. El contenido total de sólidos disueltos de las muestras colectadas en este estudio encontró un máximo de 350 mg/L (SAN-2) y un mínimo de 90,3 mg/L (SAJ-1). Estos datos, sumado al alto contenido microbiano registrado en todas las muestras colectadas, a través de la medición de coliformes totales y fecales, permitieron realizar la caracterización de las aguas, que presentaron mayoritariamente características de aguas de clase II y III, de una elevada carga microbiana y una extrema baja cantidad de OD propia de ambientes muy poluidos y con elevada contaminación orgánica (Tabla 2).
Tabla 2: Parámetros físico-químicos de las muestras colectadas. La temperatura es medida en grados Celsius (ºC), la conductividad es medida en micro Siemens (μS), los sólidos disueltos totales (TDS) y salinidad se miden en partes por millón (ppm) ó mg / L. Sd= sin datos.
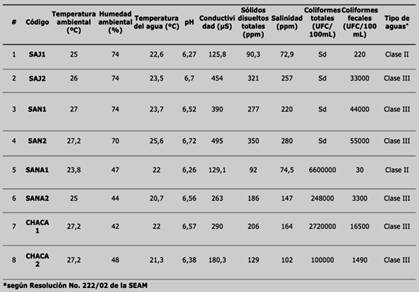

Figura 2: Registro fotográfico de los resultados de la reacción en cadena de la polimerasa (PCR) para el gen (822 bp) de la polimerasa viral de los norovirus (NoV). Gel de agarosa 2%-TAE, teñido con bromuro de etidio. Carril 1: marcador de peso molecular 100 pb.; carriles 2 al 9: SAJ-1, SAJ-2, SAN 1, SAN 2, SANA 1, SANA 2, CHACA 1 y CHACA 2, respectivamente; carril 10: control positivo NoV, carril 11: control negativo de floculación, carriles 12 al 14: controles negativos PCR. Flechas lisa y de puntos indican los productos de aproximadamente 822 pb. y 550 pb.
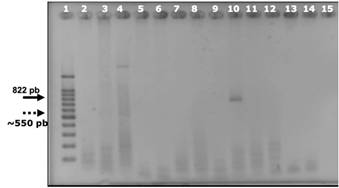
Figura 3: Registro fotográfico de los resultados de la reacción en cadena de la polimerasa (PCR) anidada para el gen (310 bp) de la polimerasa viral de los norovirus (NoV). Gel de agarosa 2%-TAE, teñido con bromuro de etidio. Carril 1: marcador de peso molecular 100 pb; carriles 2 al 5 y 8 al 11: SAJ-1, SAJ-2, SAN 1, SAN 2; y SANA 1, SANA 2, CHACA 1 y CHACA 2, respectivamente; carriles 6 y 12: controles positivos NoV de floculación; carriles 7 y 13: controles negativos de floculación; carriles 14 y 15: controles positivo y negativo de la PCR anillada.
Tabla 4: Resultados de la detección de rotavirus (RV), norovirus (NoV) y micobacterias no tuberculosas (MNT) en las muestras de agua analizadas, por el método de reacción en cadena de la polimerasa (PCR), PCR anidada y electroforesis en gel de poliacrilamida (PAGE). RdRp= gen que codifica la RNA polimerasa dependiente de RNA. GII= Genogrupo II. Los resultados negativos son indicados con un menos (-) y los positivos con un más (+).
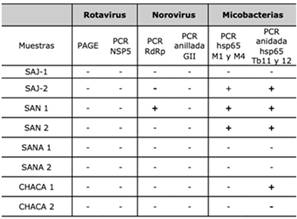

Figura 4: Registro fotográfico de los resultados de la reacción en cadena de la polimerasa (PCR) para el gen hsp65 con cebadores M1 y M4. Gel de agarosa 2%-TAE, teñido con bromuro de etidio. A. Carril 1: marcador de peso molecular de 100 pb; carril 2: control negativo de reacción; carriles 3 y 4: SAN-1 filtros de 0,7 y 0,45 µm; carriles 5 y 6: SAN-2 filtros de 0,7 y 0,45 µm; carriles 7 y 8: SAJ-1 filtros de 0,7 y 0,45 µm; carriles 9 y 10: SAJ-2 filtros de 0,7 y 0,45 µm; carriles 11 y 12: controles negativos de filtrado; carril 13: control negativo de extracción de ADN; carril 14: control positivo y carril 20 marcador de peso molecular de 50pb. B. Carril 1: marcador de peso molecular de 100 pb; carril 2: control negativo de reacción; carriles 3 y 4: SANA-1 filtros de 0,7 y 0,45 µm; carriles 5 y 6: SANA-2 filtros de 0,7 y 0,45µm; carriles 7 y 8: CHACA-1 filtros de 0,7 y 0,45 µm; carriles 9 y 10: CHACA-2 filtros de 0,7 y 0,45 µm; carriles 11 y 12: controles negativos de filtrado; carril 13: control negativo de extracción de ADN; carril 14: control positivo y carril 20 marcador de peso molecular de 50pb.
Tabla 5: Resultados de la detección de rotavirus (RV), norovirus (NoV), adenovirus entérico (AdV) y astrovirus (AstV) en las muestras de agua analizadas, por el método de reacción en cadena de la polimerasa (PCR) en Tiempo Real (Fast-TrackDiagnostics). Los resultados negativos son indicados con un menos (-) y los positivos con un más (+). Ct= Cycle Threshold (Ciclo umbral de detección); CP= control positivo; CN= control negativo.

En la BAR se revisaron 2.881 registros, de estos cumplían con la definición de casos operativa 46 pacientes cuya tasa de ataque fue de 1,6%.
El mayor número de casos ocurrió en el barrio Ricardo Brugada (Chacarita). En la primera semana de estudio se registró la mayor cantidad de casos de GEA. El sexo femenino predominó, el promedio de edad fue de 16 años con un rango de 3 meses a 83 años, el síntoma de mayor manifestación fue la diarrea seguido de fiebre.
Tabla 6: Características epidemiológicas de los casos con gastroenteritis aguda, Asunción, 2014.
| N=46 | ||||
|---|---|---|---|---|
| Variables | N | % | Rango (años) | |
| Edad (media) | 16 | - | (0 - 83) | |
| Sexo femenino | 26 | 57 | ||
| Síntomas | ||||
| Diarrea | 46 | 100 | ||
| Fiebre | 22 | 48 | ||
| Dolor abdominal | 21 | 46 | ||
| Barrios | ||||
| Ricardo Brugada | 17 | 37 | ||
| Sajonia | 15 | 33 | ||
| San Felipe | 2 | 4 | ||
| San Jerónimo | 1 | 2 | ||
| Santa Ana | 11 | 24 | ||
| Total | 46 | 100 |
DISCUSIÓN
En este estudio se analizó la presencia de RV, NoV, AstV, AdV entérico y MNT en aguas superficiales provenientes de zonas afectadas por las inundaciones producidas durante los meses de junio a agosto del año 2014 la ciudad de Asunción, Paraguay; y se presentan, además, resultados del análisis de casos de diarrea, fiebre y dolor abdominal registrados en los centros de salud más cercanos a los puntos de colecta, como datos epidemiológicos de interés.
Con respecto a la detección viral de RV por métodos clásicos de biología molecular, no se observó el patrón electroforético característico de los 11 genes del rotavirus en la electroforesis en gel de poliacrilamida. En la reacción en cadena de la polimerasa para el gen conservado que codifica para la NSP5 de los RV, no se produjo la amplificación del segmento de gen característico de 1000 pb. aproximadamente. Sin embargo, en la RT-PCR para el gen conservado que codifica para la RdRp de los calicivirus se produjo la amplificación parcial del gen y se observó una banda de aproximadamente 550 bp., diferente a la banda de 822 bp. característica de la reacción en la muestra SAN-1. A pesar de esto, no se produjo la amplificación del fragmento interno a éste, en la PCR anidada, dado que no se observó la banda de 310 pb. característica de la reacción. Esto pudo haberse debido a sustancias inhibitorias de la reacción de la polimerasa presentes en la muestra o a los propios límites de detección de la técnica.
A pesar que los protocolos moleculares, a diferencia de métodos tradicionales basados en el cultivo, no distinguen entre organismos viables y no viables, la PCR convencional es una técnica altamente específica y muy sensible para la detección de patógenos en muestras ambientales; especialmente, considerando que la técnica de cultivo celular aplicada a la detección de algunos virus es inviable o muy compleja60.
En el estudio de muestras ambientales, sin embargo, es necesaria la aplicación de metodologías de concentración, debido a que en muchos ambientes la cantidad de partículas virales es muy baja o se encuentran muy diluidas en el cuerpo de agua, como es el caso de las aguas de inundación(46). La PCR en Tiempo real ha presentado sensibilidad incluso mayor, en mucho casos, a la del método de cultivo bacteriano o que el ensayo de placa viral, se ha consolidado rápidamente en el estudio de matrices ambientales61. Además, la PCR cuantitativa con transcriptasa inversa (qRT-PCR), que utiliza ARN como molécula molde, se basa en la amplificación de un segmento del genoma utilizando colorantes o sondas fluorescentes para su detección, lo que conduce a una mayor especificidad y sensibilidad.
Para aumentar la sensibilidad de la metodología y poder realizar una mejor detección viral en las muestras analizadas, se realizó la RT-PCR en Tiempo Real utilizando un Kit de reacción comercial específico para la amplificación de genes de rotavirus del grupo A, norovirusgeno grupo I y II, astrovirus y adenovirus entérico (Fast-track Diagnostics, Luxemburgo). Esta metodología permitió confirmar la detección específica de norovirus de los genogrupos I y II en la muestra SAN-1, además de permitir la amplificación genética de adenovirus entérico humano (Ct=21), no detectado en la muestra por la RT-PCR convencional. Con respecto a las demás muestras, se logró la detección de rotavirus grupo A en la muestra SAN-1 (Ct=34); astrovirus y adenovirus entérico humano en las muestras SAJ-2 con Ct= 31 y Ct=21, respectivamente; y SAN-2 con Ct= 29 y Ct= 24, respectivamente. Estos resultados indican que la metodología de RT-PCR en Tiempo Real, con la previa aplicación de una metodología de concentración viral, resulta ideal y elemental para la detección viral en aguas superficiales de inundaciones, con respecto a las metodologías convencionales de biología molecular.
Teniendo en cuenta resultados anteriores obtenidos en nuestro laboratorio, para la detección de micobacterias, se realizó el macerado de los filtros previo a la extracción por el método orgánico con CTAB55,56;y no por métodos químicos buscando una mayor recuperación del material genético, para posteriormente someterlo a PCR de una región del gen hsp65, observándose amplificación con los cebadores externos M1 y M4 de ADN micobacteriano en las muestras colectadas en San Jerónimo tanto en SAN-1 región de viviendas donde existía agua estancada producto de la inundación, sobre la calle San Agustín esq. Estela Maris, como en SAN-2 agua superficial colectada del curso del canal “Arroyo Jaén” y en Sajonia la muestra SAJ-2 zona de viviendas donde existía agua estancada producto de la inundación de la calle Capitán Bozzano.
Mientras que con los cebadores internos Tb 11 y Tb 12 a las muestras citadas anteriormente se adiciona la amplificación en la muestra CHACA- 1 de la zona de viviendas de la calle Mompox esq. Zayas del barrio de la Chacharita donde también existía agua estancada producto de la inundación. La amplificación se dio en el ADN extraído a partir de la membrana de 0,45 µm que es la de menor porosidad por lo que posee mayor retención que en el filtro de 0,7 µm, logrando de este modo aumentar la sensibilidad de la técnica aplicando la PCR anidada. Cabe resaltar que con los cebadores Tb11 y Tb12 se logra amplificar un fragmento del gen hsp65 presente en todas las especies micobacterianas descriptas hasta la fecha y que mediante cortes con enzimas de restricción se podrían identificar a más de 140 especies micobacterianas62.
Si bien la mayoría de las micobacterias ambientales puede crecer en un medio mínimo de sales en casi la misma tasa en la que crecen en M7H9 o M7H10, algunas especies de micobacterias ambientales pueden requerir una fuente exógena de ácidos grasos para el crecimiento. El cultivo de los filtros en medio liquido podría utilizarse para individualizar las colonias micobacterianas, sin embargo, en trabajos anteriores se describe que se debe tener mucho cuidado a la hora de la elección del medio de cultivo debido que se podría estar inhibiendo el crecimiento de alguna de las especies dependiendo de los componentes que contengan los diferentes medios que podrían ser utilizados62,63.
Con respecto a los datos epidemiológicos registrados en los servicios de salud seleccionados, se encontró una baja incidencia en el número de casos de GEA; si bien la resolución de problemas de urgencia en atención primaria ha mejorado considerablemente, hay que precisar que los medios que disponen varias USF para resolver estos problemas urgentes son limitados por lo que muchos de los pacientes deciden acudir a servicios de mayor complejidad, se presume que la baja incidencia observada podría verse aumentada si se revisan los registros de los hospitales de mediana o alta complejidad del área64,65.
De entre los casos, los niños menores de 5 años fueron los más afectados lo que coincide con el reporte de la OMS donde se menciona que las enfermedades diarreicas son la segunda mayor causa de muerte de niños menores de cinco años66.
Varios estudios reportan la aparición de brotes de enfermedades infecciosas en periodos posteriores a eventos climáticos adversos, especialmente de lluvia pesada e inundaciones, seguido de sequías67,68. En las regiones tropicales y subtropicales incluso las enfermedades endémicas y transmitidas por alimentos como la hepatitis han generado brotes debido a la exposición al agua contaminada y la inundación, tanto en países en desarrollo como en regiones desarrollada67,68. En este contexto, el sistema de manejo de aguas residuales y alcantarillado público cobra extrema importancia, dado que su desbordamiento durante eventos de precipitación extrema a menudo podría presentar riesgos de propagación de patógenos en las áreas y comunidades afectadas. En nuestro estudio se reporta la existencia de situaciones similares en las muestras SAJ-2, SAN-1 y SAN-2, relativas a entornos urbanos de barrio con alcantarillado desbordado y aguas residuales expuestas. Si bien existió la limitación de acceso a muestras de eventos de gastroenteritis o enfermedades asociadas a MNTs en pobladores de la zona; se ha registrado una susceptibilidad intrínseca en la exposición a eventos de inundación, especialmente en poblaciones ribereñas, usualmente en situación de pobreza (Ricardo Brugada - Chacarita) y que las personas más afectadas son niños menores a 5 años de edad (41%, 19/46).
En conclusión, en este trabajo se rescatan datos del análisis de las muestras colectadas en los periodos de inundación debido a las precipitaciones intensas que sufrió la ciudad de Asunción de junio a agosto del 2014 y reportamos la presencia y circulación de agentes infecciosos en los sitios de colecta, tanto MNTs como virus de gastroenteritis humana, de diversos tipos y genogrupos: rotavirus, norovirus (genogrupos I y II), astrovirus, adenovirus entéricos. Además, se evaluaron las metodologías para la detección concluyendo que la PCR en tiempo real y la PCR-asociada al análisis de restricción enzimática (PRA), aplicadas luego de metodologías de concentración, son elementales y poseen buena sensibilidad para la detección de los agentes infecciosos estudiados. Estas metodologías tienen amplia aplicación ya que pueden ser utilizadas para la vigilancia ambiental de agentes infecciosos de interés para la salud humana.