INTRODUCCIÓN
La endoftalmitis infecciosa es una rara pero severa inflamación de los tejidos intraoculares, que resulta de la introducción de microorganismos en el segmento posterior del ojo, requiere de atención médica inmediata, e incluso con la intervención terapéutica y quirúrgica, puede conducir a la pérdida parcial o completa de la visión1. Según el mecanismo de adquisición, la endoftalmitis se clasifica en: postoperatoria (aguda o de aparición tardía), postraumática, endógena y otras en las que se incluye la secundaria a la queratitis2. La endoftalmitis postoperatoria es la presentación más común y se asocia frecuentemente con la cirugía de cataratas. Los patógenos causales generalmente son originarios de la microbiota conjuntival normal y los párpados, siendo las bacterias gram-positivas más frecuentes (94,2%) que las gram-negativas (6,5%) en los casos agudos de endoftalmitis postoperatorias, principalmente los estafilococos, estreptococos y enterococos3. La endoftalmitis postraumática es también causada principalmente por la flora ocular normal, siendo los estafilococos y B. cereus los más comunes4,5. La endoftalmitis endógena es menos frecuente, 2 a 8% de los casos de endoftalmitis y cualquier patógeno que causa bacteriemia o sepsis puede causar endoftalmitis al tener acceso al segmento posterior del ojo, especialmente en huéspedes inmunocomprometidos o adictos a drogas intravenosas6.
El diagnóstico microbiológico de la endoftalmitis se realiza de forma rutinaria mediante coloración de Gram y cultivo de muestras tomadas del humor vítreo y humor acuoso. Sin embargo, las técnicas microbiológicas convencionales son a menudo insuficientes para confirmar los casos clínicos sospechosos de endoftalmitis debidos a la baja sensibilidad y al tiempo que demora. La falta o retraso de una confirmación por un laboratorio microbiológico puede conducir a la mala utilización de la terapia farmacológica oftalmológica con toxicidad ocular potencial, así como al uso empírico de antibióticos de amplio espectro durante varios días, retrasándose el inicio del tratamiento antibiótico correcto hasta la identificación definitiva del microorganismo causal7-9.
La aplicación de las técnicas de biología molecular en microbiología clínica, tales como la reacción en cadena de la polimerasa (PCR), hace posible la detección de microorganismos difíciles de detectar por métodos microbiológicos tradicionales y reducir el tiempo de confirmación por el laboratorio, esencial en la caracterización de los microorganismos implicados en las infecciones graves y de rápido desarrollo10.
Varios autores mostraron cómo la utilización de la PCR en la detección directa de patógenos a partir de muestras de humor acuoso y vítreo podría afectar efectivamente el diagnóstico de endoftalmitis bacteriana 11-14. Carroll et al.15 estandarizaron una reacción de PCR anidada para diferenciar microorganismos Gram positivos de Gram negativos. La reacción consistió en un par de cebadores universales en la primera reacción, y una combinación de cebadores universales y gram-específico en la segunda ronda de la reacción. Numerosas otras técnicas han sido probadas para el diagnóstico de las endoftalmitis con resultados variables, la PCR convencional y a tiempo real, así como las reacciones post-PCR tales como RFLP, hibridación de la sonda de ADN y la secuenciación del ADN se han utilizado con éxito para la elucidación de diagnóstico de casos clínicamente sospechosos de endoftalmitis bacterianas, por lo que la aplicación de técnicas moleculares es prometedora para utilizar en el diagnóstico de rutina de las endoftalmitis16-18.
Ante la necesidad de contar con un método eficaz para el diagnóstico etiológico de las endoftalmitis infecciosas en nuestro país, se realizó este estudio para evaluar el método microbiológico convencional que consta de coloración de Gram, cultivo e identificación y una técnica de PCR anidada basada en la desarrollada por Carroll et. al15 en pacientes provenientes de centros oftalmológicos de Paraguay, a fin de aumentar sustancialmente la confirmación de los casos sospechosos de endoftalmitis infecciosa, mejorar el tratamiento correcto y oportuno y por ende la evolución del paciente.
METODOLOGÍA
Se realizó un estudio observacional descriptivo de corte transverso en pacientes con sospecha de endoftalmitis que consultaron entre enero de 2014 a julio de 2018 en tres centros oftalmológicos de Asunción: Cátedra de Oftalmología del Hospital de Clínicas, Fundación Visión y Banco de Ojos.
Toma de muestra
La muestra de humor vítreo fue recolectada de los pacientes con sospecha de endoftalmitis en condiciones estériles, por los médicos oftalmólogos responsables de cada centro, con aguja estéril 27-gauge (0.33-mm de diámetro). Posterior a una inyección subconjuntival de anestesia, una abertura vítrea fue realizada y la aguja introducida a través de la pars plana 3 mm detrás del limbo en ojos afaquicos y 4 mm detrás del limbo en ojos faquicos. Fue aspirado un volumen de 200 a 500 µl y enviado dentro de las dos horas al laboratorio de Microbiología del Instituto de Investigaciones en Ciencias de la Salud.
Estudio microbiológico
Una vez recibida la muestra fue alicuotada y una parte transferida a un microtubo estéril (50 a 100 µl) y almacenada a -20°C para ser utilizada posteriormente para el diagnóstico etiológico por PCR. La parte restante de la muestra fue inoculada en agar sangre de carnero al 5%, agar chocolate, caldo tioglicolato, agar sabouraud y una gota depositada en lámina estéril para coloración de Gram. La placa de agar sangre y el caldo tioglicolato se incubaron en condiciones aeróbicas a 35°C, el agar chocolate en anaerobiosis a 35°C y el agar sabouraud a 28°C. Los cultivos fueron incubados por 15 días.
Identificación de microorganismos y pruebas de sensibilidad
La identificación de especies fue realizada por los métodos microbiológicos convencionales, mediante pruebas bioquímicas (catalasa, manitol, DNAsa, urea, bilis esculina, prueba de CAMP, oxidasa, TSI, citrato, decarboxilación de la lisina y ornitina, indol, rojo de metilo, Voges-Proskauer) y pruebas de aglutinación19. La identificación de estafilococos coagulasa-negativo realizado según el diagrama de identificación de Paulis et al., con algunas modificaciones20 y en los casos en que no fue posible la identificación de algún microorganismo por los métodos convencionales, se utilizó el microsistema comercial API. Además se realizó la prueba de sensibilidad por el método de difusión con discos según Kirby Bauer, en placas de Müeller Hinton a 35°C e interpretó de acuerdo a las normas del CLSI (Clinical Laboratory Standard Institute)21. Se utilizaron cepas de referencia Staphylococcus aureus ATCC 25923, Enterococcus faecalis ATCC 29212, Pseudomonas aeruginosa ATCC 27853 y Escherichia coli ATCC 25922, para controlar la calidad de las pruebas de identificación y del antibiograma.
A medida se obtenían los resultados microbiológicos, coloración de Gram, identificación y prueba de sensibilidad se informaron al médico tratante.
Obtención y cuantificación de ADN de cepas de referencia: Las cepas de referencia utilizadas para la estandarización fueron Staphylococcus aureus ATCC 25923, Streptococcus pneumoniae ATCC 49619 y Escherichia coli ATCC 25922. Las cuales se inocularon en agar sangre por 24 a 48 horas para realizar la extracción de ADN, la cual se llevó a cabo con un kit comercial Wizard® Genomic DNA (PROMEGA, Madison, WI, USA) a partir de las colonias frescas. Posteriormente se realizaron diluciones seriadas del ADN extraído para ensayar la sensibilidad del método. El ADN fue cuantificado por Nanodrop.
Extracción de ADN de las muestras de Humor Vítreo: Se utilizó un volumen de 50 uL de humor vítreo para la extracción de ADN con el kit comercial Wizard® Genomic DNA (PROMEGA, Madison, WI, USA) siguiendo las indicaciones del fabricante. El ADN fue conservado a -20ºC hasta la realización de la PCR.
PCR anidada de las muestras de humor vítreo: Una vez descongeladas las muestras conservadas a -20ºC, se utilizaron alícuotas de 2 µL de ADN en la primera corrida de PCR. Los controles positivos utilizados fueron 10 ng de ADN de cepas ATCC E. coli y S. aureus. Posteriormente se realizó la PCR anidada descrita por Carroll et al. (2000)15 con algunas modificaciones en las condiciones de reacción como aumento del número de ciclos en la primera ronda y de los tiempos de reacción en ambas rondas.
Primera ronda de amplificación: Se utilizó 0,5 µM de cebadores 16SF/16SR, 0,4 mM de desoxinucleótido trifosfato (dNTP) Invitrogen (Thermo Fisher Scientific - USA), 3 mM de MgCl2 en un volumen final de 25 µL. La reacción se llevó a cabo con un ciclo de desnaturalización inicial a 94°C por 5 minutos, seguido de 94°C por 30 s, 54°C por 30 s y 72°C por 1 min por 30 ciclos, y un ciclo de extensión final de 72°C por 5 min
Segunda ronda de amplificación: Se realizó una PCR multiplex a partir de 1 µL del amplicón de la primera ronda con 0,5 µM de los cebadores NF/NR/P2F, 3 mM de MgCl2 en un volumen final de 25 µL. La reacción se llevó a cabo con un ciclo de desnaturalización inicial a 94°C por 5 minutos, seguido de 94°C por 30 s, 60°C por 30 s y 72°C por 1 min por 30 ciclos, y un ciclo de extensión final de 72°C por 5 min.
Electroforesis y revelado: Como resultado de la segunda ronda de PCR se obtuvo un amplicón único de 1025 pb para bacterias Gram Negativas y dos amplicones de 1025 pb y de 355 pb para bacterias gram Positivas, estos productos fueron visualizados por electroforesis en gel de agarosa al 2% previa tinción con SYBR® Safe DNA gel stain (Invitrogen, USA).
Gestión y análisis de los datos
Los datos de los cuestionarios fueron consignados en una planilla electrónica (Excel) y posteriormente analizados utilizando el paquete estadístico Epi-Info 2002.
Aspectos éticos
El protocolo de investigación fue aprobado por el Comité de Ética en Investigación del IICS-UNA. El estudio se ajustó a los principios éticos de la investigación clínica. No se trabajaron directamente con los pacientes sino con las muestras de los mismos que fueron tomadas por los oftalmólogos como parte de la rutina de los servicios. Se mantuvo la confidencialidad de los datos, los estudios fueron gratuitos y los resultados por métodos convencionales remitidos a los médicos tratantes para el tratamiento adecuado.
RESULTADOS
Se estudiaron 51 muestras de humor vítreo de pacientes provenientes de 3 Centros Oftalmológicos de Paraguay; Hospital de Clínicas, Fundación Visión y Banco de Ojos. El rango de edad de los pacientes fue de 24 a 77 años, predominaron los pacientes del sexo masculino, en 72,5% (37/51).
El origen de las endoftalmitis solamente pudo ser establecido en 31 pacientes (60%), entre los cuales predominó el antecedente de trauma con diversos objetos como cuchillos, piedras, alambres, clavos, madera etc., en 15 pacientes (48%), seguido de aquellos con antecedente de cirugía en 8 pacientes (26%), en tercer lugar las presumiblemente endógenas 5 (16%) y posterior a queratitis 3 casos (10%).
Del total de muestras analizadas, 12 dieron cultivo positivo, en 11 desarrollaron bacterias y en 1 muestra desarrolló un hongo filamentoso. Entre las bacterias predominaron las Gram Positivas 73% (8/11). De las muestras con cultivo positivo 9 presentaron examen microscópico positivo. En la Tabla 1 se presenta los microorganismos aislados y el resultado obtenido en la Coloración de Gram.
Tabla 1: Gérmenes aislados en los cultivos y resultados en la Coloración de Gram
| Coloración de Gram | Germen aislado |
|---|---|
| Cocos Gram (+) | Streptococcus sp (Grupo viridans) |
| Cocos Gram (+) | Streptococcus sp (Grupo viridans) |
| No se observan gérmenes | Enterococcus faecalis |
| Cocos Gram (+) | Enterococcus faecalis |
| Cocos Gram (+) y Bacilos Gram (+) | Enterococcus casseliflavus y Bacillus sp |
| No se observan gérmenes | Enterococcus casseliflavus |
| No se observan gérmenes | Staphylococcus epidermidis |
| Bacilos Gram (-) | Pseudomonas stutzeri |
| Bacilos Gram (-) | Stenotrophomonas maltophilia |
| Bacilos Gram (-) | Serratia marcescens |
| Bacilos Gram (+) | Bacillus sp |
| Hifas septadas | Aspergillus fumigatus |
La técnica de la PCR utilizada se basó en el protocolo de Carroll et al., al cual se introdujeron modificaciones en las condiciones de reacción, como en el aumento del número de ciclos en la primera ronda y de los tiempos de reacción en ambas rondas según como se lo ha descripto. No se logró la amplificación con el cebador N6R específico para bacterias Gram Negativas (NF/N6R), por lo tanto se realizó la PCR con los amplicones de 355 pb para las bacterias Gram Positivas y 1025 pb para los Gram Negativos. En la Tabla 2 se muestra la sensibilidad obtenida.
Tabla 2: Sensibilidad de la detección de la PCR anidada
| Cepas de referencia utilizadas | Límite de detección obtenido |
|---|---|
| Staphylococcus aureus | hasta 3.10-5 ng/ul |
| Streptococcus pneumoniae | hasta 2,5.10-3 ng/ul |
| Escherichia coli | hasta 6.110-7 ng/ul |
Los productos de la PCR anidada visualizados por electroforesis en gel de agarosa al 2% previa tinción con SYBR®, se pueden apreciar en la Figura 1.
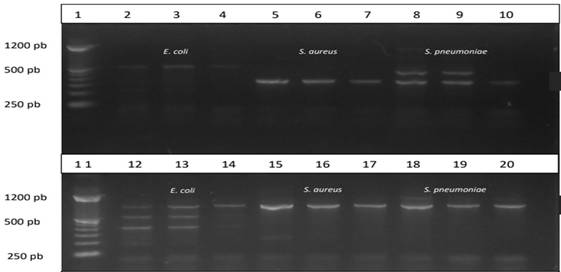
Figura 1: Productos de la PCR anidada visualizados por electroforesis en gel de agarosa al 2% previa tinción con SYBR® Safe DNA gel stain. Carriles 1 al 10: Mix P2F/NR (Gram positivo-355 pb): 1-Molecular marker; 2-E. coli 6,0 ng/uL ; 3-E. coli 0,6 ng/uL; 4-E. coli 0,06 ng/uL; 5-S. aureus 3,0 ng/uL; 6-S. aureus 0,3 ng/uL; 7-S. aureus 0,03 ng/uL; 8-S. pneumoniae 25,0 ng/uL; 9-S. pneumoniae 2,5 ng/uL; 10-S. pneumoniae 0,25 ng/uL. Carriles 11 al 20: Mix NF/NR (PanBacteria Universal 1025 pb): 1-Molecular marker; 2-E. coli 6,0 ng/uL; 3-E. coli 0,6 ng/uL; 4-E. coli 0,06 ng/uL; 5-S. aureus 3,0 ng/uL; 6-S. aureus 0,3 ng/uL; 7-S. aureus 0,03 ng/uL; 8-S. pneumoniae 25,0 ng/uL; 9-S. pneumoniae 2,5 ng/uL; 10-S. pneumoniae 0,25 ng/uL.
Por la técnica de PCR, se obtuvieron 26 muestras positivas, siendo la tasa de detección del 51%. En 15 muestras que resultaron negativas por cultivo se logró la detección de ADN bacteriano. Solo una muestra positiva por cultivo, en la cual desarrolló un hongo, no pudo ser detectada por la PCR utilizada. La discriminación en Gram positivos y Gram negativos, con respecto a lo obtenido en los cultivos coincidió en un 100%. También por esta técnica resultaron predominante los Gram positivos, 22/26 (84,6%). Tabla 3
Tabla 3: Resultado comparativo por ambos métodos
| N° de aislados n=51 | Cultivo | PCR |
|---|---|---|
| 8 | Positivo para Grampositivos | Positivo Gram (+) (355bp) |
| 3 | Positivo para Gramnegativos | Positivo Gram (-) (1025bp) |
| 1 | Positivo para hongo | Negativo |
| 14 | Negativo | Positivo Gram (+) (355bp) |
| 1 | Negativo | Positivo Gram (-) (1025bp) |
| 24 | Negativo | Negativo |
La tasa de detección por método microbiológico fue de 23,5% y por el método molecular 51,0%; la sensibilidad fue 91,7% y la especificidad 61,5%. Tabla 4
DISCUSIÓN
Los estudios microbiológicos convencionales para el diagnóstico de las endoftalmitis mediante cultivo de humor vítreo, humor acuoso y coloración de Gram, tienen el gran inconveniente de presentar baja sensibilidad. Según los diversos estudios, los cultivos muestran una positividad variable, entre 24% a 56% de los casos sospechosos16,22-24. En el presente trabajo un 23,5% de las muestras analizadas resultaron ser positivas por el método convencional. La baja sensibilidad puede deberse a diversos factores tales como: escasa cantidad de la muestra, fijación de los microorganismos en superficies sólidas (lente intraocular, fragmentos de cristalino, cápsula) y la consiguiente disminución de las células en el humor vítreo / acuoso, el uso de antibióticos antes de la toma del material clínico y la presencia de microorganismos exigentes. Además los métodos de diagnóstico por cultivo requieren la presencia de microorganismos viables para que pueda ser detectado.
La aplicación de las técnicas de biología molecular en microbiología clínica, como la reacción en cadena de la polimerasa (PCR), estableció una nueva era en la detección y caracterización de microorganismos. Varios autores mostraron que la utilización de la PCR en la detección directa de patógenos a partir de muestras de humor acuoso y vítreo podría mejorar el diagnóstico etiológico de las endoftalmitis con un rango de sensibilidad de detección de entre 57% a 100%11-14. En este estudio la tasa de detección por el método molecular fue del 51%, este método permitió confirmar la presencia de bacterias en 14 muestras con cultivo negativo, mejorando la tasa del diagnóstico microbiológico en un 27,5%.
La detección de patógenos por PCR sólo depende de la presencia de ADN bacteriano en la muestra, independientemente si existen o no microorganismos viables, esta característica es una de las ventajas de esta técnica sobre el cultivo, por lo cual es útil, especialmente para líquidos biológicos en los cuales la microbiota normal no está presente y en aquellos casos en que se ha iniciado tratamiento antimicrobiano. Además la utilización de métodos moleculares permite el diagnóstico de microorganismos fastidiosos, de difícil detección por métodos microbiológicos tradicionales, y constituyen una excelente opción para mejorar la tasa y reducir el tiempo de diagnóstico de las endoftalmitis, especialmente en casos severos, en los cuales es crucial iniciar oportunamente la terapia apropiada, ya que de ello depende la evolución favorable del paciente26. En el presente estudio, con la técnica molecular utilizada el tiempo necesario para la confirmación de la etiología bacteriana fue de aproximadamente de 5 h, mucho menor en comparación con el tiempo mínimo necesario para el diagnóstico por cultivo, que fue de 48 h. Si bien en muchos de los casos con cultivo positivo, la microscopía fue capaz de establecer un correcto diagnóstico presuntivo inicial.
La detección del genoma eubacterial por el método de PCR ha sido desarrollado basado en las regiones conservadas de la secuencia del ADNr/ARNr 16S de E. coli por varios grupos27,28. La subunidad pequeña del rRNA contiene segmentos que se conservan a nivel de especie, género y reino. Cebadores universales elegidos del 16S rDNA tienen una gran cantidad de información de secuencia y regiones altamente conservadas del gen lo cual permite la síntesis de cebadores que amplifican una gran variedad de bacterias. La detección de ADN bacteriano utilizando cebadores universales es útil como prueba de diagnóstico rápida para determinar la etiología bacteriana, pero no proporciona información en relación con la tinción de Gram y la identificación de las especies involucradas en cada caso13.
Anand et al. han intentado la discriminación entre gram positivos y negativos sometiendo los productos de PCR del gen del ARNr 16S a hibridación con sondas de ADN capaces de diferenciar los productos obtenidos en gram positivos y gram negativos. Estos autores evaluaron 57 muestras de pacientes con diagnóstico clínico de endoftalmitis, la positividad de los métodos microbiológicos (cultivo y microscopía) fue 56,1%, la de PCR seguida de hibridación fue de 91,2%, y la diferenciación entre los microorganismos gram positivos y gram negativos mostró un 100% de correlación cuando se comparó los resultados de hibridación con los de microscopía y cultivo16. En el presente trabajo se ha obtenido total correspondencia en la discriminación del Gram comparando el cultivo con la técnica de PCR, coincidiendo con otros autores13,15,16. La confirmación del Gram es sumamente útil, pues con ello se consigue orientar el tratamiento antibiótico adecuado.
Carroll et al. lograron discriminar Gram Positivos de Gram Negativos mediante una PCR anidada, empleando un par de cebadores universales en la primera ronda de la reacción y una combinación de cebadores universales y gram-específico en la segunda ronda, detectando como producto de amplificación de los patógenos gramnegativos una banda de 985 pb, y en la PCR múltiplex como producto de amplificación del genoma bacteriano y de los grampositivos, bandas de 1025 pb y 355 pb respectivamente15. La PCR utilizada en el presente trabajo está basada en el protocolo de estos autores, pero fue necesaria la introducción de modificaciones en las condiciones de reacción, aumento del número de ciclos en la primera ronda y de los tiempos de reacción en ambas rondas, sin embargo no se pudo lograr la amplificación con el cebador N6R específico para bacterias Gram Negativas, por lo se realizó la PCR considerando los amplicones de 355 pb para las bacterias Gram Positivas y en los casos en que solamente se detectó la banda de 1025 pb y no se consiguió amplificar gram positivos, el producto fue considerado como Gram negativo.
Numerosas otras técnicas han sido probadas para el diagnóstico de las endoftalmitis con resultados variables, tanto por PCR convencional y PCR a tiempo real, así como las reacciones post-PCR tales como análisis de restricción de productos de PCR por RFLP (polimorfismo de longitud de fragmento de restricción), hibridación con sondas de ADN o secuenciación directa de estos producto16-18,29.
La aplicación de la PCR en tiempo real para el diagnóstico de la endoftalmitis podría ser de gran utilidad. Esta técnica combina la amplificación y detección de una secuencia blanco de ADN utilizando sondas específicas18. Tiene una alta sensibilidad y elimina pasos posteriores a la PCR, obteniéndose resultados en menos tiempo. Bispo et al. aplicaron un sistema de PCR a tiempo real en muestras clínicas intraoculares para la detección y clasificación bacteriana. Obtuvieron cultivos positivos en el 47,6% y con la PCR a tiempo real en el 95,2%29.
Para la identificación de la especie del agente causal por el método molecular, la secuenciación es una de las técnicas post PCR muy empleada, pero requiere de un volumen importante de producto amplificado lo que no siempre es posible debido al bajo inóculo bacteriano del fluido intraocular. La secuenciación permite conocer el género y especie pero no así la sensibilidad del agente bacteriano frente a los antibióticos. Ecker et al. han desarrollado una plataforma que utiliza espectrometría de masas de los productos de PCR ionizadas (incluidos los productos de gen del ARNr 16S), que es capaz de integrar en el mismo sistema, la identificación del microorganismo, genotipado, y la detección de la virulencia y los genes de resistencia a antibióticos30.
Con respecto al origen de los casos de endoftalmitis de esta serie, si bien no conocemos en su totalidad, se cuenta con los antecedentes del 60% de los pacientes entre los cuales predominaron las endoftalmitis posterior a traumas en aproximadamente el 50%, seguidos de los casos postquirúrgicos,y en tercer lugar los de origen endógeno. Esto constituye una limitante del presente estudio, pues no se pudieron recabar todos los datos que podrían haber aportado mayor información.
Las bacterias gram positivas son las que se reportan como agente etiológico de las endoftalmitis más frecuentemente, tanto en las postoperatorias como postraumáticas, en las cuales los patógenos causales generalmente son originarios de la flora conjuntival normal y los párpados4-6. Coincidente con la literatura hemos aislado con mayor frecuencia Gram positivos entre ellos; Enterococus spp, Streptococcus sp, Staphylococcus sp y Bacillus sp y como ya se ha mencionado predominantemente de casos posteriores a traumas y cirugías.
Otra bacteria gram positiva reportada frecuentemente como causante de endoftalmitis, principalamente en las postquirúrgicas tardías y que no la hemos aislado en este estudio es Propionibacterium acnes, Therese et al. utilizando cebadores universales para el gen bacteriano ARNr 16S y un conjunto de cebadores específicos de especie para P. acnes, detectaron en 9 de 58 muestras (15,5%)13, y por cultivo obtuvieron 27 aislados (46,5%), 20 bacterias (34,5%) y 7 hongos (12%), entre las bacterias no aislaron P. acnes. Considerando estos hallazgos podría ser necesario el agregado de cebadores específicos para P. acnes. Con respecto al cultivo para hongos, obtuvimos un aislado de un hongo filamentoso, el cual como era de esperar dio negativo por PCR, pues el método utilizado no es capaz de detectar ADN fúngico. Este caso correspondió a una endoftalmitis endógena en un paciente con severo inmunocompromiso.
Por el método convencional además se pudo determinar la sensibilidad de las bacterias que desarrollaron en el cultivo frente a los antibióticos. La gran mayoría de las bacterias aisladas presentaron buena sensibilidad frente a los antibióticos (datos no mostrados), si bien algunos de los aislados como Enterococcus faecalis y Stenotrophomonas maltophilia presentan un patrón de sensibilidad muy particular característico del género y especie, en los cuales la identificación fue fundamental.
Debido a que la PCR anidada utilizada mejoró significativamente la sensibilidad y la rapidez para la detección bacteriana, se pretende implementar la utilización de la técnica para el estudio etiológico rutinario de los casos sospechosos de endoftalmitis infecciosas y así aumentar sustancialmente la confirmación bacteriana de las endoftalmitis, con lo que se logrará reducir las terribles consecuencias de esta patología por falta o demora del correcto diagnostico etiológico.