INTRODUCCIÓN
Escherichia coli productor de toxina Shiga (STEC) es un patógeno de importancia alimentaria mundial. Los brotes causados por este microorganismo se asocian típicamente con alimentos contaminados de origen bovino o productos vegetales contaminados con heces bovinas. Las STEC, también conocidas como E. coli verotoxigénicas (VTEC) o enterohemorrágicas (EHEC), se caracterizan por la producción de dos potentes citotoxinas llamadas toxinas Shiga 1 y 2, codificadas respectivamente por los genes stx1 y stx2. En los seres humanos estas bacterias se asocian con un amplio espectro de manifestaciones clínicas que varían desde diarrea leve y autolimitada hasta procesos más graves, muchas veces con secuelas importantes, como colitis hemorrágica (CH) o síndrome urémico hemolítico (SUH)1,2. El SUH es un cuadro clínico que se caracteriza por la tríada diagnóstica de: anemia hemolítica microangiopática, trombocitopenia e insuficiencia renal aguda (IRA). Es una enfermedad que afecta principalmente a los niños y que se presenta fundamentalmente como casos esporádicos en los países del Cono Sur, especialmente Argentina ocupando el primer lugar en el mundo en incidencia del síndrome que es alrededor de 200 a 400 casos por año3,4.
Algunos estudios previos han demostrado que ciertos aislados STEC tienen la capacidad de unir, colonizar y formar biofilm en alimentos y otras superficies5. Los biofilm son comunidades complejas de microorganismos que crecen embebidos en una matriz orgánica polimérica autoproducida y adherida a una superficie viva o inerte, aumentando su potencial como fuente de contaminación, además de reducir la eficacia de desinfección de las superficies6,7.
Las fimbrias se han asociado con la adhesión tisular de importantes cepas patógenas de E. coli como las STEC8. Las fimbrias de tipo 1 codificada por el gen fimH son las adhesinas más comunes que se encuentran en los aislamientos comensales y patógenos de E. coli, así como en otras Enterobacteriaceae. Son orgánulos de superficie adhesiva de aproximadamente 7 nm de ancho y 1 μm de longitud. El análisis de la formación de biofilm en cepas de E.coli con fimH mutada indicó que son drásticamente defectuosos en la unión inicial a superficies abióticas9. Estas observaciones ilustran que las fimbrias de tipo 1 son críticas para la unión inicial estable de célula a superficie para varias cepas de E. coli.
En la industria alimentaria es muy común la presencia de biofilm en tuberías, equipos y materiales ya que pueden formarse en cualquier tipo de superficie, incluyendo plástico, cristal, madera, metal y sobre los alimentos7.
Puesto que estas formaciones pueden contener microorganismos patógenos y presentan una mayor resistencia a la desinfección, se incrementan las probabilidades de contaminación del producto y de provocar infecciones alimentarias, razón por la que se considera que la presencia de biofilm en las superficies de contacto de la industria alimentaria constituye un evidente riesgo para la salud.
Según una investigación anterior se demostró que la frecuencia de E. coli patógena en ganado bovino paraguayo fue elevada, detectándose en más del 90% de las muestras al menos un factor de virulencia compatible con el patotipo STEC10. Sin embargo, aún no existen estudios que registren la portación del gen fimH y la capacidad de formación de biofilm en aislados STEC proveniente de ganado bovino paraguayo. En este contexto, el presente trabajo determinó mediante el uso de técnicas moleculares la frecuencia de portación del gen fimH en aislados STEC provenientes de ganado bovino, generando así los primeros datos nacionales. Esta información es de suma importancia para fortalecer medidas de control que puedan generar soluciones para combatir la formación de biofilm y permitir situarnos en la problemática que conlleva este fenómeno en las industrias alimentarias a nivel país.
MATERIALES Y MÉTODOS
Este estudio es observacional, descriptivo, de corte transverso. El protocolo de estudio cuenta con la aprobación de Comités Científicos y de Ética del IICS con código M09/17. Los aislados empleados en este estudio provinieron de un proyecto marco denominado ´´Caracterización molecular de factores de virulencia de Escherichia coli aislados del ganado bovino´´ que también cuenta con la aprobación de Comités Científicos y de Ética del IICS con el código de aprobación P06/2013. Los mismos han sido criopreservados en el biobanco del IICS. La caracterización molecular y fenotípica del proyecto marco incluyó la determinación de especie por PCR del gen 16S y pruebas bioquímicas, así como la detección por PCR de los genes stx1 y stx2. En el proyecto marco se analizaron un total de 1006 aislados de los cuáles 269 fueron identificados como STEC. Las muestras biológicas han sido manipuladas cumpliendo medidas estrictas de bioseguridad y todos los datos han sido codificados a fin de mantener la confidencialidad. Se realizó la detección del gen fimH por medio de PCR convencional. Se utilizó el protocolo descrito por Johnson et al.11 )(11) con algunas modificaciones que se describen a continuación. Las secuencias de oligonucleótidos se muestran en la Tabla 1. El producto generado fue un fragmento de ADN de 508 pb.
Las condiciones de la mezcla para cada reacción de PCR fueron los siguientes: solución buffer 1X (Bioron, Alemania), MgCl2 4 mM (Bioron, Alemania), dNTP 0,2 mM (Bioron, Alemania), primers para el gen fimH 0,6 µM (Macrogen, Corea), Taq polimerasa 1U (Bioron, Alemania). El volumen final de la reacción fue de 25 µL conteniendo 2,5 µL de muestra de ADN como molde.
Las condiciones de termociclado fueron las siguientes: 25 ciclos de 95°C por 4 minutos inicialmente, luego 95°C por 30 segundos, 63°C por 30 segundos y 72°C por 1 minuto, un paso final de 10 minutos a 72°C. Se empleó el termociclador Applied Biosystems-2720 Thermal Cycler (Singapur).
Como no se contaba con cepa control del gen fimH se realizó un sistema screening a los 269 aislados STEC para determinar la portación del gen mencionado. Se conformaron 27 pooles de un promedio de 10 aislados por pool. De los pooles de ADN que presentaron productos amplificados de tamaño esperado se sometieron al análisis individual de ADN extraído de cada aislado que conformaba el pool, con la finalidad de individualizar cuál de ellos era el portador del gen.
Para confirmar la identidad de los amplicones obtenidos en las reacciones de PCR, se secuenció por el método de Sanger un producto positivo de tamaño esperado para el gen fimH (Macrogen, Corea) y se analizó la secuencia obtenida mediante el alineamiento con las de la base de datos de GenBank. La muestra con 95 % o más de homología fue empleada posteriormente como control positivo para las reacciones de PCR.
La visualización de los productos de PCR se realizó mediante electroforesis en gel de agarosa al 2%. Para tal fin, se sembraron 5 μL de producto con loading buffer a una concentración final 1X y se realizó la corrida en cubas electroforéticas con buffer TAE (Tris, acetato, EDTA) al 1X a 120V por 40 min aproximadamente. La tinción se realizó empleando sybr safe al 10% (Invitrogen, USA).Un marcador molecular de 100 pb (Thermo Scientific GeneRuler, USA) fue utilizado para estimar el tamaño de los productos separados. Las bandas de los geles de agarosa se visualizaron mediante el uso del transluminador UV con cámara digital (UVDI, Taiwán).
RESULTADOS
El sistema de screening empleado para encontrar los aislados portadores del gen fimH en las STEC en estudio fue exitoso (Figura 1). Se analizaron 269 aislados STEC agrupados en 27 pooles, identificándose a los aislados portadores del gen fimH en cada pool positivo en las reacciones de PCR. La secuenciación de un producto representativo y su posterior alineamiento con secuencias almacenadas en la base de datos del GenBank permitió la confirmación de la presencia del gen analizado, con porcentaje de identidad del 100% (Figura 2 y Tabla 2). El aislado cuyo producto de amplificación se secuenció fue empleado como control positivo en las reacciones de PCR.
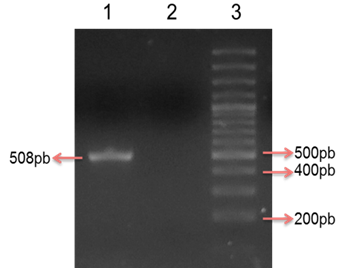
Figura 1: Amplificación por PCR del gen fimH. Electroforesis en gel de agarosa al 2% con tinción de sybr safe al 10%. Carril 1: Aislado positivo para el gen fimH (508pb), Carril 2: Control negativo, Carril 3: Marcador de peso molecular de 100 pb.
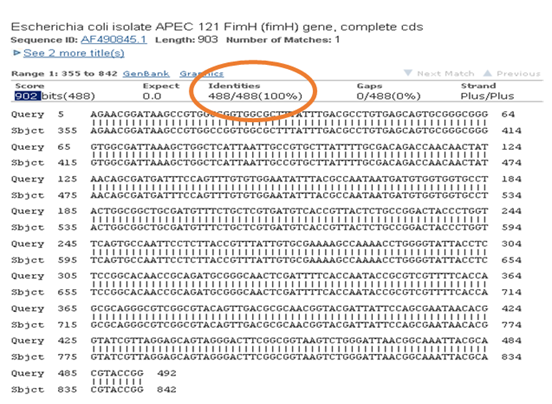
Figura 2: Confirmación de aislado a emplear como control positivo. Alineamiento de la secuencia del producto de PCR de 508 pb obtenido por amplificación de fimH con una secuencia del gen fimH de E. coli almacenada en la base de datos del NCBI, obteniéndose 100% de identidad (Código de acceso de la secuencia alineada en el GenBank: AF490854.1).
Tabla 2: Aislado STEC portador del gen fimH
| Gen | Aislado portador | Secuencia alineada* | Identidad | Código de acceso** |
| fimH | 212.9 | Escherichia coli isolate APEC 121 FimH (fimH) gene, complete cds | 100% | AF490845.1 |
*Datos obtenidos mediante el empleo de la herramienta BLAST (Basic Local Alignment Search Tool).
** Código de acceso a la secuencia en la base de datos del GenBank.
Habiendo realizado la estandarización de las condiciones de PCR fueron analizados los 269 aislados STEC para determinar la frecuencia de portación del gen fimH, resultando el 48% (n: 129) portador de dicho gen y por tanto con potencial de formar biofilm (Figura 3).
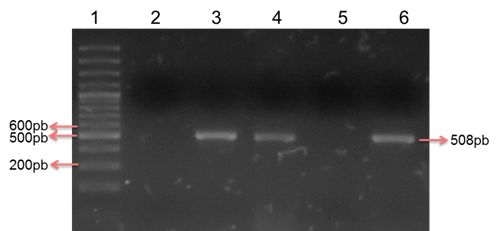
Figura 3: Detección del gen fimH en STEC provenientes de material fecal de bovinos. Electroforesis en gel de agarosa al 2% con tinción de sybr safe al 10%. Carril 1: Marcador de peso molecular de 100 pb, Carril 2: Control negativo, Carril 3: Control positivo para el gen fimH (508 pb). Carriles 4 y 6: Aislados positivos para el gen fimH. Carril 5: Aislado negativo para el gen fimH.
En adición se comparó la frecuencia de portación de los genes stx1, stx2 y stx1/stx2 con la frecuencia de portación del gen fimH en los aislados STEC. Se observó que el 40% (2/5) de los aislados STEC que portaban sólo el gen stx1 resultaron ser portadores del gen fimH, las STEC que portaban sólo el gen stx2 presentaron 54,6% (77/141) de portación de fimH y las STEC con stx1 y stx2 presentaron 40,6% (50/123) de portación del gen fimH. (Tabla 3).
Tabla 3: Frecuencia de portación de los genes stx1 y/o stx2 con respecto a la frecuencia de portación del gen fimH en aislados STEC
| Portación de stx1 y /o stx2 (N) | Portación de fimH n (%) |
| stx1+ (n: 5) | 2 (40%) |
| stx2+ (n: 141) | 77 (54,6%) |
| stx1+ stx2+ (n: 123) | 50 (40,6%) |
| Total de aislados STEC (n: 269) | 129 (48%) |
DISCUSIÓN
En Paraguay actualmente no existen investigaciones acerca del desarrollo de biofilm por parte de aislados STEC, por lo que se planteó estudiar este mecanismo de resistencia que generan las bacterias y analizar su comportamiento. En cuanto al porcentaje de portación genotípica realizada se observó una elevada frecuencia del gen en estudio, siendo el 48% de los aislados STEC portadores del gen fimH, lo que representa un alto potencial de formar biofilm. Se requiere una investigación adicional de la expresión de dicho gen para determinar su importancia relativa en la formación de biolfim. Esta frecuencia de portación se comparó con los resultados de Biscola et al. 20115 también encontraron frecuencias elevadas (98%) en cepas STEC no-O157 aisladas de bovinos. Los mismos sugieren que se debe tener en cuenta que la expresión de los genes de biofilm está en general relacionada con condiciones de cultivo particulares y que puede variar entre diferentes cepas incluso dentro del mismo serotipo. Al igual que Polifroni y Angel Villegas que presumen que la formación de biofilm no depende sólo de la información genética, sino también del ecosistema y las condiciones que rodean a la bacteria3,10-13.
Aunque los brotes de infecciones por STEC se han relacionado principalmente con comer carne molida de res mal cocida, también se han implicado una variedad de otros alimentos como vehículos. Además, puede ocurrir contaminación cruzada en las plantas procesadoras de alimentos, durante su manipulación y preparación, lo que resulta en una amplia gama de alimentos que están implicados en los brotes de infecciones por STEC. La capacidad de las bacterias para adherirse y producir biofilm en las superficies puede influir en su persistencia durante la fabricación y el comercio minorista, así como en su capacidad de causar enfermedades. Considerando a las cepas STEC patógenas asociados directamente al SUH, se fortalece la importancia en la prevención de la contaminación de cualquier alimento14.
Las características propias de estas estructuras bacterianas implican un comportamiento diferente ante los procesos de desinfección, por lo tanto, con el fin de proporcionar al consumidor un producto con riesgos mínimos es necesario considerar como estrategias futuras para controlar las STEC medidas que controlen eficazmente el biofilm14.
Si bien las muestras analizadas provienen de un solo establecimiento, esta técnica queda disponible para ser aplicada futuramente al resto de los aislados STEC del Biobanco del IICS que cuenta con el mayor número de aislados STEC del país, contribuyendo así con una herramienta esencial para seguir con la línea de investigación, obteniendo datos de suma importancia que ayuden a describir la situación de riesgo de contaminación alimentaria en que se encuentra el país.















