INTRODUCCIÓN
La leishmaniasis se define como una histoparasitosis producida por protozoos parásitos del género Leishmania, de localización intracelular (macrófagos), caracterizada por diversas manifestaciones clínicas y transmitidas por la picadura de insectos del género Lutzomyia en el nuevo mundo1. Afecta a mamíferos entre los que se incluye el hombre. Clínicamente los cuadros pueden variar desde lesiones cutáneas ulcerosas que cicatrizan; leishmaniasis cutánea (LC), las no ulcerosas o difusas que no curan fácilmente; leishmaniasis cutáneo difusa (LCD), la forma mucosa llamada leishmaniasis mucocutánea (LMC) y la forma sistémica fatal llamada leishmaniasis visceral (LV). Las manifestaciones clínicas dependen tanto del estado inmunológico del hospedador como de la especie causante de la infección, con al menos 20 especies patógenas para el humano2,3. La leishmaniasis es considerada como un importante problema de salud pública, debido al aumento en el número de casos. El riesgo de infección parasitaria se asocia no sólo con factores ambientales tales como migraciones masivas, urbanización, deforestación; sino también se encuentran factores individuales como infección con VIH, desnutrición, genética, entre otros3. Los perros domesticos han sido implicados como reservorios del parásito, pudiendo ser asintomáticos o sufrir la enfermedad, dependiendo de la respuesta inmunológica del animal4.
De esta forma, la leishmaniasis visceral canina (LVC) está ampliamente distribuida en América del Sur y es una de las enfermedades caninas más importantes de la región, principalmente debido a su importancia zoonótica5. Varias especies de parásitos de Leishmania sp. han sido aislados de perros en América del Sur y caracterizados molecularmente, entre estos se incluyen L. braziliensis, L. peruviana, L. colombiensis, L. mexicana, L. panamensis, L. guyanensis, Leishmania amazonensis, L. pifanoi y L. infantum5,6. Diversos métodos de caracterización se han aplicado para el estudio de este género, tales como electroforesis de isoenzimas, análisis del ADN del kinetoplasto (ADNk), la técnica de ADN polimórfico amplificado al azar (RAPD) y la tipificación molecular7-11. En los últimos años, la reacción en cadena de la polimerasa, seguida del análisis de la longitud de los fragmentos de restricción del producto amplificado (PCR-RFLP), se ha erigido en una importante herramienta, no solo para la identificación, sino para la caracterización de especies de este género, en distintas zonas geográficas12,13. Es así que la identificación y caracterización precisa de las especies de Leishmania sp. es importante y necesaria para conocer sus características epidemiológicas, así como para documentar su distribución, lograr nuevos esquemas de tratamiento y finalmente lograr el diseño de medidas de control apropiadas a cada region14,15. Tomando en cuenta los antecedentes mencionados, este trabajo tuvo como objetivo, optimizar las condiciones de la PCR-RFLP/ITS1, utilizando ADN de aislados de parásitos en medio de cultivo bifásico NNN y muestras de biopsias (bazo y ganglio linfático poplíteo) obtenidas de perros con diagnóstico serológico positivo para la leishmaniasis visceral.
MATERIALES Y MÉTODOS
Muestras: Se utilizaron 10 aislados obtenidos previamente en cultivos de promastigotes de la siembra del bazo y 36 muestras biológicas de perros con diagnóstico clínico y serológico positivo para la LV. Fueron seleccionados animales procedentes del Departamento Central, destinados a eutanasia humanitaria a cargo del Programa Nacional de Control de Zoonosis y Centro Antirrábico Nacional (PNCZ y CAN), dependiente del Ministerio de Salud Pública y Bienestar Social, previo consentimiento de los dueños. Las muestras fueron colectadas durante el periodo de marzo a agosto del 2015. A cada animal se le extrajo una porción del bazo, y del ganglio linfático poplíteo. Una porción de cada muestra del bazo fue sometida a cultivo en medio bifásico NNN. Las cepas utilizadas como controles positivos fueron mantenidas en el mismo medio de cultivo durante las pruebas realizadas, para lo cual se utilizaron cepas de referencia internacional de tres especies del género Leishmania, tales como Leishmania (Leishmania) infantum (MHOM/FR/78/LEM75), Leishmania (Viannia) braziliensis (MHOM/BR/75/M2903), Leishmania (Leishmania) amazonensis (MHOM/BR/73/M2269), las cuales fueron proveídas por el Laboratorio de Biología Molecular del Programa de Post-graduación en Ingeniería de Bioprocesos y Biotecnología de la Universidad Federal del Paraná, Brasil.
Cultivo in vitro de Leishmania: Pequeñas porciones del bazo (aproximadamente 2-3 mm2) fueron sometidas a lavado con buffer salino fosfato (PBS 1X), esterilizado, pH 7,2, con el fin de aislar promastigotes en medio de cultivo bifásico NNN suplementado con medio Schneider (Sigma, EEUU), gentamicina, estreptomicina 200 µg/mL y suero fetal bovino al 10% inactivado por calor16. Los cultivos fueron mantenidos en incubadoras a 26 ºC. El crecimiento exponencial de los parásitos fue el parámetro obligado para la extracción de ADN y fueron descartados aquellos medios que demostraron algún tipo de modificación durante el procedimiento. En forma paralela, restos del órgano y ganglio linfático poplíteo fueron conservados a -20ºC hasta su posterior extracción de ADN.
Extracción de ADN: La extracción de ADN de los aislados se realizó por el método convencional (fenol-cloroformo) tomando como referencia la propuesta de Sambrook J, et al17) y de Meredith SEO, et al18. Se procedió a la extracción a partir de la fase líquida del medio de cultivo que contenía las formas promastigotes. Las muestras fueron sometidas previamente a lisis celular y digestión de ARN mediante un buffer de lisis (10 mM de Tris, 10% SDS p/v a concentración final de 0,5% y ARNasa a concentración final de 100 µg/mL) a 37ºC por 30 min., posteriormente la digestión de proteínas se realizó con proteinasa K mediante incubación a 60ºC por toda la noche. La eliminación de proteínas y los procesos de precipitación, lavado y resuspensión, se realizó según el protocolo ya mencionado. Por otra parte, las muestras de biopsias de los órganos mantenidos a -20 ºC, fueron descongeladas y lavadas con buffer salino fosfato (PBS 1X) esterilizado, pH 7,2 y maceradas para la extracción de ADN mediante el Kit comercial Wizard(r) Genomic DNA Purification kit (PROMEGA, EEUU), siguiendo el protocolo recomendado por el fabricante.
Cuantificación de ADN: La concentración y pureza del ADN extraído se estimó midiendo la absorbancia a 260/280 nm. Este procedimiento se realizó en un cuantificador de ADN/ARN/PROTEÍNA (NanoVueTM Plus Spectrophotometer, RU). El grado de pureza se calculó dividiendo la absorbancia a 260 nm entre la absorbancia a 280 nm. Valores óptimos de pureza se encuentran entre 1,7-1,919.
Reacción en cadena de la polimerasa (PCR): Con el fin de obtener fragmentos correspondientes al ADN de la región ITS1 de Leishmania sp., se optimizaron las condiciones de la reacción en un volumen final de 25 μL mediante la variación de la concentración de determinados componentes. Se utilizaron los cebadores: LITSR: 5'-CTGGATCATTTTCCGATG-3' (directo) y L5,8S: 5'-TGATACCACTTATCGCACTT-3' (reverso), ya reportados por Schönian et al (20) y Kuhls et al (21), que amplifican una región de 300 a 350 pb. denominada ITS1 que se encuentra entre los genes SSU rRNA y 5,8S rRNA del género Leishmania. La longitud del amplicón obtenido depende de la especie del género Leishmania que haya sido aislado. La mezcla de reacción para la amplificación contenía Buffer 1X, 1,5 mM de MgCl2, 1µg/µL de BSA (Sigma, EEUU), 0,2 mM de dNTPs (dATP, dTTP, dCTP y dGTP), 0,1 pmol de cada cebador, 1 U de Taq polimerasa, y agua destilada libre de ADNasa/ARNasa UltraPure(tm) (Invitrogen, EEUU). Para la amplificación de muestras de cultivos se requirieron 3 ng/µL de ADN molde. La amplificación a partir de muestras de biopsia solo fue posible a partir 60 ng/µL del ADN molde. En todos los casos, se utilizó un control negativo que contenía todos los componentes de la mezcla de reacción, excepto el ADN molde, el cual fue sustituido por agua libre de ADNasa/ARNasa UltraPureTM (Invitrogen, EEUU).
La amplificación se realizó en un termociclador de ADN (Veriti(r) Thermal Cycler, EEUU), siguiendo las modificaciones propuestas por el Laboratorio de Biología Molecular de la Universidad Federal del Paraná y tomando como referencia la propuesta de Schönian et al20 y Kuhls et al21. Las condiciones de tiempo y temperatura utilizadas fueron desnaturalización inicial a 94ºC por 2 min, seguida de 40 ciclos de procesos de desnaturalización (94ºC por 30 s), hibridación (53ºC por 30 s) y extensión (72ºC por 30 s). Finalmente, un último proceso de extensión se realizo a 72ºC por 10 min.
Análisis de los productos de amplificación: Los amplicones fueron visualizados por electroforesis en gel de agarosa al 1%, en buffer TBE 1X (45 mM Tris - borato, 1 mM EDTA) tanto para la fase móvil como para la fase estacionaria, se utilizaron cubas horizontales, y los geles fueron corridos a 75 Volts durante 40 min. Para la verificación del tamaño de los amplicones, se utilizó un marcador de tamaño molecular en escalera de 1Kb (Invitrogen, EEUU), el gel fue visualizado bajo luz UV, previa tinción con Bromuro de etidio (0,5 µg/mL) por 20 min. y un lavado en agua destilada por 10 min. El resultado final de la electroforesis se fotodocumentó mediante la captura de imágenes en alta resolución bajo luz ultravioleta (UV) utilizando el transiluminador L-PIX TOUCH (LOCCUS Biotecnología, BR).
Digestión enzimática de los amplicones: La restricción de los productos de PCR se realizó con la enzima Hae lll (Invitrogen, EEUU) siguiendo las instrucciones del fabricante. La mezcla contenía agua ultra pura, Buffer M 10X, la enzima de restricción Hae lll (10 U/µL) y finalmente el producto de PCR. La mezcla fue incubada a 37 ºC durante toda la noche. Los perfiles de bandas fueron identificados por electroforesis en gel de agarosa al 3%, utilizando tampón TBE 1X (45 mM Tris - borato, 1 mM EDTA). Para la verificación de la longitud de los productos de la digestión se utilizó un marcador de tamaño molecular en escalera de 50 pb (Invitrogen, EEUU), el gel fue visualizado bajo luz UV, previa tinción con Bromuro de etidio (0,5 µg/mL) por 20 min. Se realizó la captura de imágenes en alta resolución bajo luz ultravioleta (UV) utilizando un transiluminador L-PIX TOUCH (LOCCUS Biotecnología, BR).
El análisis consistió en la comparación de los patrones obtenidos para cada muestra con los controles utilizados. Se analizaron las bandas presentes y ausentes en cada especie, tanto a través de la observación de la corrida electroforética, como de un diagrama en que se reprodujeron las bandas aparecidas en ésta.
Consideraciones éticas: Las muestras de los órganos de perros, tanto ganglio linfático poplíteo y bazo, que se utilizaron en este estudio, fueron obtenidas a partir de animales con serología positiva para la leishmaniasis visceral, luego de la eutanasia humanitaria realizada con el consentimiento previo de sus dueños quienes firmaron un acta para llevar a cabo el procedimiento, teniendo en cuenta las normas previstas por el Programa Nacional de Control de Zoonosis y Centro Antirrábico Nacional del Ministerio de Salud Pública y Bienestar Social. Previamente al sacrifico, el personal del Ministerio considero las precauciones necesarias para evitar sufrimientos innecesarios a los animales, según los principios Internacionales de Guías de la Investigación Biomédica que utiliza animales, elaborados por el consejo de Organizaciones Internacionales de Ciencias Médicas (CIOMS) y el Consejo Internacional de Ciencia y Animales de Laboratorio (ICLAS), además se tuvo en cuenta lo dispuesto en la Ley 4840/2013 sobre protección y bienestar animal dictada por el Poder Legislativo Nacional22-24.
RESULTADOS
ITS1-PCR a partir de ADN de parásitos en medio de cultivo bifásico NNN:
Para la amplificación de la región ITS1 de parásitos mantenidos en medio de cultivo, se detectó el blanco molecular con 5 µL de ADN a concentración de 3 ng/µL, por lo que fue suficiente para observar bandas nítidas. En estas reacciones además fueron testadas concentraciones de 8, 15, 31 y 51 ng/µL del ADN, en las cuales de manera similar se observaron bandas bien diferenciadas con los productos de 300-350 pb en los 10/10 aislados de parásitos que reunían las condiciones del criterio de inclusión del trabajo. La amplificación obtenida se visualiza en la Figura 1.

Figura 1 Electroforesis en gel de agarosa al 1% con los productos obtenidos por la amplificación de la región ITS1 con los cebadores LITSR y L5,8S. MM: Marcador de peso molecular de 1 kb (Invitrogen, EEUU). Carriles 1 al 5, muestras de parásitos aislados en medio de cultivo NNN. CN: Control negativo. CP: Control positivo - Leishmania (L.) infantum (MHOM/FR/78/LEM75).
ITS1-PCR a partir de ADN de parásitos en muestras de biopsias:
Para la amplificación de la región ITS1 de parásitos del ganglio linfático poplíteo, las concentraciones menores a 60 ng/µL no fueron suficientes para detectar el blanco molecular, a pesar de la realización de varias reacciones de amplificación sólo se lograron amplificar 10 de las 18 muestras. La sensibilidad de la técnica para este blanco se determinó en una concentración de 60 ng/µL utilizando 5 µL del ADN en la mezcla final de reactivos de la PCR. Esta concentración permitió la observación de bandas nítidas con los productos de 300-350 pb (Figura 2). Los perfiles de bandas obtenidos con el blanco de ADN (ITS1) de los parásitos encontrados en el bazo, también fueron visualizados de forma nítida con la misma concentración utilizada a partir del ADN de los parásitos en ganglio linfático (60 ng/µL), en la que se logró amplificar 10 de las 18 muestras. Esto se muestra en la Figura 2.

Figura 2 Electroforesis en gel de agarosa al 1% con los productos obtenidos por la amplificación de la región ITS1 con los cebadores LITSR y L5,8S. MM: Marcador de peso molecular de 1 kb (Invitrogen, EEUU). Carriles 1 y 3: muestra de parásitos aislados de biopsias (bazo). Carril 2: muestra de parásitos aislados de biopsias (ganglio linfático poplíteo). CN: Control negativo. CP1: Control positivo Leishmania (L.) infantum (MHOM/FR/78/LEM75). CP2: Control positivo Leishmania (L.) amazonensis (MHOM/BR/73/M2269). CP3: Control positivo Leishmania (V.) braziliensis (MHOM/BR/75/M2903).
Análisis de la longitud de los fragmentos de restricción del producto amplificado (PCR-RFLP)
La digestión enzimática fue desarrollada con 10 µL del producto de la PCR mezclado con 17 µL de agua ultra pura y mezcla de reacción para restricción enzimática, que contenía la enzima y el buffer proveído por el fabricante (Invitrogen, EEUU). La visualización del perfil en geles de agarosa comparado con un marcador de tamaño molecular permitió observar fragmentos resultantes del corte enzimático. Sin embargo, los resultados obtenidos son aún preliminares, considerando que será necesario ajustar la nitidez de las bandas con tamaños menores a 100 pb, ya que a partir del producto amplificado de 300-350 pb se espera que la endonucleasa Hae lll resulte en fragmentos de 184, 72 y 55 pares de bases, correspondiente a la especie Leishmania (L.) infantum. Nuestro resultado permitió la observación del fragmento de mayor tamaño, mientras que los dos fragmentos menores se observaron de manera difusa, sin embargo, se observó que la enzima realiza tales cortes, lo que pudo verificarse con los controles positivos utilizados.
Con el fin de facilitar el análisis del patrón de bandas obtenido, en la fotografía correspondiente al gel de RFLP (Figura 3), se muestra una representación esquemática del mismo en la Figura 4.
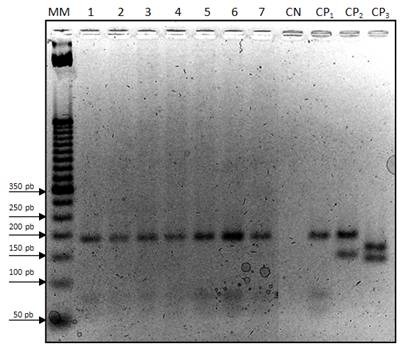
Figura 3 Electroforesis en gel de agarosa al 3%. Patrones de bandas obtenidos, a partir de la digestión del producto de ITS1-PCR con la enzima de restricción Hae lll. MM: Marcador de peso molecular de 50 pb (Invitrogen, EEUU). Carriles 1 al 7: muestra de parásitos aislados en medio de cultivo bifásico NNN. CN: Control negativo. CP1: Control positivo Leishmania (L.) infantum (MHOM/FR/78/LEM75). CP2: Control positivo Leishmania (L.) amazonensis (MHOM/BR/73/M2269). CP3: Control positivo Leishmania (V.) braziliensis (MHOM/BR/75/M2903)
Figura 4 Diagrama en el que se reproducen los fragmentos observados en el gel de agarosa al 3% con los patrones de bandas obtenidos en la figura 3.
Comparación de la amplificación por PCR obtenida a partir del ADN de parásitos aislados de cultivo y muestras de biopsias.
En la tabla se muestra un resumen de las amplificaciones obtenidas para cada grupo de muestras procesadas. Los resultados muestran, como era de esperarse, que la sensibilidad de la PCR es mayor cuando se trata de amplificar el ADN a partir de muestras de cultivo (estadío promastigote), en este caso en el 100% de las muestras se detectó la presencia del género Leishmania sp. con la amplificación del ITS1. Con las muestras obtenidas a partir de muestras de biopsias (bazo y ganglio linfático), la PCR ha demostrado ser menos sensible (Tabla 1).
DISCUSIÓN
Desde el punto de vista epidemiológico, la caracterización de parásitos de Leishmania a nivel de subgénero y especie es importante para establecer la precisa distribución geográfica y el modo de transmisión de éstos patógenos hacia los humanos, así como para establecer el diagnóstico clínico y la evaluación de esquemas terapéuticos adecuados para la enfermedad25. Está demostrado que la leishmaniasis visceral es producida por L. infantum y es endémica en nuestro país, especialmente en Asunción, departamentos Central y Cordillera, en donde se ha observado una elevada seroprevalencia en el reservorio principal, el perro doméstico26. Con la utilización de diversas herramientas moleculares han sido observados altos niveles de variación inter e intra especies tanto en el Viejo como en el Nuevo Mundo. En la actualidad se utilizan técnicas moleculares tales como, la secuenciación, RAPD (Random Amplified Polymorphic DNA), IRT (Intergenic Ribosomal Typing), entre otras27. Diferentes blancos de amplificación han sido utilizados, pero los que presentan mayor sensibilidad son los que se encuentran en secuencias multicopias, como el gen de la subunidad pequeña ribosomal (SSU rRNA), secuencias repetitivas del ADN genómico o de los minicírculos del ADN del kinetoplasto (kDNA), el gen del locus gp63, los cuales han sido utilizados no solo para la detección sino para la identificación de los parásitos27. El uso de la Reacción en Cadena de la Polimerasa, unido a la técnica de Polimorfismo en la longitud de los fragmentos de restricción (PCR-RFLP) también se ha utilizado con el propósito de identificación y caracterización de Leishmania28. El método de amplificación de la región ITS1 seguido del método de RFLP proporciona alta sensibilidad en el diagnóstico e identificación utilizando diferentes tipos de muestras, tal como se ha verificado en este trabajo. Por otra parte, este método puede constituir una herramienta útil para lograr la diferenciación de especies mediante la digestión con endonucleasas de restricción. Así la técnica permite la diferenciación entre las diversas especies29.
En nuestro país se han registrado estudios previos acerca de la caracterización molecular de Leishmania, utilizando marcadores tales como kDNA, SLME, Hsp70, entre otros30-33.
La amplificación de la región ITS1 lograda en nuestro estudio permitió la identificación de bandas nítidas pertenecientes a Leishmania sp., principalmente cuando fueron amplificados los fragmentos de ADN extraídos de muestras derivadas de cultivos, que a pesar de generar una buena cantidad de producto presentó diferencia en comparación a la amplificación obtenida a partir del ADN derivado de las biopsias. Esto concuerda con lo observado por algunos autores que reportaron diferencias en la carga parasitaria y cantidad de ADN, según la (s) especies de Leishmania que estarían parasitando al animal, así como se han observado diferencias entre órganos de un mismo huésped. Esto como consecuencia afecta la sensibilidad de las técnicas moleculares, por lo que pudiese tener menor número de copias de alguna diana para determinadas especies y localizaciones del parásito dentro de su huésped20, lo que nos lleva a pensar que una óptima concentración de reactivos de PCR podría ser determinante para la detección de algunas especies en particular. Nuestros resultados permitieron observar cómo la concentración de 0,2 mM de dNTPs, 1,5 mM de MgCl2 y 0,1 pmol de cada cebador fue óptima para amplificar la región en cuestión.
Por otra parte, los cortes en fragmentos de los productos amplificados que permiten observar diferencias en la longitud de estos fragmentos de restricción y por ende diferencias entre especies a través de la técnica de la PCR-RFLP de la región ITS1, en nuestro estudio sólo se logró detectar parte del fragmento que correspondería a la especie Leishmania infantum, es decir, que la técnica aún no permitió detectar con claridad y nitidez los dos fragmentos de menor tamaño, por lo que será necesario continuar las pruebas relacionadas a la caracterización de especies de Leishmania. Sin embargo, a pesar de lo mencionado, se observó mayor similitud de los cortes procedentes de las muestras caninas, comparadas con los fragmentos de la cepa de referencia internacional MHOM/FR/78/LEM75 perteneciente a Leishmania infantum, utilizada como control positivo.
Esta técnica constituye una herramienta valiosa para el diagnóstico molecular de la leishmaniasis en sus diversas manifestaciones clínicas (LV, LC, LMC)(34) directamente en muestras biológicas o en aislados de medio de cultivo, como así también para el control de calidad (pureza) de cepas de referencia internacional mantenidas en laboratorios. Similarmente, la PCR constituye una herramienta muy útil para el diagnóstico de la leishmaniasis canina y la implicación de los caninos como reservorios en las distintas zonas endémicas para la LV, siendo de gran importancia para la detección de casos asintomáticos. A pesar de esta afirmación, se debe enfatizar que no siempre la técnica de PCR muestra mejores índices diagnósticos que las técnicas convencionales35, por lo que es recomendable su uso en conjunto con otras técnicas para lograr un diagnóstico certero de leishmaniasis y así, basados en esto, fortalecer los programas de control vigente en nuestro país36.
El ensayo de PCR-RFLP es considerado una de las herramientas multipropósito para el diagnóstico directo de muestras clínicas, incluso sin aislamiento previo del parásito; por lo que es recomendado para la caracterización de especies del género Leishmania en zonas endémicas donde circulan varias especies, y por ende exigen una identificación a bajo costo. Este trabajo sugiere continuar con las pruebas realizando algunas modificaciones adecuadas a las condiciones de laboratorio, considerando que, un principio fundamental para el uso de la PCR continúa siendo su optimización, con el fin de garantizar una amplificación exitosa35.
Finalmente, esta técnica estandarizada será muy valiosa en el diseño e implementación de sistemas de control y vigilancia de enfermedades.














