Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Memorias del Instituto de Investigaciones en Ciencias de la Salud
On-line version ISSN 1812-9528
Mem. Inst. Investig. Cienc. Salud vol.11 no.2 Asunción Dec. 2013
TEMAS DE ACTUALIDAD
Cepas de Trypanosoma cruzi en el Paraguay
Trypanosoma cruzi strains in Paraguay
*Acosta N, López E
Departamento de Medicina Tropical, Instituto de Investigaciones en Ciencias de la Salud. Universidad Nacional de Asunción, Paraguay
RESUMEN
Las poblaciones del Trypanosoma cruzi que circulan en Paraguay han mostrado una gran diversidad genética, la cual ha sido evaluada usando diferentes metodologías y técnicas. Se han identificado varios genotipos de este parásito infectando una diversidad de hospederos, incluyendo pacientes, vinchucas, animales silvestres y domésticos de diferentes regiones. La diversidad genética observada en los aislados de nuestro país podría implicar diferencias en las manifestaciones clínicas de la enfermedad de Chagas, así como diferencias en sus características biológicas, como ser el grado de infectividad, virulencia, patogenicidad, capacidad antigénica y susceptibilidad o resistencia a las drogas tripanocidas. Hasta el momento, estos aspectos han sido poco explorados y evaluados en las cepas paraguayas. El aislamiento e identificación del grupo y subgrupos de T. cruzi, que circulan en los diferentes ciclos de transmisión en una determinada región geográfica, siguen siendo relevantes porque permiten conocer la dinámica de transmisión del parásito y evaluar el riesgo de introducción a partir de focos selváticos.
Palabras claves: cepas de Trypanosoma cruzi - Enfermedad de Chagas – Paraguay.
ABSTRACT
Trypanosoma cruzi populations circulating in Paraguay have shown a great genetic diversity, which has been evaluated by different methodologies and techniques. Several genotypes of this parasite have been identified infecting a diversity of hosts, including patients, triatomines and wild and domestic animals from different regions. The genetic diversity observed in the isolates of our country could imply differences in the clinical manifestations of Chagas disease, as well as differences in their biological characteristics such as grade of infectivity, virulence, pathogenicity, antigenic capacity and susceptibility or resistance to trypanocidal drugs. Up to now, these aspects have been little explored and evaluated in Paraguayan strains. The isolation and identification of T. cruzi groups and subgroups that circulate in the different transmission cycles in a determined geographic region are still relevant because they allow knowing the dynamics of parasite transmission and evaluating the introduction risk from wild foci.
Keywords: Trypanosoma cruzi strains – Chagas disease – Paraguay.
INTRODUCCIÓN
El hemoflagelado Trypanosoma cruzi, agente causal de la enfermedad de Chagas, presenta un marcada variabilidad genética así como un comportamiento biológico muy variable. Sumado a esta variabilidad o debido a ella, este parásito tiene la capacidad de infectar a un gran número de especies de mamíferos, alrededor de 200, incluyendo animales domésticos y selváticos, seres humanos y también diferentes especies de insectos triatominos (hematófagos) (1,2). Las manifestaciones clínicas de esta enfermedad pueden estar relacionadas, entre otros factores, a las características de su agente causal. Varios marcadores moleculares han sido utilizados para la genotipificación de este parásito. Actualmente y de acuerdo al último consenso, se lo considera dividido en seis unidades taxonómicas discretas o unidades discretas de tipificación (UDTs) o DTUs por sus siglas en inglés (discrete typing unit) (3,4). El término unidades discretas de tipificación fue propuesto para definir a un conjunto de aislados que son genéticamente más parecidos entre ellos que con otros aislados y que son identificables por marcadores genéticos, moleculares e inmunológicos (5). Las UDTs se denominan respectivamente como TcI, TcII, TcIII, TcIV, TcV y TcVI. Se ha observado que estas UDTs aparentemente se distribuyen diferencialmente entre las diversas especies de triatominos, hospedadores mamíferos y ciclos de transmisión en distintas áreas geográficas de las Américas (4,6,7). A pesar de que todas las cepas de T. cruzi son consideradas infectivas para el ser humano, algunos estudios sugieren que TcII, TcV y TcVI estarían más asociados con ambientes domésticos y con pacientes con manifestaciones crónicas de la enfermedad; T. cruzi TcIII y TcIV con ambientes silvestres y TcI con ambos ambientes (4,6,7). Estudios recientes, basados en secuenciación y microsatélites, indican que el UDT TcI es un grupo muy diverso y que se divide a su vez en 5 subgrupos, denominados TcIa, TcIb, TcIc, TcId y TcIe (4,8,9).
En nuestro país, se han realizado varios trabajos sobre la diversidad genética de las poblaciones de T. cruzi circulantes tanto en el ciclo doméstico como selvático, aunque no muchos referidos al comportamiento biológico de las mismas. La determinación del grupo y subgrupos del T. cruzi que circulan en una determinada región geográfica, en ambos ciclos de transmisión, permite reconocer a los vectores principales y secundarios del mismo y verificar si hay transmisión selectiva del algún grupo en particular entre los diferentes reservorios. Esta información es de importancia para los estamentos encargados de la elaboración de estrategias de control y de vigilancia epidemiológica, porque permite evaluar la dinámica de transmisión del parásito y el riesgo de introducción a partir de focos selváticos.
AISLAMIENTO Y CARACTERIZACIÓN DE CEPAS DE Trypanosoma cruzi
Para el aislamiento de cepas de T. cruzi, se pueden emplear varias técnicas tales como el hemocultivo, xenocultivo, xenodiagnóstico y la inoculación en ratones de laboratorio. En general, los parásitos extraídos del hospedero son colocados en recipientes estériles (tubos o frascos) que contienen medio de cultivo adecuado para el crecimiento y multiplicación de los mismos, tales como el medio LIT (liver infusion tryptose) (10) o también pueden ser medios bifásicos como el 3N (Medio de Novy, McNeal, Nicolle) (11). Este procedimiento puede solapar infecciones mixtas presentes en el individuo infectado, ya que por lo general crecen las sub-poblaciones del parásito mejor adaptadas a los medios de cultivos. Por ello, se recomienda además analizar directamente el material extraído del hospedero (sangre, tejidos, contenido intestinal) con técnicas más sensibles y específicas, como lo son las técnicas moleculares. Las mismas sirven para detectar la presencia del ADN (ácido desoxirribonucleico) o ARN (ácido ribonucleico) del parásito, aún en cantidades pequeñas, aunque tienen la desventaja de que no discriminan si provienen de parásitos vivos o muertos. Por lo tanto, siempre es importante realizar el procedimiento de aislar el parásito, ya que de esta manera se tiene la certeza de su presencia y circulación en el hospedero. Además, así se tienen disponibles poblaciones del parásito para realizar otros estudios complementarios, como comportamiento biológico, patogenicidad, virulencia, susceptibilidad a drogas tripanocidas, capacidad antigénica, entre otros, proporcionando datos que son muy importantes en la caracterización epidemiológica de la enfermedad de Chagas en una determinada región.
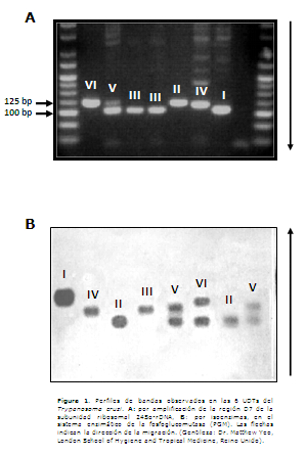
Se han utilizado varias técnicas en la genotipificación del T. cruzi, tales como esquizodema (12), isoenzimas (13,14), amplificación de la región del mini-exón, a-rDNA y 18S rDNA del ADN ribosómico (15-17), RAPD (randomly amplification of polymorphic DNA) (18), PCR-RFLP (Polymerase chain reaction-Restriction fragment length polymorphism) (19,20), hibridización con sondas específicas (21), reacción en cadena de la polimerasa (PCR) en tiempo real (22), inmunogenotipificación utilizando el antígeno TSSA (trypomastigote small-surface antigen) (23,24) y últimamente secuenciación de múltiples genes (25). La Figura 1 muestra el perfil de bandas de las diferentes UDTs del T. cruzi con la técnica de amplificación de región polimórfica D7 de la subunidad ribosomal 24Sα-rDNA y por la técnica de isoenzimas, con el sistema enzimático de la fosfoglucomutasa (PGM). Por lo general, se requiere del uso combinado de varias técnicas en algoritmos o flujogramas para la adecuada discriminación de las seis UDTs de T. cruzi actualmente reconocidas, utilizando las respectivas cepas de referencia (20,26,27). La elección de las técnicas a ser utilizadas en la caracterización de nuevos aislados de T. cruzi es de suma importancia, porque de ello depende el conocimiento real de la distribución de un determinado linaje y de las asociaciones con hospederos y ciclos de transmisión.
DIVERSIDAD GENÉTICA DEL Trypanosoma cruzi EN PARAGUAY
La diversidad genética de las cepas que circulan en Paraguay fue estudiada por diferentes técnicas. Uno de los primeros trabajos fue realizado por Chapman et al., en 1984 (28), en el cual se analizaron por la técnica de isoenzimas, veintidós aislados en total, provenientes de Triatoma infestans domésticos (trece aislados) y de animales domésticos (perros) (nueve aislados) de una localidad del Chaco. Se utilizaron seis sistemas enzimáticos y se detectaron dos genotipos diferentes. Uno de ellos, presente en tres aislados de perros, presentó patrones de bandas homocigotas parecidos a los descritos en el ciclo selvático del Brasil. El otro genotipo, presente en la mayoría de los aislados, mostró perfiles de bandas heterocigotas, diferentes al de las cepas bolivianas que fueron utilizadas como referencia.
En 1990, Yamasaki et al. (29), analizaron tres aislados de pacientes en fase aguda de la enfermedad por la técnica del esquizodema, utilizando una enzima de restricción, la EcoRI. Los perfiles de bandas obtenidos con las cepas paraguayas fueron similares a los observados en cepas brasileras que fueron utilizadas como referencia. Más tarde, en 1992, utilizando la misma técnica, Mimori et al. (30) caracterizaron once aislados de T. cruzi, provenientes de pacientes y T. infestans domésticos de cuatro departamentos (Presidente Hayes, Central, Cordillera y Paraguarí). Entre los aislados de pacientes, se incluyeron cinco en fase aguda de la enfermedad, dos en fase indeterminada y dos con manifestaciones cardíacas crónicas. En este trabajo se utilizaron seis enzimas de restricción (HaeIII, MspI, EcoRI, HimfI, TaqI y RsaI), de las cuales cuatro fueron útiles para la discriminación de subpoblaciones. Cepas de Colombia, Brasil y Chile fueron utilizadas como referencia. De acuerdo a los patrones de bandas observados, los once aislados se dividieron en cuatro grupos. Por comparaciones con trabajos previos se concluyó que dos de estos grupos que incluían a cepas provenientes de Paraguarí, Central, Cordillera y Presidente Hayes eran parecidos a cepas de Brasil y un grupo formado por aislados de Cordillera y Presidente Hayes parecidos a cepas de Bolivia. Todos los grupos fueron muy diferentes a las cepas colombianas.
En 1999, Baba et al. (31) analizaron nueve aislados (siete de pacientes y dos de T. infestans domésticos) por RAPD (randomly amplification of polymorphic DNA) utilizando seis primers, de los cuales cuatro permitieron realizar comparaciones entre cepas. Los aislados provenían de cuatro regiones diferentes (Cordillera, Paraguarí, Canindeyú y Central). Como referencia se utilizaron cepas de Colombia, Brasil y Chile. De acuerdo al perfil de bandas, los aislados fueron divididos en dos grupos, un grupo formado por aislados provenientes de Paraguarí y Central parecidos a cepas brasileras y el otro grupo constituido por aislados provenientes de Paraguarí, Cordillera y Canindeyú parecidos a cepas de Chile. También en este caso, los aislados fueron diferentes a las cepas colombianas.
En el 2001, Acosta et al. (32) analizaron veintiún aislados en total, provenientes de pacientes (doce aislados), T. infestans domésticos (ocho aislados) y de un armadillo, por la técnica de isoenzimas, utilizando trece sistemas enzimáticos (quince loci en total). En este estudio, se incluyó por primera vez el aislado de un animal silvestre. Las cepas provenían de seis regiones diferentes de Paraguay, incluyendo Central, Caaguazú, Cordillera, Paraguarí, San Pedro y Presidente Hayes. De acuerdo al perfil de bandas obtenido, se observó una gran diversidad y se identificaron catorce zimodemas diferentes divididos en tres subgrupos. En el subgrupo 1 se incluyeron aislados provenientes de Central, San Pedro y Paraguarí, con perfiles heterocigotas parecidos a los observados en cepas de Bolivia. En el subgrupo 2 (el más frecuente) se incluyeron los aislados provenientes de Presidente Hayes, San Pedro y Caaguazú, los cuales también presentaron perfiles heterocigotas pero diferentes a los observados en el subgrupo 1 y parecidos a los reportados en cepas de Chile. En el subgrupo 3 se incluyeron aislados provenientes de Paraguarí, Presidente Hayes, San Pedro y Central con perfiles homocigotas, parecidos a cepas brasileras. La región con mayor diversidad fue San Pedro, en la cual se observaron aislados pertenecientes a los 3 subgrupos. Más tarde, Higo et al. en 2004 (33), usando también la técnica de isoenzimas, pero con dieciocho sistemas enzimáticos, analizaron ciento cuarenta y seis aislados de diferentes países, incluyendo nueve de Paraguay. Se observó una gran variabilidad entre los aislados de nuestro país, ya que de acuerdo al análisis de los zimogramas, los mismos pertenecían a cuatro zimodemas diferentes, de los cuales dos mostraron perfiles parecidos a cepas de Bolivia, uno a cepas de Chile y el otro a cepas de Brasil.
En 2005, Yeo et al. (6) analizaron cincuenta y cuatro aislados provenientes de T. infestans domésticos (treinta y un aislados), armadillos (veintidós aislados) y de 1 marsupial, de la zona de San Pedro y Chaco central. Los mismos se analizaron usando una combinación de las técnicas de isoenzimas, amplificación de la región del mini-exón, 24Sa rDNA y 18S rDNA. Se observaron tres genotipos diferentes en los aislados de los armadillos y uno de ellos se observó con mayor frecuencia en diecinueve de los veintidós aislados analizados de estos animales. También en los aislados de las vinchucas se observaron tres genotipos diferentes, con predominio de uno de ellos sobre los demás, presente en trece de los treinta y un aislados.
Del Puerto et al. en 2010 (34) analizaron en forma directa, sin realizar aislamiento de los parásitos, muestras de heces provenientes de T. infestans domésticos y peridomésticos, y muestras sanguíneas de recién nacidos infectados por transmisión congénita de la zona de Cordillera y Paraguarí. Para la genotipificación, las muestras se analizaron por técnicas de amplificación de la región del mini-exón, 24Sα-rDNA y por hibridización con sondas especificas para estos dos genes. En las vinchucas se observaron tres diferentes genotipos, con predominio de uno de ellos, presente en veintisiete de las treinta y tres muestras positivas provenientes de estos insectos. En los niños se observaron dos diferentes genotipos, con predominio de uno de ellos, presente en treinta y seis de los cincuenta y ocho analizados.
En un trabajo reciente, se analizó el contenido intestinal de ejemplares de T. sordida capturados en domicilio y peridomicilio de la zona de Concepción (35). El material extraído de las vinchucas se analizó con técnicas de amplificación de la región del mini-exón y 24Sa-rDNA y por hibridización con sondas específicas para el gen del mini-exón. Se identificaron cinco diferentes genotipos, siendo uno de ellos el más frecuentemente observado en once de las veinte muestras que resultaron positivas.
Adicionalmente a estos estudios, varias cepas de T. cruzi aisladas en nuestro país ya se han caracterizado por secuenciación, incluyendo diferentes genes nucleares y mitocondriales (9,25,36,37) y por microsatélites (37,38).
UNIDADES TAXONÓMICAS DISCRETAS (UDTs) DEL Trypanosoma cruzi CIRCULANTES EN PARAGUAY
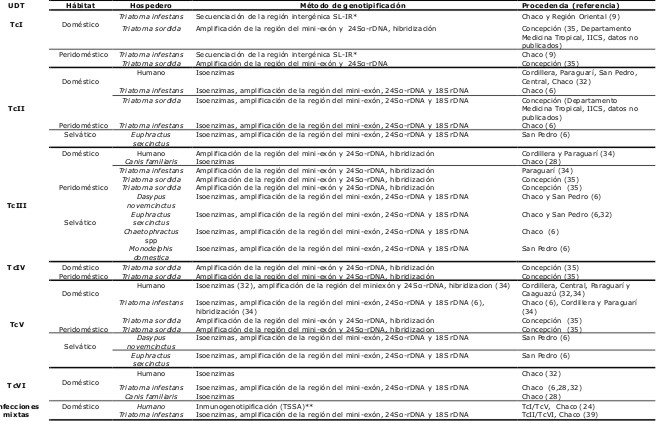
En nuestro país, utilizando diferentes técnicas se ha reportado la circulación de las seis unidades taxonómicas discretas (UDTs) de T. cruzi, aunque varían en frecuencia, distribución geográfica, tipo de hospederos y en los ciclos de transmisión (Tabla 1).
El UDT TcI junto con TcIV son los grupos que se han reportado con menor frecuencia. El TcI se identificó en el suero de un paciente de la zona chaqueña en infección mixta con TcV (24), en especímenes domésticos y peridomésticos de T. sordida de la zona de Concepción (35) y de acuerdo a registros del Departamento Medicina Tropical, IICS (datos no publicados) y en ejemplares domésticos y peridomésticos de T. infestans de la región Oriental y del Chaco (9). En las muestras provenientes de T. infestans se identificaron los subgrupos TcId (el más frecuente) y en una vinchuca mezcla de TcIa y TcId (9).
El UDT TcII se identificó en casos de pacientes provenientes de Cordillera, Paraguarí, San Pedro, Central y Chaco (32), en ejemplares domésticos y peridomésticos del T. infestans del Chaco (6), en ejemplares domésticos de T. sordida de Concepción (Departamento Medicina Tropical, IICS, datos no publicados) y en un armadillo (selvático) de la zona de San Pedro (6).
El UDT TcIII se ha reportado con mayor frecuencia en el ciclo selvático, especialmente en armadillos de la zona del Chaco y San Pedro (6). También se lo ha identificado en un marsupial de la zona de San Pedro (6), en muestras sanguíneas de niños con transmisión congénita de la zona de Cordillera y Paraguarí (34), en un ejemplar doméstico de T. infestans de Paraguarí (34), en ejemplares domésticos y peridomésticos de T. sordida de la zona de Concepción (35) y en tres aislados de perros de una localidad de la región del Chaco (28).
El UDT TcIV se ha identificado en ejemplares domésticos y peridomésticos de T. sordida en Concepción (35).
El UDT TcV se ha reportado con mayor frecuencia en el ciclo doméstico, tanto en la región Oriental como en el Chaco. Así, se lo ha identificado en aislados y muestras sanguíneas de pacientes de Cordillera, Paraguarí, Central y Caaguazú (32,34), en ejemplares domésticos de T. infestans del Chaco (6) y de Cordillera y Paraguarí (34), en ejemplares domésticos y peridomésticos de T. sordida en Concepción (35), y en dos armadillos (selváticos) de San Pedro (6).
En nuestro país, el UDT TcVI hasta la fecha solo se ha reportado en el ciclo doméstico del Chaco, en tres casos de pacientes (32), en ejemplares domésticos y peridomésticos de T. infestans (6,28,32) y en tres aislados de perros (28). Así mismo, se identificó en aislados de T. infestans en infecciones mixtas con el UDT TcII (6,39).
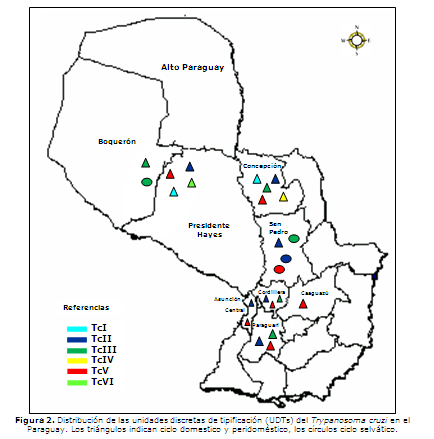
De acuerdo a estos datos, las regiones en las cuales se observó mayor diversidad en las poblaciones del T. cruzi fueron el Chaco y el departamento de Concepción (Figura 2). En el Chaco se han identificado cinco UDTs circulando en diferentes ciclos de transmisión y hospederos. En el ciclo doméstico se ha encontrado al TcI (en T. infestans y un paciente), TcII (en T. infestans), TcIII (en perros), TcV (en T. infestans) y TcVI (en pacientes, perros y T. infestans). En el ciclo selvático se ha observado el TcIII (en armadillos). En ejemplares de T. sordida de Concepción se observaron cinco UDTs diferentes (TcI, TcII, TcIII, TcIV y TcV). En el departamento de San Pedro, se observó la mayor variedad de cepas circulando en el ciclo selvático. Se identificaron tres UDTs: TcII (en un armadillo), TcIII, el más frecuente (en armadillos y un marsupial) y TcV (en dos armadillos). En el ciclo doméstico sólo se observó TcII (en pacientes). En los departamentos de Cordillera y Paraguarí, hasta la fecha sólo se tienen muestras pertenecientes al ciclo doméstico, las cuales correspondieron a tres UDTs diferentes (TcII, TcIII y TcV). En Central, se identificaron dos UDTs (TcII y TcV) en el ciclo doméstico.
CARACTERIZACIÓN BIOLÓGICA DE CEPAS PARAGUAYAS DE Trypanosoma cruzi
Aunque se han realizado numerosos trabajos referentes a la diversidad genética de las cepas de T. cruzi que circulan en nuestro país, no se tiene mucha información sobre sus características biológicas. En un trabajo realizado por Acosta et al. en 1995 (40), se estudió el comportamiento biológico en el modelo murino (ratones BALB/c) de cinco cepas aisladas de pacientes en fase aguda de la enfermedad de Chagas, provenientes de cuatro departamentos (Cordillera, Central, Presidente Hayes y Paraguarí). Las cepas se caracterizaron basándose en la evolución de la parasitemia (curva), virulencia y tasa de mortalidad. Además, se determinó la capacidad de metaciclogénesis, biometría y aglutinación con lectinas. Las cepas fueron muy variables con respecto a los parámetros estudiados. En una de las cepas se observó un pico de parasitemia temprano (al 4to día post-infección) con bajas parasitemias (promedio: 2.5 x 104 p/ml), mientras que dos cepas tuvieron picos de parasitemia más tardíos (entre los 11 y 14 días post-infección) con parasitemias más altas (entre 7 x 104 y 2 x 105 p/ml). En las restantes dos cepas, sin embargo, no se pudo determinar la curva parasitémica por los bajos niveles de parásitos observados en sangre, que oscilaban con períodos de parasitemia sub-patentes (no detectables). Se observó el 100% de sobrevida de los ratones a los 90 días post infección. También se observaron diferencias entre las cepas estudiadas en su capacidad de metaciclogénesis, que varió entre el 36% y 50%, así como en la longitud de los parásitos, la cual osciló entre 22.8 µm y 20.6 µm en las formas metacíclicas. Las cepas paraguayas presentaron diferencias en el comportamiento biológico en ratones con la cepa brasilera Y, que se caracteriza por tener picos de parasitemia entre los 7 y 10 días, con muy elevada parasitemia (1 x 107 p/ml) y alta mortalidad (95-100%) entre los 12 y 15 días post-infección (41).
La variabilidad genética de este parásito es uno de los factores determinantes de la patogénesis de la enfermedad de Chagas en su fase crónica, ya que se ha observado tropismo tisular preferencial en algunos clones de este parásito (27,42). En nuestro país la principal manifestación clínica observada en los pacientes chagásicos es la cardiopatía, seguida por el megacolon y el megaesófago (43). Aún no se han realizado estudios experimentales con cepas paraguayas para determinar el tropismo tisular de las mismas y si existen diferencias o no entre los aislados de pacientes, animales silvestres y vinchucas de diferentes regiones.
La efectividad de los tratamientos con drogas tripanocidas también puede verse influenciada por la variabilidad genética del parásito. El benznidazol es la droga de elección para el tratamiento de los pacientes chagásicos en nuestro país. En un trabajo reciente, se evaluó la susceptibilidad in vitro de cepas paraguayas a esta droga (Hamuy R, manuscrito en preparación). Sin embargo, este es un tema aún poco estudiado en cepas paraguayas. Se han reportado cepas con resistencia natural al benznidazol en Bolivia (44), Brasil (45) y Colombia (46).
COLECCIÓN DE CEPAS DE Trypanosoma cruzi DEL DEPARTAMENTO DE MEDICINA TROPICAL (IICS-UNA).
El departamento de Medicina Tropical del Instituto de Investigaciones en Ciencias de la Salud (IICS-UNA) cuenta con una colección de cepas paraguayas del Trypanosoma cruzi. La misma se fue conformando como producto de diferentes proyectos de investigación y cooperaciones con otras instituciones. En la actualidad, cuenta con 249 cepas, 32 aisladas de pacientes, 40 de reservorios mamíferos silvestres y 177 de vinchucas, de diferentes regiones del país. A esto se suman 73 cepas de referencia, provenientes de casos humanos, mamíferos silvestres y vinchucas de diferentes países.
CONCLUSIONES
Las poblaciones del T. cruzi que circulan en el Paraguay presentan una gran diversidad genética. Las seis unidades discretas de tipificación, que corresponden a la actual clasificación de este parásito, se han identificado en nuestro país utilizando diferentes técnicas. Las mismas fueron encontradas circulando en diferentes hospederos, tales como insectos triatominos, incluyendo al T. infestans y T. sordida, en pacientes provenientes de diferentes departamentos y en distintas fases de la enfermedad, en animales silvestres, tales como armadillos y marsupiales, y en animales domésticos (perros). Las regiones en las cuales se observó mayor diversidad de genotipos fueron el Chaco y el departamento de Concepción. En el ciclo doméstico se han identificado los seis UDTs, aunque la distribución varía según la zona geográfica, con predominio de los grupos TcII, TcV y TcVI. En el ciclo selvático, se han identificado hasta la fecha tres genotipos, TcII, TcIII y TcV, todos aislados de animales silvestres, con predominio del TcIII.
Aunque se han realizado numerosos trabajos referentes a la diversidad genética de las cepas de T. cruzi que circulan en nuestro país, poco se sabe acerca de sus características biológicas. Por lo tanto, se necesitan más trabajos para establecer el grado de infectividad, virulencia, patogenicidad y capacidad antigénica de las cepas y evaluar si existen diferencias entre los aislados de pacientes, animales silvestres y vinchucas de diferentes regiones.
También son necesarios estudios in vitro e in vivo relacionados a la susceptibilidad a drogas tripanocidas tradicionales y las que se encuentran en fase de experimentación, ya que en otros países se ha reportado la existencia de cepas naturalmente resistentes.
Todos estos aspectos relacionados al agente causal son relevantes para la caracterización epidemiológica de la enfermedad de Chagas en nuestro país.
REFERENCIAS BIBLIOGRÁFICAS
1. Dias JCP.- Epidemiologia. En: Brener Z, Andrade ZA, ed. Trypanosoma cruzi e doença de Chagas. Rio de Janeiro: Guanabara Koogan; 2000. p. 48-54. [ Links ]
2. Barretto MP. - Epidemiologia. En: Brener Z, Andrade ZA, ed. Trypanosoma cruzi e doença de Chagas. Rio de Janeiro: Guanabara Koogan; 1979. p. 89-151. [ Links ]
3. Zingales B, Andrade SG, Briones MR, Campbell DA, Chiari E, Fernandes O et al. Second Satellite Meeting. A new consensus for Trypanosoma cruzi intraspecific nomenclature: second revision meeting recommends TcI to TcVI. Mem Inst Oswaldo Cruz. 2009 Nov; 104(7):1051-4. [ Links ]
4. Zingales B, Miles MA, Campbell DA, Tibayrenc M, Macedo AM, Teixeira MM et al. The revised Trypanosoma cruzi subspecific nomenclature: rationale, epidemiological relevance and research applications. Infect Genet Evol. 2012 Mar; 12(2):240-53. [ Links ]
5. Tibayrenc, M. Genetic epidemiology of parasitic protozoa and other infectious agents: the need for an integrated approach. Int J Parasitol. 1998; 28(1):85-104. [ Links ]
6. Yeo M, Acosta N, Llewellyn M, Sanchez H, Adamson S, Miles GA et al. Origins of Chagas disease: Didelphis species are natural hosts of Trypanosoma cruzi I and armadillos hosts of Trypanosoma cruzi II, including hybrids. Int J Parasitol. 2005; 35(2):225-33. [ Links ]
7. Miles MA, Llewellyn MS, Lewis MD, Yeo M, Baleela R, Fitzpatrick S et al. The molecular epidemiology and phylogeography of Trypanosoma cruzi and parallel research on Leishmania: looking back and to the future. Parasitology. 2009 Oct; 136(12):1509-28. [ Links ]
8. Herrera C, Bargues MD, Fajardo A, Montilla M, Triana O, Vallejo GA et al. Identifying four Trypanosoma cruzi I isolate haplotypes from different geographic regions in Colombia. Infect Genet Evol. 2007 Jul; 7(4):535-9. [ Links ]
9. Cura CI, Mejía-Jaramillo AM, Duffy T, Burgos JM, Rodriguero M, Cardinal MV et al. Trypanosoma cruzi I genotypes in different geographical regions and transmission cycles based on a microsatellite motif of the intergenic spacer of spliced-leader genes. Int J Parasitol. 2010 Dec; 40(14):1599-607. [ Links ]
10. Camargo E. Growth and differentiation in Trypanosoma cruzi I. Origen of metacyclic trypanosomes in liquid media. Rev Inst Med Trop São Paulo. 1964;6:93-100. [ Links ]
11. Walton BC, Shaw JJ, Lainson R. Observations on the "in vitro" cultivation of Leishmania braziliensis. J Parasitol 1977; 63:1118-9. [ Links ]
12. Morel C, Chiari E, Camargo EP, Mattei DM, Romanha AJ, Simpson L. Strains and clones of Trypanosoma cruzi can be characterized by pattern of restriction endonuclease products of kinetoplast DNA minicircles. Proc Natl Acad Sci USA. 1980; 77:6810-4. [ Links ]
13. Miles MA, Souza A, Povoa M, Shaw JJ, Lainson R, Toye PJ. Isozymic heterogeneity of Trypanosoma cruzi in the first autochthonous patients with Chagas disease in Amazonian Brazil. Nature. 1978; 272:819-821. [ Links ]
14. Tibayrenc M, Ayala FJ. Isoenzyme variability of Trypanosoma cruzi the agent of Chagas disease: genetical, taxonomical and epidemiological significance. Evolution. 1988; 42:277-292. [ Links ]
15. Souto RP, Fernandes O, Macedo AM, Campbell DA, Zingales B. DNA markers define two major phylogenetic lineages of Trypanosoma cruzi. Mol Biochem Parasitol. 1996; 83:141-52. [ Links ]
16. Fernandes O, Souto R, Castro J, Pereira J, Fernandes N, Junqueira A et al. Brazilian isolates of Trypanosoma cruzi from humans and triatomines classified into two lineages using mini-exon and ribosomal RNA sequences. Am J Trop Med Hyg. 1998; 58:807-811. [ Links ]
17. Brisse S, Verhoef J, Tibayrenc M. Characterisation of large and small subunit rRNA and mini-exon genes further supports the distinction of six Trypanosoma cruzi lineages. Int J Parasitol. 2001; 31:1218-26. [ Links ]
18. Martinez-Diaz RA, Escario JA, Nogal-Ruiz JJ, Gomez-Barrio A. Relationship between biological behaviour and randomly amplified polymorphic DNA profiles of Trypanosoma cruzi strains. Mem Inst Oswaldo Cruz. 2001; 96(2):251-6. [ Links ]
19. Rozas M, Doncker SD, Adaui V, Coronado X, Barnabe C, Tibyarenc M et al. Multilocus Polymerase Chain Reaction Restriction Fragment-Length Polymorphism Genotyping of Trypanosoma cruzi (Chagas Disease): Taxonomic and Clinical Applications. J Infec Dis. 2007; 195:1381-1388. [ Links ]
20. Lewis MD, Ma J, Yeo M, Carrasco HJ, Llewellyn MS, Miles MA. Genotyping of Trypanosoma cruzi: systematic selection of assays allowing rapid and accurate discrimination of all known lineages. Am J Trop Med Hyg. 2009; 81:1041-1049. [ Links ]
21. Veas F, Brenière SF, Cuny G, Brengues C, Solari A, Tibayrenc M. General procedure to construct highly specific kDNA probes for clones of Trypanosoma cruzi for sensitive detection by polymerase chain reaction. Cell Mol Biol. 1991; 37:73–84. [ Links ]
22. Duffy T, Bisio M, Altcheh J, Burgos JM, Diez M, Levin MJ et al. Accurate real-time PCR strategy for monitoring bloodstream parasitic loads in Chagas disease patients. PLoS Negl Trop Dis. 2009; 3(4): e419. [ Links ]
23. Bhattacharyya T, Brooks J, Yeo M, Carrasco HJ, Lewis MD, Llewellyn MS et al. Analysis of molecular diversity of the Trypanosoma cruzi trypomastigote small surface antigen reveals novel epitopes, evidence of positive selection and potential implications for lineage-specific serology. Int J Parasitol. 2010 Jul; 40(8):921-8. [ Links ]
24. Risso MG, Sartor PA, Burgos JM, Briceño L, Rodríguez EM, Guhl F et al. Immunological identification of Trypanosoma cruzi lineages in human infection along the endemic area. Am J Trop Med Hyg. 2011 Jan; 84(1):78-84. [ Links ]
25. Yeo M, Mauricio IL, Messenger LA, Lewis MD, Llewellyn MS, Acosta N et al. Multilocus sequence typing (MLST) for lineage assignment and high resolution diversity studies in Trypanosoma cruzi. PLoS Negl Trop Dis. 2011; 5(6):e1049. [ Links ]
26. DAvila DA, Macedo AM, Valadares HM, Gontijo ED, de Castro AM, Machado CR et al. Probing population dynamics of Trypanosoma cruzi during progression of the chronic phase in chagasic patients. J Clin Microbiol. 2009; 47:1718–25. [ Links ]
27. Burgos JM, Diez M, Vigliano C, Bisio M, Risso M, Duffy T, et al. Molecular identification of Trypanosoma cruzi discrete typing units in end-stage chronic Chagas heart disease and reactivation after heart transplantation. Clin Infect Dis. 2010 Set; 51(5):485-95. [ Links ]
28. Chapman MD, Baggaley RC, Godfrey-Fausset PF, Malpas TJ, White G, Canese J, Miles MA. Trypanosoma cruzi from the Paraguayan Chaco: isoenzyme profiles of strains isolated at Makthlawaiya. J Protozool. 1984; 31:482-6. [ Links ]
29. Yamasaki H, Kita K, Oya H, Pavon B, Arias O, Moran M, Vera M. Shizodeme characterization of kinetoplast DNA minicircles of Trypanosoma cruzi and Leishmania isolates from Paraguayan Chagas; disease and leishmaniasis patients. Japan J Trop Med Hyg. 1990; 18:62-63. [ Links ]
30. Mimori T, Maldonado M, Samudio M, Rojas de Arias A, Moreno R, Sakamoto M. Characterization of Trypanosoma cruzi isolates from Paraguay, using restriction enzyme analysis of kinetoplast DNA. Ann Trop Med Parasitol. 1992;86:231-237. [ Links ]
31. Baba S, Matsumoto T, Kanbara H, Sakamoto M, Maldonado M, Rojas de Arias A et al. Comparative study of random amplified polymorphic DNA of Trypanosoma cruzi isolates from Paraguay. Japan J Trop Med Hyg. 1999; 18:62-63. [ Links ]
32. Acosta N, Samudio M, López E, Vargas F, Yaksic N, Brenière SF, Rojas de Arias A. Isoenzyme profiles of Trypanosoma cruzi stocks from different areas of Paraguay. Mem Inst Oswaldo Cruz. 2001 May; 96(4):527-33. [ Links ]
33. Higo H, Miura S, Horio M, Mimori T, Hamano S, Agatsuma T et al. Genotypic variation among lineages of Trypanosoma cruzi and its geographic aspects. Parasitol Int. 2004 Dec; 53(4):337-44. [ Links ]
34. del Puerto F, Sánchez Z, Nara E, Meza G, Paredes B, Ferreira E et al. Trypanosoma cruzi lineages detected in congenitally infected infants and Triatoma infestans from the same disease-endemic region under entomologic surveillance in Paraguay. Am J Trop Med Hyg. 2010 Mar; 82(3):386-90. [ Links ]
35. Sánchez Z. Rol del Triatoma sórdida en la transmisión de la Enfermedad de Chagas en el Paraguay. (Tesis de Maestría). Instituto de Investigaciones en Ciencias de la Salud, Universidad Nacional de Asunción. 2011. [ Links ]
36. Machado CA, Ayala FJ. Nucleotide sequences provide evidence of genetic exchange among distantly related lineages of Trypanosoma cruzi. Proc Natl Acad Sci USA. 2001; 98:7396-401. [ Links ]
37. Lewis MD, Llewellyn MS, Yeo M, Acosta N, Gaunt MW, Miles MA. Recent, independent and anthropogenic origins of Trypanosoma cruzi hybrids. PLoS Negl Trop Dis. 2011 Oct; 5(10):e1363. [ Links ]
38. Llewellyn MS, Lewis MD, Acosta N, Yeo M, Carrasco HJ, Segovia M et al. Trypanosoma cruzi IIc: phylogenetic and phylogeographic insights from sequence and microsatellite analysis and potential impact on emergent Chagas disease. PLoS Negl Trop Dis. 2009 Sep; 3(9):e510. [ Links ]
39. Yeo M, Lewis MD, Carrasco HJ, Acosta N, Llewellyn M, da Silva Valente SA et al. Resolution of multiclonal infections of Trypanosoma cruzi from naturally infected triatomine bugs and from experimentally infected mice by direct plating on a sensitive solid medium. Int J Parasitol. 2007 Jan; 37(1):111-20. [ Links ]
40. Acosta N, Maldonado M, Sanabria L, Yaluff G, Fuentes S, Torres S et al. Characterization of Paraguayan Trypanosoma cruzi strains isolated from acute patients of Chagas disease. Trop Med Parasitol. 1995 Sep; 46(3):195-200. [ Links ]
41. Magalhães JB, Pontes AL, Andrade SG. Behavior of the Y and Peruvian strains of Trypanosoma cruzi in mice, after passage through various media. Mem Inst Oswaldo Cruz. 1985 Jan-Mar; 80(1):41-50. [ Links ]
42. Andrade SG, Magalhães JB. Biodemes and zymodemes of Trypanosoma cruzi strains: correlations with clinical data and experimental pathology. Rev Soc Bras Med Trop. 1996 Jan-Feb; 30(1):27-35. [ Links ]
43. Rojas De Arias A. Chagas disease in Paraguay. PAHO/HCP/HCT/72/96, 1996. [ Links ]
44. Andrade SG, Magalhães JB, Pontes AL. Evaluation of chemotherapy with benznidazole and nifurtimox in mice infected with Trypanosoma cruzi strains of different types. Bull World Health Org. 1985; 63(4):721-6. [ Links ]
45. Gomes ML, Toledo MJ, Nakamura CV, Bittencourt Nde L, Chiari E, de Araújo SM. Trypanosoma cruzi: genetic group with peculiar biochemical and biological behavior. Mem Inst Oswaldo Cruz. 2003 Jul; 98(5):649-54. [ Links ]
46. Mejia-Jaramillo AM, Fernández GJ, Montilla M, Nicholls RS, Triana-Chávez O. Estudio de la susceptibilidad al benznidazol de cepas de Trypanosoma cruzi sugiere la circulación de cepas naturalmente resistentes en Colombia. Biomédica. 2012; 32(2):196-205. [ Links ]
*Autor Correspondiente:Dra. Nidia Acosta. Departamento de Medicina Tropical, Instituto de Investigaciones en Ciencias de la Salud. Universidad Nacional de Asunción.
Email: nidacostag@gmail.com
Fecha de recepción: febrero 2013; Fecha de aceptación: agosto de 2013