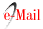Serviços Personalizados
Journal
Artigo
Indicadores
-
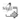 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Memorias del Instituto de Investigaciones en Ciencias de la Salud
versão On-line ISSN 1812-9528
Mem. Inst. Investig. Cienc. Salud v.10 n.1 Asunción jun. 2012
ARTICULO ORIGINAL
Caracterización de cepas de Leishmania, por medio de la técnica de PCR-RFLP de la región del Spliced Leader Miniexon (SLME), aisladas de humanos y caninos en Paraguay
Characterization of Leishmania strains using PCR-RFLP of Spliced Leader Miniexon(SLME) in human and canine isolates in Paraguay
Chena LI,II, *Nara EI, Canese AIII, Oddone RIII,IV, Morán MII, Russomando GI
IDpto de Biología Molecular y Genética. Instituto de Investigaciones en Ciencias de la Salud. UNA. Paraguay
IIDepartamento de Parasitología. Laboratorio Central de Salud Pública, MSP y BS. Paraguay
IIIPrograma Nacional de Control de Leishmaniosis. SENEPA. MSP y BS. Paraguay
IVDpto de Producción Bioquímica. Instituto de Investigaciones en Ciencias de la Salud. UNA. Paraguay
RESUMEN
La leishmaniosis es una enfermedad parasitaria con varias formas clínicas, desde lesiones cutáneas leves hasta enfermedades fatales con comprometimiento visceral. Existen varias técnicas moleculares como por ejemplo la reacción en cadena de la polimerasa (PCR) que amplifica diferentes secuencias blanco, una de ellas es la región SLME (spliced leader miniexon), el producto se corta con enzimas de restricción (PCR-RFLP), permitiendo la identificación de las especies de Leishmania. Este trabajo fue realizado con el objetivo de caracterizar las cepas de Leishmania, empleando una PCR-RFLP de la región SLME, aisladas de humanos y caninos, provenientes de distintas zonas del país. Se analizaron 12 aislados de humanos y 40 de caninos debidamente codificados. Se empleó un par de cebadores para la región SLME, los productos amplificados fueron cortados con las enzimas Hae III y Nco I (RFLP) y los patrones de bandas analizados. Se detectó en un primer paso la presencia de parásitos del subgénero Viannia en 7 aislados de humanos y correspondientes al subgénero Leishmania en 5 aislados de humanos y 40 de caninos. La RFLP según los patrones de bandas permitió identificar a L. braziliensis en aislados de leishmaniosis tegumentaria y L. chagasi en los casos de leishmaniosis visceral. Es importante resaltar que este trabajo es el primero en el país en realizar la caracterización molecular a nivel de especies de Leishmania y los hallazgos descritos tienen una implicación a nivel epidemiológico, contribuyendo con las estrategias de vigilancia y control de la enfermedad. Adicionalmente, se sugiere el uso de otros marcadores genéticos para identificar genotipos o perfiles genéticos diferentes, entre cepas de estas especies.
Palabras claves: Leishmania, genotipificación, PCR-RFLP, miniexón.
ABSTRACT
Leishmaniasis is a parasitic disease with various clinical forms from minor skin lesions to fatal diseases with visceral compromise. There are several molecular techniques such as the polymerase chain reaction (PCR) that amplifies specific target sequences like the SLME(spliced leader miniexon) region. Subsequently, the PCR products are digested with specific restriction enzymes (PCR-RFLP) allowing the identification of Leishmania species. This study was carried out in order to characterize Leishmania strains by using a PCR-RFLP of the SLME region in human and canine isolates from different regions of the country. We analyzed 12 human isolates and 40 dog isolates properly codified. Two primers for SLME region were used and the amplified products were digested with the Hae III and Nco I (RFLP) enzymes, then the banding patterns were analyzed. In 7 human isolates, parasites from the Viannia subgenus were detected while in 5 human isolates and 40 dog isolates, parasites from the Leishmania subgenus were identified. RFLP, according to the restriction banding patterns, allowed the identification of L. braziliensis species in the tegumentary leishmaniasis isolates and L. chagasi in visceral leishmaniasis isolates. This is the first study conducting molecular characterization of Leishmania species in the country, and its findings have implications at the epidemiological level by contributing to the improvement of surveillance strategies for the disease control. Additionally, the use of other genetic markers is suggested in order to identify genotypes or different genetic profiles among the strains of these species.
Keywords: Leishmania, genotyping, PCR-RFLP, miniexon.
INTRODUCCIÓN
La leishmaniosis es una enfermedad parasitaria endémica en muchas áreas tropicales y subtropicales de América, constituyendo un importante problema de salud pública (1), es considerada por la Organización Mundial de la Salud como una de las cinco enfermedades infecto parasitarias de mayor relevancia (2). Es causada por protozoarios del género Leishmania, que son trasmitidos por flebótomos entre animales y el hombre que al interactuar en el hábitat puede adquirir la enfermedad (3,4).
Se encuentra asociada con un amplio espectro de manifestaciones clínicas que pueden variar desde lesiones cutáneas ulcerosas que cicatrizan, la leishmaniosis cutánea (LC); las no ulcerosas o difusas que no curan fácilmente, la leishmaniosis cutáneo difusa (LCD); hasta notorias desfiguraciones en la forma mucosa, leishmaniosis mucocutánea (LMC) y una forma sistémica fatal, la leishmaniosis visceral (LV) (1,5,6). Estas manifestaciones clínicas están sujetas no solamente al estado inmunológico del paciente sino también a especies particulares del parásito, presencia de ciertas especies de flebótomos y reservorios involucrados (4,5,7).
Debido a la amplia variedad de especies de Leishmania y formas clínicas de la enfermedad, un correcto diagnóstico con la identificación de la especie de Leishmania involucrada es importante para realizar un pronóstico de la situación, el manejo más adecuado del paciente o de la población afectada que además permite posteriormente hacer un análisis epidemiológico de la distribución geográfica del parásito (6).
En el Paraguay, la zona endémica, para la forma tegumentaria abarca principalmente los departamentos de San Pedro, Caaguazú, Canindeyú y Alto Paraná, que juntos poseen casi el 80% de los casos totales del país (8) y para la forma visceral el 75% de los casos registrados en los últimos años pertenecían al departamento Central y el 25% restante distribuidos en Asunción, Cordillera, Paraguarí, Alto Paraná, Guaira e Itapúa (9).
Con el empleo de técnicas moleculares como la reacción en cadena de la polimerasa (PCR por sus siglas en inglés: polymerase chain reaction) se ofrece una alternativa de buena sensibilidad y especificidad que permite identificar el género y complejos de Leishmania (7). Últimamente se han desarrollado varios ensayos basados en PCR, que utilizan diferentes blancos de amplificación, como el gen cromosómico que codifica la unidad pequeña ribosomal (SSU por sus siglas en inglés small subunit) rARN (10), secuencias repetitivas (11), espaciadores internos transcritos (ITS, por sus siglas en inglés internal transcribed spacers), ubicado entre los genes genómicos ribosomales que codifican las unidades pequeña y grande (12), el gen del locus de la glicoproteína de superficie gp63 (13), al igual que las secuencias repetitivas de los minicírculos del ADN del kinetoplasto (kADN) (14).
También se ha diseñado una técnica de genotipificación que consiste en la amplificación mediante una PCR de una región del spliced leader miniexon (SLME) que se presenta como repeticiones en tandem (100 a 200 copias) en el género Leishmania y otros protozoarios del orden Kinetoplastida. Esta técnica de PCR que utiliza un único par de cebadores combinada con una restricción enzimática (PCR-RFLP, por sus siglas en inglés: polimerase chain reaction – restricted fragments length polimorfism) permite analizar los patrones que son característicos para cada especie (6).
La caracterización molecular de aislados de Leishmania, no solamente ofrece un apoyo a las técnicas convencionales de diagnóstico, confirmando la presencia del agente causal de la enfermedad, sino que también permite realizar estudios epidemiológicos que brinden un mejor conocimiento de la variabilidad genética del parásito, de su distribución geográfica y de su relación con la patología que desarrolla.
Este trabajo tiene como objetivo la caracterización de cepas de Leishmania aisladas de pacientes humanos y caninos, empleando la técnica PCR-RFLP de la región SLME, provenientes de distintas zonas del país.
MATERIALES Y MÉTODOS
1. Cultivo in vitro de Leishmania: Las formas promastigotes de cultivo de Leishmania fueron aisladas en la fase liquida del medio de cultivo NNN, a 26º C. Una vez que se obtuvo el crecimiento de las formas promastigotes, se transfirió una alícuota a un medio bifásico NNN, cuyo sobrenadante era RPMI 1640 suplementado con suero bovino fetal al 20%. Finalmente, para una mayor multiplicación de los parásitos, estos se cultivaron en RPMI 1640 suplementado con suero bovino fetal al 20%. A los 7 días de incubación en RPMI 1640, los parásitos se cosecharon y separaron por centrifugación a 2.000 rpm lavándolos 4 veces con buffer salino-fosfato pH= 7,2 (PBS).
2. Muestras: Fueron utilizadas cepas de referencia de tres especies de Leishmania: Leishmania (V.) braziliensis (MHOM/BR/75/M2903), Leishmania chagasi (MHOM/BR/1974/PP75) y Leishmania (L.) amazonensis (IFLA/BR/1967/PH8) (provistas por el Laboratorio de Pesquisa em Leishmaniose/LPL. Instituto Oswaldo Cruz-FIOCRUZ, Brasil); para las pruebas de sensibilidad y de capacidad de discriminación entre especies de Leishmania de las reacciones de PCR y como controles para comparación en las reacciones de caracterización.
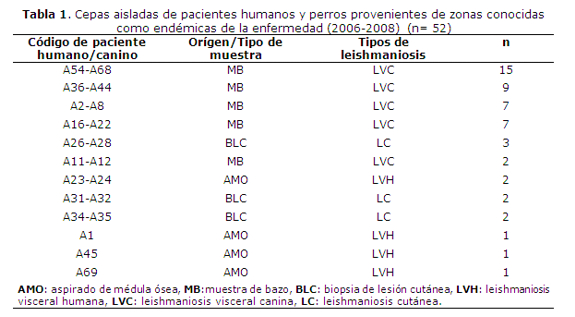
Fueron estudiadas 52 cepas de Leishmania aisladas de 7 pacientes con leishmaniosis tegumentaria (LT), de 5 pacientes con leishmaniosis visceral humana (LVH) y de 40 perros con leishmaniosis visceral canina (LVC) en el periodo comprendido entre los años 2006 y 2008 (tabla 1).
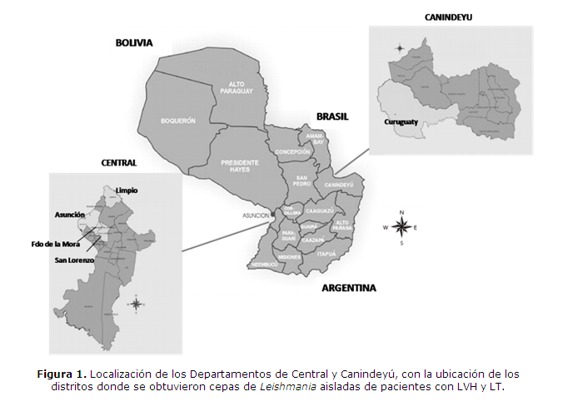
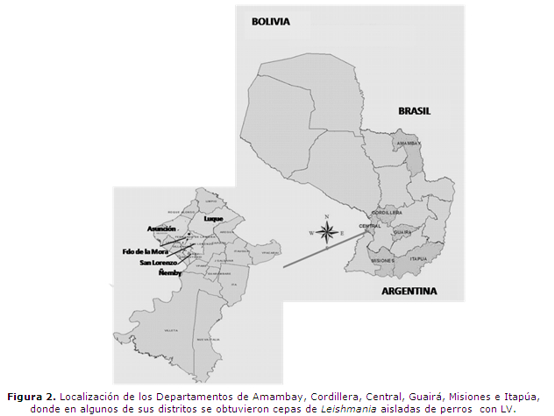
Las cepas de Leishmania aisladas, de LT fueron obtenidas por cultivo de biopsia de la lesión de piel y provenían de casos ocurridos durante el brote en la localidad de Tava Yopoi y alrededores, distrito de Curuguaty, departamento de Canindeyú y fueron reportados anteriormente (15) (figura 1). Los aislados de LVH fueron obtenidos a través del aspirado de médula ósea y provenían de casos ocurridos en Asunción, capital del país y en distritos cercanos como Limpio, Fernando de la Mora y San Lorenzo del Departamento Central (figura 1). Las cepas de LVC fueron obtenidas de muestras de bazo de perros con diagnóstico serológico positivo para LVC, 12 provenían de las zonas de Asunción y distritos cercanos; como Fernando de la Mora, San Lorenzo, Ñemby y Luque del Departamento Central y 28 de los departamentos de Itapúa, Amambay, Guairá, Misiones y Cordillera (figura 2).
3. Extracción de ADN: la extracción de ADN de los aislados se realizó por el método de fenol-cloroformo. Los cultivos fueron sometidos previamente a una lisis de proteínas (over night a 55°C) mediante un tampón de lisis (10 mM Tris, 10% SDS, 200 µg/mL), proteinasa K (concentración final: 200 µg/mL). El proceso de extracción consistió en dos pasos de fenol-cloroformo (25:24) y uno de cloroformo puro final. El ADN fue precipitado con acetato de sodio 3M, pH 5.3, etanol 99,5% p.a. frío y una noche a –20°C. Finalmente, el producto de la extracción se resuspendió en 100 µl de agua destilada, siendo empleado como molde para la reacción de PCR. Esta metodología desarrollada se basó en la combinación de diversos protocolos preestablecidos y modificados (7,16), tomando como referencia la propuesta por Sambrook et al (16).
4. Reacción en cadena de la polimerasa (PCR): se empleó el siguiente par de cebadores: Fme: 5´-TAT TGG TAT GCG AAA CTT CCG-3´ (Forward) y Rme: 5´-ACA GAA ACT GAT ACT TAT ATA GCG-3´ (Reverse), diseñados por Marfurt et al. (6) que amplifican la región del spliced leader miniexón (SLME). Las reacciones de PCR se realizaron en un termociclador DNA Engine PTC 200 (BioRad, California, USA) con el siguiente esquema de ciclado: 5 min a 94°C, seguido de 35 ciclos de 30 seg a 94°C, 30 seg a 54°C, y 45 seg a 72°C. La mezcla de reacción contenía: Buffer 1X, 1,5 mM MgCl2, 5% DMSO (SIGMA, USA), 0,2 mM dNTPs (BIOLINE, UK), 0,2 mM de cada cebador, 1 unidad de Taq polimerasa (FERMENTAS, EU) para un volumen total de 50 µL.
5. Análisis de los productos amplificados: los productos amplificados los cuales tienen un tamaño aproximado entre 220 y 226 pb para el subgénero Viannia y entre 283 y 443 pb para el subgénero Leishmania fueron analizados por electroforesis en geles de agarosa al 2%, empleando solución tampón TAE (0.04 M Tris-acetato, 0.001 M EDTA) (15). La electroforesis se realizó en cámaras MUPID-3 (Japón) a 100 V durante 30 minutos. Los tamaños de las bandas se estimaron empleando un marcador de tamaño molecular de 100 pb (hasta 2000 pb) (BIOLINE, UK) y visualizados con luz UV previa tinción con bromuro de etidio (0,5 µg/mL). Los geles de agarosa fueron registrados empleando un analizador de imágenes (KODAK Digital Science DC120, USA).
6. Digestión con enzimas de restricción (RFLP): los productos de PCR obtenidos con los cebadores Fme y Rme fueron digeridos con las enzimas de restricción Hae III (10 U/µL) y Nco I (10 U/µL) (New England Biolabs®, USA) incubando las muestras a 37º C durante toda la noche en baño o estufa y los patrones obtenidos fueron visualizados en geles de poliacrilamida al 10%, utilizando solución tampón de corrida TBE (0.045M Tris-borato, 0.001 M EDTA). La corrida se realizó durante 90 minutos a 5 Watts constante equivalentes a 100 Volts. Para la confirmación de los tamaños de los productos de digestión los patrones fueron comparados con un marcador de tamaño molecular de 100 bp (hasta 2000 pb) (BIOLINE, UK). Los resultados fueron registrados con un analizador de geles y analizados con el software de KODAK Digital Science DC 120 (USA).
7. Prueba de sensibilidad de la PCR: para determinar la sensibilidad analítica de la reacción de PCR fueron preparadas diluciones seriadas de ADN de cepas de referencia de L. (V.) braziliensis, L. chagasi y L. (L.) amazonensis. La cantidad de ADN probada oscilaba entre 104 y 1 parásito, equivalente de 1 ng a 100 fg de ADN.
También fue determinada la capacidad de la técnica para diferenciar entre especies de Leishmania. Se realizaron ensayos utilizando cultivos de cepas de referencia de L. (V.) braziliensis, L. (L.) amazonensis y L. chagasi y el posterior análisis de los tamaños de los fragmentos de restricción en correspondencia con el análisis del corte con enzimas (6).
8. Consideraciones éticas: el protocolo de este estudio ha sido aprobado por el Comité de Ética de la Investigación del Instituto de Investigaciones en Ciencias de la Salud. Los aislados en cultivo para este proyecto han sido obtenidos a partir de otro proyecto financiado por la Comunidad Europea que ha obtenido los consentimientos informados de los pacientes que han participado del estudio, las muestras han sido debidamente codificadas, respetando en todo momento la confidencialidad de los datos y resultados de los pacientes. Los pacientes han sido diagnosticados, tratados y controlados por los profesionales del Programa Nacional de Control de las Leishmaniosis del Ministerio de Salud Pública. Las muestras de bazo de perros fueron obtenidas de animales con serología positiva para LVC luego de la eutanasia de los mismos, con el consentimiento previo de sus dueños.
RESULTADOS
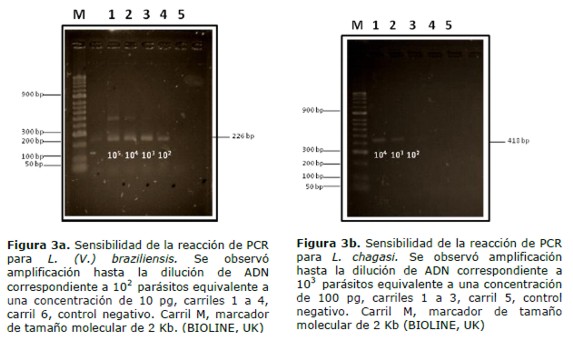
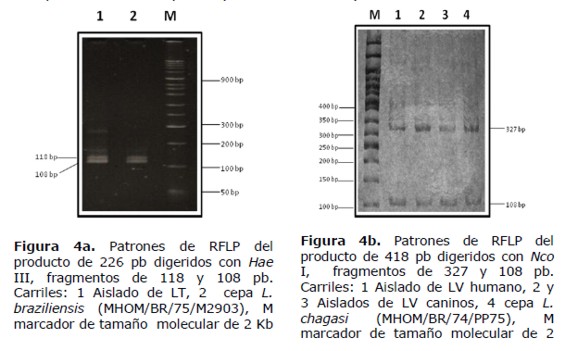
1. Prueba de sensibilidad de la PCR de la región SLME Se realizaron ensayos de sensibilidad analítica de la reacción de PCR, utilizando DNA aislado de cultivos de cepas de referencias de las especies L. (V.) braziliensis, L. (L.) amazonensis y L. chagasi. Se encontró que el límite de detección de ADN de L. (V.) braziliensis fue de 100 parásitos lo que equivale a 10 pg de ADN, dando un producto aproximado de 226 pb (figura 3a), para L. chagasi fue de 1000 parásitos, equivalente a 100 pg de ADN, con un producto aproximado de 418 pb (figura 3b) y para L. (L.) amazonensis fue de 1000 parásitos, correspondiente a 100 pg de ADN y con un producto aproximado a 285 pb (figura 3c).
2. Evaluación de la capacidad de la PCR-RFLP de la región SLME para identificar especies de Leishmania en aislados clínicos
Se realizaron ensayos para determinar la capacidad de la técnica para diferenciar entre especies de Leishmania amplificando DNA de purificado de aislados humanos de LC y LV, aislados caninos de LV y cepas de referencia y el posterior análisis de los tamaños de los fragmentos de restricción (RFLP) (figuras 4a y 4b) en correspondencia con el análisis del corte con enzimas, según Marfurt et al (6). El análisis de los patrones de restricción obtenidos (RFLP) muestra fragmentos de 118 y 108 pb con Hae III, que corresponden a L. (V.) braziliensis (figura 4a) y fragmentos de 327 y 108 pb con NcoI que corresponden a L. chagasi (figura 4b).El fragmento de 285 pb correspondiente a L. (L.) amazonensis no fue cortado por ninguna de las dos enzimas HaeIII y Nco I, en correspondencia con lo esperado (datos no mostrados).
3. Identificación de los aislados clínicos por PCR-RFLP
De los 52 aislados clínicos analizados, inicialmente fueron detectados productos de amplificación correspondientes al subgénero Viannia, (entre 223 y 226 bp) en 7 aislados de humanos y correspondientes al subgénero Leishmania (entre 418 y 443 bp), en 5 aislados de humanos y en 40 de caninos.
Posteriormente con el análisis de los patrones de bandas digeridos con Hae III, los 7 aislados que amplificaron productos asociados con el subgénero Viannia generaron los fragmentos de 118 y 108 bp al igual que la cepa de referencia de L. braziliensis y para los 45 aislados que amplificaron fragmentos entre 418 y 443 pb asociados con el subgénero Leishmania generaron los fragmentos de 327 y 108 bp con Nco I al igual que la cepa de referencia L. chagasi.
DISCUSIÓN
El presente ensayo de genotipificación basado en PCR-RFLP del gen miniexón mostró la capacidad de detectar en un primer paso los complejos de Leishmania circulantes en el Paraguay en una reacción única de PCR y en un segundo paso de digestión con enzimas de restricción fue posible identificar las cepas dentro de los complejos de Leishmania prevalentes en Paraguay. La mayoría de los ensayos para diagnóstico son basados en secuencias genómicas repetitivas o secuencias del kDNA, pero están limitados a la detección a nivel de géneros y complejos del parásito (6,7). La identificación a nivel de especies es una importante ventaja; en el caso de leishmaniosis cutánea, por ejemplo, porque diferentes especies pueden causar lesiones cutáneas similares y requerir distintos tratamientos o esquemas de tratamiento y además, la información es epidemiológicamente relevante para identificar el foco de transmisión activa y el diseño de estrategias de control (17).
Con el análisis de los patrones de bandas, se define como especie circulante a L. braziliensis, causante de la leishmaniosis cutánea, en concordancia con otras publicaciones (1,17), aunque en países como Brasil la diversidad de especies de parásitos es grande, donde al menos siete especies han sido descritas como agente etiológicos de la forma cutánea de la enfermedad; L. braziliensis, L. guyanensis, L. shawi, L. lainsoni, L. naiffi, L. lindenbergi y L. amazonensis (14).
Si bien la técnica no permite discriminar entre L. braziliensis y L. peruviana, según lo publicado por Marfurt et al, porque generan el mismo patrón de bandas con el corte con la enzima usada en el estudio, esto no es un problema para nosotros ya que no hay antecedentes de L. peruviana, en nuestra zona, y se reporta esta especie limitada geográficamente a la zona alta de los Andes de Perú, pero por otro lado se podría emplear otros genes que permiten diferenciar estas dos especies, por ejemplo, usando el gen gp 63 (13), o el gen cpb ambos muestran patrones característicos para L. braziliensis y L. peruviana (18).
Por otro lado también se definió como circulante a la especie L. chagasi causante de la leishmaniosis visceral en distintas zonas del país. Se encontró el mismo patrón de bandas tanto en aislados de origen humano como los de origen canino y a su vez en los aislados de origen canino procedentes de la zona de Asunción y distritos de los departamentos; Central, Cordillera, Encarnación, Amambay, Guairá y Misiones. En vista que la especie circulante es única, se podría buscar en estudios posteriores otros genes como blanco que permitan identificar genotipos o perfiles genéticos diferentes entre cepas de esta especie (19,20) y que puedan correlacionarse con algún factor epidemiológico.
Otra ventaja que presenta esta técnica, es que a diferencia de otras utilizadas para la identificación de especies; como electroforesis de isoenzimas, serodemas, lo cual requiere el aislamiento del parásito, esta técnica no requiere del cultivo de parásitos y puede ser empleada directamente en muestras clínicas. Adicionalmente mostró ser sensible debido al carácter repetitivo del marcador genético empleado (6) y se pudo observar en el presente estudio, que el límite de detección del ensayo fue de 10 pg, asumiendo que el ADN total contenido en el parásito de Leishmania es aproximadamente 100 fg por genoma (6), igual sensibilidad fue reportada por Acardi et al (20).
Los hallazgos descritos en este trabajo tienen una implicación a nivel epidemiológico, pudiendo contribuir a seguir diseñando e intensificando las estrategias de vigilancia y control de la enfermedad en sus diversas formas en todas las áreas consideradas como endémicas dentro del territorio nacional.
Es interesante resaltar que este trabajo es el primero en el país en realizar la caracterización molecular a nivel de especies de Leishmania con un número significativo de aislados, de distintas procedencias y tanto de la forma visceral como cutánea de la enfermedad.
AGRADECIMIENTOS
Al Dr. Edgar Galeano y a la Dra. Mónica Ozorio por su colaboración en la toma de muestra de perros, a la Dra. Cristina Romero por su colaboración en el mantenimiento de las cepas. Este trabajo ha recibido financiación parcial de la Unión Europea (Proyecto LeishEpiNetSA, Contr. No. 15407).
REFERENCIAS BIBLIOGRAFICAS
1. Cupolillo E, Brahim LR, Toaldo CB, Oliveira-Neto MP, Felinto de Brito ME, Falqueto A, et al. Genetic polymorphism and molecular epidemiology of Leishmania (Viannia) braziliensis from different hosts and geographic areas in Brazil. J. Clin. Microbiol. 2003; 41 (7): 3126-32. [ Links ]
2.World Health Organization. Control of the leishmaniases. Geneva: World Health Organization; 1990. (Technical Report Series, 793). [ Links ]
3.Grimaldi G, Tesh RB. Leishmaniases of the New World: current concepts and implications for future research. Clin Microbiol Rev. 1993; 6(3): 230-50. [ Links ]
4. Bañuls A, Hide M, Prugnolle F. Leishmania and the leishmaniases: a parasite genetic update and advances in taxonomy, epidemiology and pathogenicity in humans. Adv Parasitol 2007; 64:1-109. [ Links ]
5. García AL, Kindt A, Quispe-Tintaya KW, Bermudez H, Llanos A, Arévalo J, et al. American tegumentary leishmaniasis: antigen-gene polymorphism, taxonomy and clinical pleomorphism. Infect. Gen. and Evol. 2005; 5: 109-16. [ Links ]
6. Marfurt J, Niederwieser I, Makia ND, Beck H, Felger I. Diagnostic genotyping of Old and New World Leishmania species by PCR-RFLP. Diagn Microbiol Infect Dis 2003; 46(2): 115-24. [ Links ]
7. Harris E, Kropp G, Belli A, Rodriguez B, Agabian N. Single-step multiplex PCR assay for characterization of New World Leishmania complexes. J Clin Microbiol 1998; 36 (7): 1989-95. [ Links ]
8. Servicio Nacional de Erradicación de Enfermedades Vectoriales (SENEPA- MSP y BS) Boletín informativo semanal N° 11. Asunción; 2010. [ Links ]
9. Servicio Nacional de Erradicación de Enfermedades Vectoriales (SENEPA- MSP y BS) Boletín informativo semanal N° 7. Asunción; 2010. [ Links ]
10. Van Eys, GJ, Schoone GJ, Kroon NC, Ebeling SB. Sequence análisis of small subunit RNA genes and its use for detection and identification of Leishmania parasites. Mol. Biochem. Parasitol. 1992; 51, 133-42. [ Links ]
11. Piarroux R, Gambarelli F, Dumon H, Fontes M, Dunan S, Mary C, et al. Comparison of PCR with direct examination of bone marrow aspiration, myeloculture, and serology for diagnosis of visceral Leishmaniasis in immunocompromised patients. J Clin Microbiol 1994; 32(3): 746-9. [ Links ]
12. Cupolillo E, Grimaldi G, Momen H, Beverley SM. Intergenic region typing (IRT): a rapid molecular approach to the characterization and evolution of Leishmania. Mol. Biochem. Parasitol. 1995; 73: 145-55. [ Links ]
13. Victoir K, Bañuls AL, Arévalo J, Llanos-Cuentas A, Hamers R, Noel S, et al. The gp63 gene locus, a target for genetic characterization of Leishmania belonging to subgenus. Viannia Parasitol. 1998; 117: 1-13. [ Links ]
14. Rodgers MR, Popper SJ, Wirth DF. Amplification of kinetoplast DNA as a tool in the detection and diagnosis of Leishmania. Exp Parasitol. 1990; 71(3): 267-75. [ Links ]
15. Sambrook J, Fritsch E, Maniatis T. Molecular Cloning. A Laboratory Manual. New York : Cold Spring Harbor Laboratory Press; 1994. [ Links ]
16. Belli A, Rodriguez B, Aviles H, Harris E. Simplified polymerase chain reaction detection of new world Leishmania in clinical specimens of cutaneous leishmaniasis. Am J Trop Med Hyg 1998; 58(1): 102-9. [ Links ]
17. Perez JE, Veland N, Espinosa D, Torres K, Ogusuku E, Llanos-Cuentas A, et al. Isolation and molecular identification of Leishmania (Viannia) peruviana from naturally infected Lutzomyia peruensis (Diptera: Psychodidae) in the Peruvian Andes. Mem Inst Oswaldo Cruz 2007; 102(5): 655-8. [ Links ]
18. Guerbouj S, Victoir K, Guizani I, Seridi N, Nuwayri-Salti N, Belkaid M, et al. Gp 63 gene polymorphism and population structure of Leishmania donovani complex: influence of the host selection pressure?. Parasitology. 2001; 122: 25-35. [ Links ]
19. Peres Alonso D, Lamounier Costa D, Lopes de Mendonça I, Nery Costa CH, Martins Ribolla PE. Short Report: Heterogeneity of Leishmania infantum chagasi Kinetoplast DNA in Teresina (Brazil). Am J Trop Med Hyg. 2010; 82 (5): 819–21. [ Links ]
20. Acardi SA, Liotta DJ, Santini MS, Romagosa CM, Salomón OD. Detection of Leishmania infantum in naturally infected Lutzomyia longipalpis (Diptera: Psychodidae: Phlebotominae) and Canis familiaris in Misiones, Argentina: the first report of a PCR-RFLP and sequencing-based confirmation assay. Mem Inst Oswaldo Cruz 2010; 105 (6): 796-9. [ Links ]
*Autor Correspondiente: Dra. Eva Nara, Dpto. de Biología Molecular y Genética. Instituto de Investigaciones en Ciencias de la Salud. UNA. Paraguay.
Email:megunara@hotmail.com.
Fecha de recepción: abril de 2012, Fecha de aceptación: mayo 2012.