Introducción
Los hongos entomopatógenos (HE) se consideran componentes importantes de un manejo integrado de plagas, en particular para un sistema silvopastoril, sea esto como biopesticidas o como microorganismos naturales conservados en el medio ambiente (Quesada-Moraga et al., 2007). Sin embargo, hay poca información como potencial supresor natural de insectos y sus posibles efectos sobre los insectos bajo un manejo de la silvicultura. Los cambios en los paisajes naturales, debido a la intensificación agrícola, han causado una pérdida considerable de la biodiversidad y, por consiguiente, diezmar los servicios de un ecosistema. Sin embargo, la adopción de prácticas más amigables como el sistema silvopastoril puede fomentar condiciones abióticas y bióticas que conserven la biodiversidad y, en consecuencia, pueden restablecer la provisión de servicios naturales a los ecosistemas.
Existen más de 700 especies de entomopatógenas derivadas de la división de Ascomycota y Entomophthoromycota (Hibbett et al., 2007; Humber, 2012; Gryganskyi et al., 2013) tienen el potencial de controlar un rango de insectos plaga con su acción de contacto única; constituyen un grupo heterogéneo desde el punto de vista sistemático y también presentan diferencias en cuanto a su biología, actúan por la vía tegumentaria y su virulencia frente a insectos, les convierte en un importante factor de regulación natural de sus poblaciones (Quesada-Moraga et al., 2007).
El objetivo general del presente trabajo fue identificar HE presentes en la zona de influencia de Atta sexdens (L., 1758) en un sistema silvopastoril (Urochloa brizantha MG-5 y Eucalyptus spp.); y cuyos objetivos específicos fueron: a) Colectar del área de estudio muestras de artrópodos muertos con crecimiento fúngico o por otras causas; b) Inspeccionar fuentes de los servicios naturales de un ecosistema, en suelos y área de la rizosfera de la planta, con el uso de la técnica de insecto trampa c) Identificar con el análisis molecular los controladores biológicos representativos obtenidos durante la inspección de campo. (Global Biodiversity Information Facility Secretariat (GBIF), 2023)
Materiales y Métodos
Se colectaron muestras de suelos, también se colectaron suelos adheridos a raíces de las plantas así también insectos muertos colonizados y no colonizados, de una parcela con sistema silvopastoril (Urochloa brizantha MG-5 y Eucaliptus sp.), de la localidad de Santa Rosa del Aguary (S23°43’39’’ W56°27’17’’), Departamento de San Pedro, Paraguay, en dos oportunidades, noviembre y diciembre de 2019, utilizando el área de influencia de 20 nidos de hormigas cortadoras de hojas (Atta sexdens, Hymenoptera: Formicedae), de los cuales 10 nidos ubicados en áreas tratadas frecuentemente con productos químicos (Fipronil) y 10 nidos donde se dejó de utilizar tratamientos químicos por un año. Estos estaban localizados en un área de aproximadamente 5.000 m2 de manejo silvopastoril combinando U. brizantha MG-5 con Eucaliptus sp. (distante 2m x 5m entre planta e hilera). En total, se utilizaron 100 puntos de muestreo. Cada punto de muestreo comprende un área de 0,25 m2 con un intervalo de 2 m y, cada nido con un área de 8 a 10 m ancho y 15 a 20 m de largo. La distancia promedio de muestreo entre nidos fue de 40 a 50 m, localizada al azar (Figura 1).
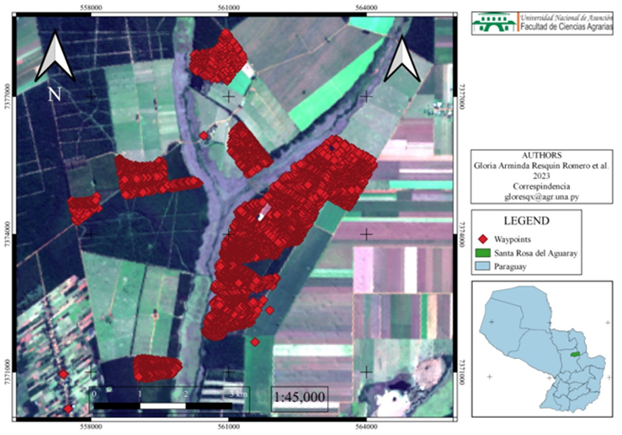
Figura 1. Geolocalización de nidos de hormigas cortadoras de hojas (Atta sexdens) e incidencia real en áreas rurales, parcelas silvopastoriles comerciales (Urochloa brizantha cv. MG-5 y Eucaliptus sp.)
La recolección de suelo se realizó en plena estación húmeda, a una profundidad de 0 a 10 cm, utilizando una barrena de metal para extraer suelos. También se extrajo raíces de plantas predominantes encontradas en cada punto de muestreo, se usó el suelo que se desprendió de las raíces para aislar los hongos (Figura 2 y 3).

Figura 2. A-B-C. Formación de nidos de hormigas cortadoras registradas bajo un sistema silvopastoril (Urochloa brizantha MG-5 y Eucaliptus sp.). Colecta de muestras: D. Suelos, E. Raíces de poáceas, E. Insectos. Localidad: Santa Rosa del Aguaray, Dpto. San Pedro, Paraguay., año 2019.
Cada muestra de suelo fue dividida en tres partes y distribuidas en bolsas de plásticos transparente de 200 ml. Una parte del suelo (200 g) fueron utilizadas para la detección de HE y otra se guardaron como material de reposición, en caso de pérdida de material. Todas estas muestras fueron conservadas a temperatura 4°C. No se utilizó control alguno en cuanto a sustrato suelo y raíces del área de la rizosfera se refiere.

Figura 3. A-B-C-D-E-F. Macromorfología de A. sexdens micosados colectados del área de influencia de nidos de hormigas cortadoras. G-H-I. Utilización de la técnica de insectos trampa (reinas aladas) como cebos para captura de hongos entomopatógenos. J-L. Obrera micosada por Beauveria bassiana en condiciones de cámara húmeda. K-M. Reinas aladas micosadas por Metarhizium sp. obtenida con uso de la técnica de insectos trampa en suelo y del área de la rizosfera de poáceas.
También se colectó insectos muertos colonizados y no colonizados in situ, particularmente poblaciones de hormigas; también otras poblaciones de insectos como cercopideos (Mahanarva fimbriolata) (Figura 4). Todas estas muestras fueron etiquetadas de acuerdo con el origen y transportados en una conservadora al laboratorio del Área de Protección Vegetal (FCA-UNA).

Figura 4. Macromorfología de otros insectos colectados del área de influencia de nidos de hormigas cortadoras. A. Adultos de Mahanarva fimbriolata. B-C-D. M. fimbriolata micosados por Metarhizium sp. E-F. Nifas de M. fimbriolata micosados por B. bassiana. G. Estado ninfal.
Para el aislamiento de HE de suelos y del área de la rizosfera de pasturas se utilizó la técnica de insectos trampa (Zimmermman, 1986), a base de hormigas reinas de Atta sexdens. Estas hormigas reinas fueron capturadas y colectadas al momento cuando se disponían a abandonar el nido materno para copular con los zánganos, en el mes de diciembre de 2019. Para aislar el HE de insectos colonizadas y no colonizadas de muestras de suelo y rizosfera obtenidas del campo, se han adoptado dos métodos de acuerdo con la condición del insecto muerto (Garrido-Jurado et al., 2020), uno por transferencia conidial en placas de Petri con contenido de medio de cultivo selectivo y el otro método, los insectos sin crecimiento fúngico externo visible, se desinfestaron superficialmente y fueron incubados en condiciones de cámara húmeda. Después de 7 a 10 días de incubación a 25°C, se aislaron los hongos desarrollados. El ensayo se realizó por triplicado. Se eligieron colonias que mostraron características compatibles a los conocidos géneros de HE y algunos ocasionales hongos.
Identificación y análisis moleculares
La identificación de los hongos a nivel de género se realizó observando las estructuras microscópicas de cada aislado, bajo microscopio óptico (Microscopio Leica DM500 Alemania). Para la identificación de las estructuras fúngicas se utilizó la llave de identificación propuesto por Barnett y Hunter (1998).
El aislamiento, purificación e identificación morfológica se realizó en el laboratorio del Área de Protección Vegetal de la FCA, UNA y la amplificación del gen e identificación molecular de los aislados se realizó en el laboratorio del Departamento de Agronomía, ETSIAM, Universidad de Córdoba, España.
El ADN total de los aislados se extrajo a partir de micelio liofilizado siguiendo el método de Raeder y Broda (1985). Se amplificó el gen del factor de elongación 1 α (EF-1α), se secuenció y se analizó en cada uno de los aislados. La amplificación se realizó con los cebadores tef1fw (50-GTGAGCGTGGTATCACCA-30) (O’Donnell et al., 1998) y 1750-R (50-GACGCATGTCACGGACGGC-30) (Garrido-Jurado et al., 2011) con las siguientes modificaciones: en un volumen total de reacción de 50 μl que contenía 1,5 μl de ADN genómico, 1X DreamTaqTM buffer con MgCl2 (ThermoScientific, Waltham, MA, USA), 20 nM de cada cebador, 40 nM de mezcla de dNTPs y 0,25 U de DreamTaqTM DNA polymerase (Thermo Scientific, Waltham, MA, USA). Las condiciones de amplificación fueron las descritas por Garrido-Jurado et al. (2011). Los productos de PCR se visualizaron en gel de agarosa al 1%, se purificaron utilizando Geneclean II kit system (QBiogene, Inc., Carlsbad, CA) y se secuenciaron por la empresa Stab Vida (Caparica, Portugal). Se analizaron las secuencias EF1-α de los aislados y el alineamiento entre estas y secuencias de la misma región de especies cercanas se realizó usando el programa MegAlign [DNASTAR package, London, UK). El análisis filogenético se llevó a cabo mediante el programa MEGA 11 (Tamura et al., 2021). Se realizó el árbol de máxima verosimilitud usando el criterio TBR (tree-bisection-reconnection), el soporte de las ramas se hizo mediante un análisis de bootstrap de 1000 réplicas. Se utilizó una distribución discreta Gamma para calcular el modelo de la tasa de cambio entre sitios (5 categorías (+G, parámetro = 0,1893)]. Hubo un total de 853 posiciones en el dataset final. Para calcular la autocorrelación de tasas evolucionarias se ha utilizado el método Corrtest (Tao et al., 2019).
Los datos generados sobre incidencia de insectos micosados fueron sometidos a Análisis de Varianza (ANOVA) y las medias por el test de Tukey (p≤ 0,05) (Statistix, 2013). La comparación de las poblaciones de hormigas cortadoras se realizó mediante la prueba no paramétrica Kruskal-Wallis para muestras independientes (SPSS 25).
Resultados y Discusión
Efectos de los hongos entomopatógenos sobre insectos trampa
Las esporas asexuales o conidios de los hongos entomopatógenos (HE) Beauveria sp. y Metarhizium sp., son los responsables de la infección de insectos y que se encuentran dispersos por el medio donde se encuentran sus hospedantes, en este caso sobre las hormigas cortadoras de hojas. En los 20 nidos muestreados, 10 nidos ubicados en áreas tratadas aplicación continua del producto químico (Fipronil), tanto en la zona de la rizosfera como en el resto del suelo, se han observado poblaciones de reinas aladas muertas por la infección de Metarhizium sp. y Beauveria sp. (Tabla 1). Sin embargo, no hubo diferencias significativas entre nidos o entre las muestras de suelo y la rizosfera (a partir de ahora sitio) tanto para las reinas que mostraron crecimiento de Metarhizium sp. (F9,19=1,95, p>0.05 entre nidos; F1,19=4,31, p>0,05 entre sitios), de Beauveria sp. (F9,19=0,94, p>0,05 entre nidos; F1,19=0,29, p>0,05 entre sitios) o muertas por otras causas (F9,19=1,38, p>0,05 entre nidos; F1,19=1,98, p>0,05 entre sitios) (Figura 5). Sin embargo, mostró diferencias significativas entre las dos situaciones de las parcelas de muestreo tanto para variable hormigas colonizadas por el hongo (H=114,249; p=0,0001) como hormigas muertas por otras causas (H=155,021; p=0,0001) (Figura 6).
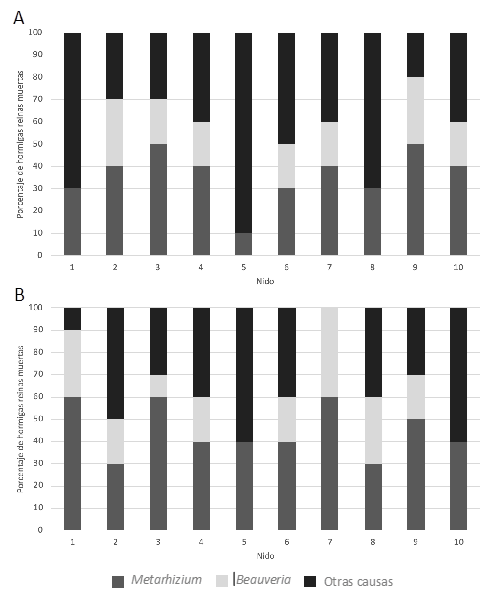
Figura 5. Porcentaje de hormigas reinas encontradas muertas por infección fúngica con Metarhizium sp. y Beauveria sp. o por otras causas para los 10 nidos ubicados en áreas de aplicación continua de Fipronil. A) Aislando del resto del suelo; B) Aislando del suelo adheridas a las raíces de las plantas.
En el caso de aquellas reinas que mostraron crecimiento de Metarhizium sp., aunque las diferencias entre sitios no fueron significativas, sí que se observó un mayor porcentaje de ellas en la zona de la rizosfera (Figura 6). Esto concuerda con los trabajos de Hu y Leger (2002) y Barelli et al. (2018) en los que indica que el género Metarhizium se encuentra preferentemente en la zona de la rizosfera.
El análisis de Kruskall-Wallis evidenció la ausencia de diferencias significativas entre los nidos para la variable hormigas colonizadas por el hongo (H=2,463; p>0,05) y para la variable muertas por otras causas (H=3,806; p>0,05), respectivamente (Figura 6).
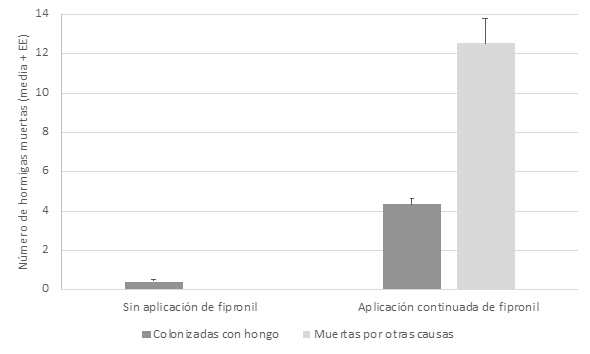
Figura 6. Número de hormigas cortadoras encontradas muertas por infección fúngica o por otras causas en una zona sin aplicación de Fipronil y en otra de aplicación continuada del mismo.
Se colectaron los individuos de 30 puntos presentes en la zona de muestreo sin aplicación de Fipronil. Se encontraron un total de 368 ninfas estando el 38 % (n=144) infectadas por hongos entomopatógenos, así como 31 adultos colonizados también por hongos entomopatógenos.
Tabla 1. Identificación de los aislados de hongos entomopatógenos colectados del área de influencia de nidos de hormigas cortadoras.
| Identificación/ aislados | Muestras s/ insectos trampas /Hospedantes | ||
|---|---|---|---|
| Suelo | Suelo adherida a las raíces | Insectos colonizados colectados del campo | |
| Beauveria bassiana | x | x | x |
| Metarhizium sp. | x | x | x |
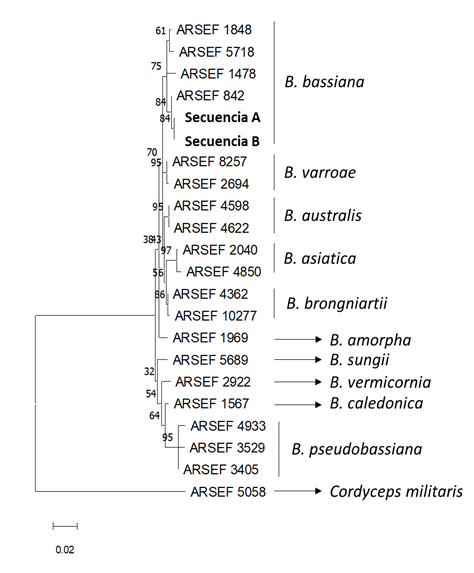
Para la identificación molecular de los hongos aislados, se obtuvieron dos aislados de los cadáveres de insectos pertenecientes a la especie Beauveria bassiana (Figura 7) y un aislado del género Metarhizium. Las secuencias obtenidas, secuencias de B. bassiana A y B. bassiana B, han sido depositadas en GenBank con los números de acceso OR176989 y OR176990, respectivamente. Ambas secuencias se alinearon y compararon con 20 secuencias obtenidas de la base de datos pertenecientes a las especies B. bassiana, B. varroae, B. asiatica, B. australis, B. brongniartii, B. pseudobassiana, B. caledonica, B. vermicornia, B. sungii, B. amorpha y Cordyceps militaris. El árbol filogenético muestra como ambas secuencias se encuentran recogidas en el cluster de B. bassiana soportado por un bootstrap de 84 %, lo que haría necesario incluir más regiones definitorias del género Beauveria como BLOC (Rehner et al., 2011). No se rechazó la hipótesis nula de la tasa de independencia de las tasas evolutivas entre linajes en la filogenia de la Figura 7 (p>0,05). El análisis Corrtest (Tao et al., 2019) arrojó una puntuación de 2,61x10-6 y el logaritmo de verosimilitud del árbol evaluado es (-2146,49). En el Paraguay, existen trabajos realizados sobre eficacia de productos formulados no nativos utilizados contra Acromyrmex landolti fracticornis (Amarilla Salinas y Arias Ruiz Díaz, 2011). En este trabajo, reportamos la identificación de Beauveria bassiana y Metarhizium sp., dos especies obtenidas in situ de las hormigas cortadoras de hojas (Atta sexdens). Ambas especies con origen monofilético a las familias Cordycipitaceae y Clavicipitaceae perteneciente a la Clase Ascomycetes y Orden Hypocreales, respectivamente (Bischoff et al., 2009; Rehner et al., 2011).
Conclusión
Se reporta por primera vez, la técnica de insectos trampa con el uso de poblaciones de reinas aladas de Atta sexdens como medio-cebo para aislar hongos entomopatógenos.
Se identificó dos aislados de los hongos entomopatógenos Beauveria bassiana (A y B) y un género Metarhiziumsp. como potenciales controladores biológicos de hormigas cortadoras (Atta sedens) in situ.












 uBio
uBio