Introducción
Glaesserella parasuis puede encontrarse como comensal colonizante del tracto respiratorio superior de los cerdos, aunque también es un patógeno bacteriano de importancia en el sector porcino. Es agente etiológico de la enfermedad de Glässer, y patógeno secundario del Complejo Respiratorio Porcino (Costa-Hurtado et al., 2020; Schuwerk et al., 2020). Las infecciones descontroladas producidas por G. parasuis resultan en pérdidas económicas significativas en el sector porcino, causadas por diferentes variables como atraso en el crecimiento, aumento en el índice de conversión alimentaria, disminución en la ganancia de peso diario, costos derivados del uso de antimicrobianos, depreciación de las carcasas y altas tasas de mortalidad (Frandoloso & Resende de Macebo, 2022).
Las tasas de morbilidad y mortalidad asociadas con la enfermedad de Glässer pueden variar del 5 al 10%, dependiendo de la gravedad de la enfermedad y virulencia de la cepa de G. parasuis implicada (Aragon et al., 2019). La enfermedad generalmente inicia de forma súbita presentando síntomas tales como anorexia, fiebre, apatía, estornudos, tos, disnea, cianosis, conjuntivitis, claudicación o signos nerviosos, los lechones afectados pueden morir durante la forma aguda o evolucionar a la forma crónica en la que se observan artritis crónica y adherencias en las serosas. Las lesiones comúnmente observadas corresponden a pleuritis, pericarditis, peritonitis, oftalmitis, poliartritis y meningitis con exudado fibrinoso o serofibrinoso (Frandoloso & Resende de Macebo, 2022).
Se han descrito 15 serotipos (Ser) con diferentes grados de virulencia, siendo considerados de alta virulencia a Ser1, Ser5, Ser10, Ser12, Ser13 y Ser14; moderada virulencia a Ser2, Ser4, Ser8 y Ser15, y baja virulencia a Ser3, Ser6, Ser7, Ser9 y Ser11. Además, actualmente existe un porcentaje de aislados que no se pueden serotipificar (Olvera et al., 2012; Pires Espíndola et al., 2019; Schuwerk et al., 2020).
De esta manera resulta clave la identificación de las serovariedades asociadas con brotes regionales para el desarrollo de estrategias preventivas en cada región productora de cerdos, que permitan el desarrollo de vacunas que contengan antígenos para los serovares locales más prevalentes (Pires Espíndola et al., 2019), lo que a su vez ayuda a limitar el uso de antimicrobianos en la producción porcina. Estudios epidemiológicos en regiones productoras de cerdos de varios países demuestran la circulación de un grupo heterólogo de serotipos, siendo los serotipos 4 y 5 los más prevalentes en la mayoría de los países con alta producción porcina (Pires Espíndola et al., 2019). En Paraguay, no hay estudios sobre la prevalencia y distribución de los serotipos de G. parasuis circulantes en el país, conocimiento necesario para poder plantear estrategias de control y manejo de esta especie.
Materiales y métodos
Muestreo. El muestreo incluyó cinco granjas productoras de porcinos ubicadas en el departamento de Itapúa desde el mes de febrero del 2022 a marzo del 2023 (Figura 1). Se identificaron cerdos con síntomas respiratorios, incluyéndose un total de 62 animales. En la figura 1, se observa la geoposición de las granjas incluidas en el trabajo. En animales vivos se aplicaron métodos de sujeción adecuados, las fosas nasales fueron higienizadas y las muestras fueron colectadas con hisopos estériles en medio de transporte semisólido Stuart. Para animales muertos se colectaron muestras de pulmón o de cornetes durante las necropsias realizadas a campo, antes de las cinco horas post mortem. La cantidad de muestras por granja se especifica en la Figura 2.
Cultivo y aislamiento de microorganismos. Las muestras fueron colectadas en las granjas y transportadas al laboratorio, sembradas en Agar columbia Chocolate + NAD e incubadas por 48 hs a 37°C en atmósfera de microaerofilia. Se realizó tinción de ram a las colonias sospechosas, posteriormente se realizaron pruebas bioquímicas como Indol, Urea y Test de catalasa (Frandoloso & Resende de Macebo, 2022).
Extracción de ADN. Para la extracción de ADN se tomaron colonias crecidas en placa y fueron suspendidas en 200 μL de solución PBS, se agitó con vórtex, y posteriormente se utilizó el kit de purificación GeneJET™ Viral DNA/RNA (ThermoFisher Scientific) siguiendo las instrucciones del fabricante (Thermoscientific, 2021).
Tipificación molecular y virulencia. Para realizar la tipificación molecular se utilizaron los cebadores descritos por Howell (2015), con modificaciones hechas por Lacouture et al. (2017). Cada reacción de PCR consistió en 12,5 μL de 2x GoTaq® G2 Colorless Master Mix (Promega); 1 mM de cada uno de los cebadores, csp de agua libre de nucleasas y 2 μL de ADN extraído, con un volumen final de 25 µL. Las condiciones de ciclado fueron las mismas descritas por Lacouture et al., (2017). Para identificar la presencia de genes relacionados a virulencia se utilizó la metodología descrita por Galofré-Mila et al., (2017) para la amplificación de genes de virulencia vtaA.
Análisis de los datos: Se clasificaron los resultados teniendo en cuenta la granja de procedencia, los mismos fueron analizados con estadística descriptiva, utilizando el Software RStudio 2022.07.0.
Resultados y Discusión
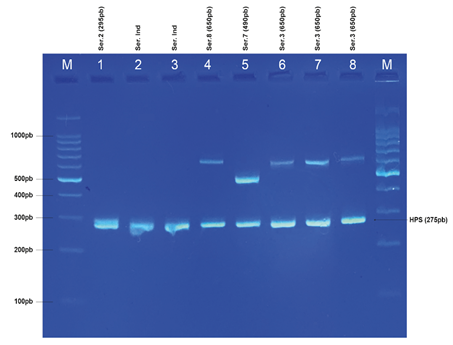
Se identificó la presencia de G. parasuis en las 5 granjas incluidas en este estudio (Figura 2). Un total de 12 cepas fueron aisladas, identificándose el serotipo al que pertenecen el 50% (6/12) de los aislados, perteneciendo a cuatro de los 15 (Ser2, Ser3, Ser7 y Ser8) serotipos reconocidos, no correspondiendo los demás aislados a ningún serotipo conocido actualmente, identificándose como NT (No Tipificables, Figura 2 y 3), correspondiéndose con lo reportado para otros países (Pires Espíndola et al., 2019; Schuwerk et al., 2020), lo cual estaría indicando la posible existencia de más serotipos que los 15 únicos reportados actualmente, evidenciando la necesidad de estudiar estas cepas NT. No se logró verificar la presencia de los serotipos 4 y 5, considerados como los más prevalentes en la mayoría de los países con alta producción porcina (Pires Espíndola et al., 2019). Es importante destacar que el 80,65% de las muestras sintomáticas, fueron ocasionadas por otras etiologías (OE), causadas por otros agentes. G. parasuis es una especie bacteriana heterogénea que comprende cepas con amplias diferencias en virulencia. Esta dualidad debe tenerse en cuenta a la hora de diagnosticar y controlar la enfermedad (Costa-Hurtado et al., 2020). Es importante destacar que, prácticamente cada granja presentó un serotipo específico, pudiendo esto relacionarse a la procedencia de los cerdos, como lo reportado en otros países productores (Pires Espíndola et al., 2019). Considerando la alta diversidad antigénica intrínseca de las diferentes serovariedades de este microorganismo, es imprescindible caracterizar minuciosamente las cepas envueltas en los casos para desarrollar vacunas adecuadas que a su vez ayuden a reducir la utilización de antimicrobianos de forma preventiva y disminuyan las probabilidades de crear resistencias en cepas clínicas como ya ocurre en Brasil (Frandoloso y Resende de Macebo, 2022).
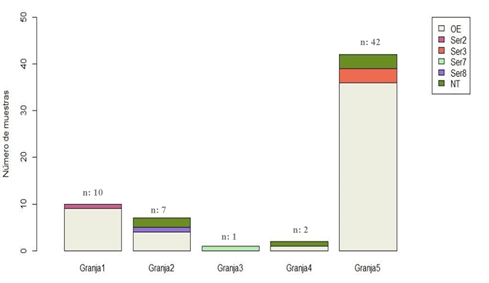
Figura 2. Distribución de muestras y serotipos encontrados en cada una de las granjas. OE: Otra etiología, G. parasuis: Ser2: Serotipo 2, Ser3: Serotipo 3, Ser7: Serotipo 7, Ser8: Serotipo 8, NT: No tipificable.
Figura 3. Perfil de la PCR multiplex para cada uno de los aislados de G. parasuis encontrados. Agarosa al 2%. M: marcador molecular; 1-8: muestras.
En cuanto a la identificación de genes relacionados a virulencia, los resultados indicaron que la amplificación del gen VtaA dio positivo en 11 de 12 de los aislados bacterianos. Aquellas que fueron positivas se obtuvieron de muestras de pulmón (9/11, 82%), cornetes (1/11, 9%) e hisopado nasal (1/11, 9%). La única cepa que dio negativo para el gen de virulencia se obtuvo de una muestra de hisopado nasal. Estos resultados sugieren que la mayoría de los aislamientos estudiados positivos al gen VtaA pueden presentar mecanismos de resistencia al suero y a la fagocitosis. Estos dos mecanismos se han reconocido como importantes factores de virulencia en la enfermedad causada por G. parasuis (Cerdà-Cuéllar & Aragon, 2008; Olvera et al., 2012).
De esta manera, se logró identificar la presencia y serotipificar los aislados de G. parasuis en cerdos con síntomas respiratorios procedentes de 5 granjas dedicadas a la producción porcina en Itapúa, Paraguay. Si bien los serotipos circulantes están clasificados dentro de los grupos de moderada y baja virulencia todos fueron aislados de animales sintomáticos, y en casi el total de los 12 aislados fue identificado el gen de virulencia VtaA.
De esta manera, este trabajo se convierte en el primer reporte de serotipos de G. parasuis en Itapúa-Paraguay, conocimiento fundamental para el control y manejo de este patógeno.












 uBio
uBio