1. INTRODUCCIÓN
La glicoproteína de la espiga (S) del SARS-CoV2 cumple un papel fundamental en el proceso de infección ya que participa en el reconocimiento de la proteína enzima conversora de angiotensina 2 (ACE2), para encaminarse a la fusión y posterior internalización del virus1. La interacción ocurre específicamente entre el ectodominio RBD de la glicoproteína viral de la espiga (S) y el dominio peptidasa (PD) de la ACE2, este acoplamiento conlleva al clivaje de la glicoproteína de la espiga (S) por parte de la furina y así dar lugar a una estructura postfusión que interactúa con la proteasa transmembrana serina 2 (TMPRSS2)2.
La glicoproteína de la espiga es un homotrímero conformado por tres protómeros (cadenas A, B y C) que se encuentran interaccionando entre sí (trimerización) para formar una estructura cuaternaria de prefusión con dos dominios (S1 y S2), conformados por los dominios N-terminal (NTD), el dominio de unión al receptor (RBD), péptido de fusión (FP), hélice central (CH), el dominio conector (CD), los dominios de repeticiones heptad 1 y 2 (HR1 y HR2), el dominio transmembranoso (TM) y finalmente el dominio C-terminal (CTD) (Figura 1) (1,3.
La comunidad científica ha puesto en la mira a esta glicoproteína de la espiga por el importante papel que cumple en el proceso de infección. Neutralizar sus funciones se ha convertido en uno de los objetivos fundamentales en la lucha contra la Covid-19, tanto en la elaboración de las vacunas, o en la búsqueda de fármacos o moléculas pequeñas de origen natural con posibles efectos sobre la glicoproteína4-7. Bongini y colaboradores8 sugieren que una posible estrategia para luchar contra el SARS-CoV2 sería evitando la formación de la estructura cuaternaria de la espiga viral, mediante el bloqueo de la trimerización de esta glicoproteína.
Las moléculas baicaleína (5,6,7-trihidroxiflavona) y baicalina (5,6-dihidroxi-7-O-glucuronido flavona), son flavonoides que se han descrito en las especies Scutellaria baicalensis, Scutellaria radix y Scutellaria lateriflora, sin embargo, también se cree que podrían encontrarse en otras especies del género Scutellaria, algunas de las cuales se encuentran presentes en países de Sudamérica9-10. La baicaleína es metabolizada en baicalina en la sangre, por lo que ambas formas baicaleína y la baicalina coexisten en el organismo11-13. Se han registrado de estas moléculas un sinnúmero de propiedades beneficiosas para la salud humana entre las cuales una de las más resaltantes son sus actividades principalmente antivirales14-16.

Figura 1 Estructura de protómeros A de la glicoproteína de la espiga (S) del SARS-CoV2. NTD: dominio N-terminal, RBD: dominio de unión a receptor, FP: péptido de fusión, HR1: dominio de repeticiones heptad 1, CH: hélice central, CD: dominio conector.
Estudios han demostrado actividades inhibitorias de la baicaleína ante el virus de la Influenza A/FM1/1/47 (H1N1), mediante interferencias en la síntesis de ARNm viral, también se ha observado actividades inhibitorias de la replicación del virus de la Encefalitis Japonesa (JEV), inhibición de la unión del virus de la Chikungunya (CHIKV) a células hospederas y sus efectos sobre la síntesis del ARN viral, así mismo, se ha descrito actividades virucidas extracelulares contra el Enterovirus 71 (EV-A71)17-20. Además, se ha observado que la baicalina podría ser capaz de inhibir a la proteína NS5 del virus del Zika (ZIKV), y también ejercer sinergismo a las actividades antivirales de la baicaleína en sistemas in vivo por la coexistencia de ambas21.
Recientemente, se ha observado que la baicaleína es capaz de disminuir la replicación del SARS-CoV2, y al igual que la baicalina podría bloquear la actividad de la proteasa viral 3CLpro22. Sin embargo, se ha descrito que el extracto etanólico de la S. baicalensis es capaz de inhibir in vitro en una baja proporción (<30%) la entrada del SARS-CoV2 en células Vero23.
En el presente estudio registramos in silico la probable interacción de las moléculas baicalina y baicaleína con el sitio de trimerización o en regiones próximas a esta dentro de la glicoproteína de la espiga (S) del SARS-CoV2, por lo que las mismas podrían bloquear o afectar a la trimerización de la glicoproteína viral durante su ensamblaje. Se espera esta investigación pueda apoyar al desarrollo de nuevas estrategias antivirales mediante el empleo de estos compuestos naturales para luchar contra el avance de la Covid-19.
2. MATERIALES Y MÉTODOS
2.1. Determinación y caracterización de sitios activos drogables en la región de trimerización de la glicoproteína de la espiga (S) del SARS-CoV2
Los sitios activos drogables en la región de trimerización de la espiga viral fueron identificados y caracterizados empleando la herramienta DoGSiteScorer24 para ello se utilizaron diferentes conformaciones de la glicoproteína de la espiga (ectodominio RBD abierto y cerrado) (PDB: 6VSB, 6VXX respectivamente). La caracterización fue realizada mediante la determinación de descriptores como las dimensiones del sitio activo drogable, grupos funcionales presentes en dicho sitio, así como la composición de residuos identificados en el mismo, también se estimó el índice de drogabilidad de dicho sitio24.
2.2. Análisis del acoplamiento molecular entre la baicaleína, baicalina y la glicoproteína de la espiga (S) del SARS-CoV2
La estructura molecular de la espiga S del SARS-CoV2 en sus diferentes conformaciones del dominio RBD (abierto y cerrado) fueron obtenidas de la base de datos Protein Data Bank (PDB: 6VSB, 6VXX)25. La estructura molecular de los ligandos baicaleína (CID: 5281605) y baicalina (CID: 64982) se obtuvieron de la base de datos PubChem26.
Se procedió a eliminar estructuras moleculares no proteicas que no fueron empleadas para el análisis, posteriormente se aplicó el método de campo de fuerza UFF (Universal Force Field) en conjunto con el algoritmo de optimización de gradientes conjugados a las estructuras moleculares de la baicaleína y baicalina. A continuación, se proporcionaron de cargas parciales y de átomos de hidrógeno polares presentes a pH de 7,4 a la proteína y a los ligandos, para todo esto se empleó el programa Avogadro 1.227.
Las pruebas de acoplamiento molecular se llevaron a cabo utilizando el modelo tipo receptor-ligando entre la glicoproteína de la espiga del SARS-CoV2 y las moléculas de baicaleína y baicalina en un total de cincuenta réplicas, a modo de disminuir el grado de incerteza en las simulaciones28, para ello se emplearon grillas de acoplamiento con dimensiones de 119x120x169 Å3 y 119x125x158 Å3 respectivamente, para lo cual se utilizó el programa AutoDock Vina29.
La visualización y el análisis de los complejos resultantes de las pruebas de acoplamiento molecular se realizaron empleando el programa Discovery Studio Visualizer v. 2030. Los datos obtenidos de energía libre de interacción (∆G b ) de los ligandos en acoplamiento con el sitio de trimerización o en regiones proximales a esta fueron analizados mediante el test de Mann-Whitney (5% de nivel de significancia), para ello se utilizó el programa PAST v.4.1031.
3. RESULTADOS Y DISCUSIÓN
El análisis de la glicoproteína de la espiga (S) en su conformación abierta del ectodominio RBD, demostró la presencia de un sitio altamente drogable importante, este se encuentra en la región de trimerización de la espiga con un índice de drogabilidad igual a 0,81. El análisis de la drogabilidad de un sitio nos permite conocer la aptitud de un sitio, un dominio o una región de la proteína para ser utilizado como blanco para la búsqueda o diseño de moléculas con potenciales inhibidores, a medida que el puntaje calculado es cercano a 1 mucho más drogable es el sitio24. Dicho sitio posee un volumen de 3702,14 Å3, una superficie de 4015,58 Å2 y una profundidad de 44,57 Å. Se encuentran 618 átomos, con la presencia de 87 grupos funcionales capaces de actuar como donadores en los puentes de hidrógeno, 195 grupos funcionales con capacidades de actuar como aceptores puentes de hidrógeno y 137 grupos funcionales capaces de formar interacciones hidrofóbicas, la proporción hidrofóbica del sitio fue del 33%. Además, se registró la presencia de residuos apolares en un 41%, residuos polares en un 36%, residuos básicos en un 15% y residuos ácidos en un 9%, siendo los residuos más abundantes la Gln, la Ala, la Leu, la Arg, la Ile, la Ser y la Thr (Figura 2.A).
En la conformación cerrada del RBD se pudo identificar la presencia de un sitio activo drogable importante también dentro de la región de trimerización, presentó un índice de drogabilidad igualmente alto con un valor de 0,80. El volumen del sitio fue de 3371,28 Å3, una superficie de 3232,56 Å2 y una profundidad de 33,12 Å. Se registró la presencia de 572 átomos, 62 grupos funcionales con capacidades de actuar como donadores en puentes de hidrógeno, 210 grupos funcionales capaces de actuar como aceptores en los puentes de hidrógeno y 114 grupos funcionales capaces de formar interacciones hidrofóbicas, la proporción hidrofóbica del sitio fue del 30%. Se encuentra conformado por 37% de residuos apolares, 44% de residuos polares, 12% de residuos básicos y 7% de residuos ácidos, hallándose en mayor proporción los residuos de Gln, Ala, Thr, Arg, Leu, Ile y Phe (Figura 2.B). Estos datos sugieren que el sitio de trimerización de la espiga viral de SARS-CoV2 es un buen blanco para dirigir contra ella la búsqueda de moléculas o fármacos con potenciales actividades inhibidoras del sitio24, esto concuerda con las observaciones de Bongini y colaboradores8 quienes describen la presencia de sitios drogables en la región de trimerización de la espiga viral del SARS-CoV2.

Figura 2 Sitio activo drogable en la región de trimerización de la glicoproteína de la espiga (S) del SARS-CoV2 (área resaltada en naranja). A. Ectodominio abierto (PDB: 6VSB). B. Ectodominio cerrado (PDB: 6VXX). Protómero A: rojo, Protómero B: blanco, Protómero C: azul.
Las pruebas de acoplamiento molecular realizadas con la conformación del ectodominio RBD abierta evidenció que la baicaleína presenta afinidades de interacción en regiones proximales del sitio de trimerización de la espiga viral, fuera del sitio drogable identificado. El complejo formado reveló un valor promedio de energía libre de unión ΔG b de -9,12±0,39 kcal.mol-1. La baicaleína posee una superficie molecular de ⁓250 Å2.molécula-1 y un volumen de ⁓223 Å3.molécula-1 alojado en regiones proximales del sitio de trimerización (Figura 3.A,B).
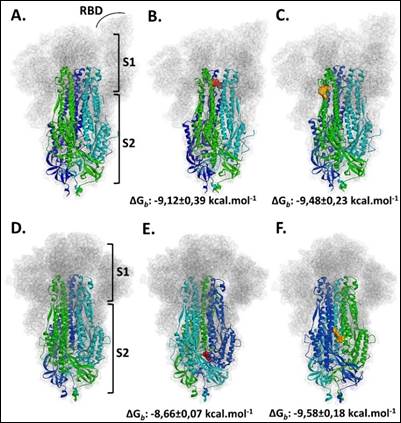
Figura 3 Sitio de trimerización de la espiga viral. (A) Conformación RBD abierta (PDB: 6VSB). Verde: Protómero A, Celeste: Protómero B, Azul: Protómero C. (B) Acoplamiento de la baicaleína (modelo rojo) con la glicoproteína de la espiga (RBD abierta). (C) Interacción de la baicalina (modelo naranja) con la espiga viral (RBD abierta). (D) Conformación RBD cerrada (PDB: 6VXX). (E) Acoplamiento de la baicaleína (modelo rojo) en la glicoproteína de la espiga (S) (RBD cerrada). (F) Interacción de la baicalina (modelo naranja) en la glicoproteína viral (RBD cerrada). ΔG b : energía libre de interacción.
La molécula baicalina no demostró afinidades por la región de trimerización sino fuera de la misma. La energía libre de interacción promedio estimada para el complejo formado fue de -9,48±0,23 kcal.mol-1 (Figura 3.A,C). Es importante destacar que en la conformación abierta del ectodominio RBD solo la baicaleína presenta una localización favorable en regiones proximales del sitio de trimerización de la glicoproteína de la espiga (protómeros A, B y C), no así la baicalina (Figura 4.A,B).
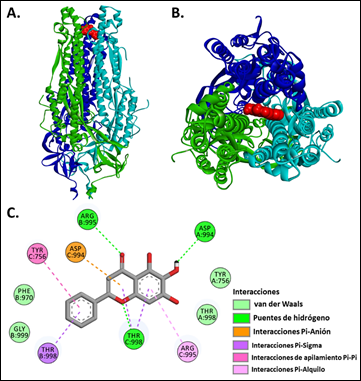
Figura 4 Acoplamiento molecular de la baicaleína en el sitio de trimerización de la glicoproteína de la espiga (S) (RBD abierto). Verde: Protómero A, Celeste: Protómero B, Azul: Protómero C. (A) Vista lateral y (B) superior del sitio de interacción de la baicaleína (modelo rojo). (C) Representación bidimensional (2D) del modelo de acoplamiento de la baicaleína. A: Protómero A, B: Protómero B, C: Protómero C.
Los residuos activos implicados en la interacción con la molécula baicaleína en el complejo de interés fueron el Asp994(A), Arg995(B), Thr998(B), Arg995(C), Thr998(C), Tyr756(C) y Asp994(C). Se registró la formación de puentes de hidrógeno entre el ligando y los residuos Asp994(A), Arg995(B) y Thr998(C) con distancias promedios de 4,25, 5,21 y 4,14 Å respectivamente entre la baicaleína y estos aminoácidos (Figura 4.C). Estos residuos se encuentran entre los reportados por Xiu y colaboradores32, quienes mencionan que los aminoácidos aa947-aa1027 interaccionan con moléculas de umifenovir evitando la trimerización de la espiga del SARS-CoV2.
También se identificó la formación de interacciones electrostáticas entre los orbitales π presentes en la baicaleína y las cargas negativas del residuo Asp994(C) con una distancia promedio de 7,21 Å. Además, se observó la formación de interacciones hidrofóbicas entre orbitales π de la baicaleína y la cadena de alquilo del residuo Arg995(C) con distancias promedio de 5,08 Å. Con el residuo Thr998(B) y Thr998(C) se registraron interacciones del entre los orbitales π y orbitales σ presentes en estos residuos y el ligando, con distancias de 4,80, 4,14 y 5,15 Å respectivamente entre baicaleína y estos residuos.
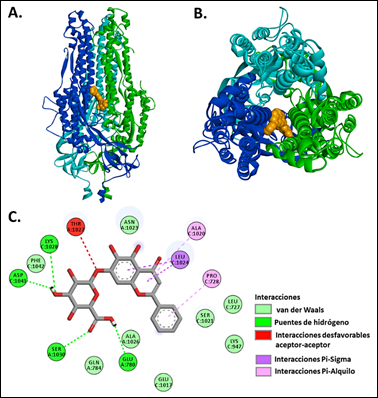
Figura 5 Acoplamiento molecular de la baicalina en el sitio de trimerización de la glicoproteína de la espiga viral (RBD cerrado). Verde: Protómero A, Celeste: Protómero B, Azul: Protómero C. (A) Vista lateral y (B) superior del sitio de interacción de la baicalina (modelo naranja). (C) Representación bidimensional (2D) del modelo de acoplamiento de la baicalina. A: Protómero A, C: Protómero C.
Así mismo se detectó la formación de interacciones generadas por apilamientos de orbitales π presentes en la baicaleína y en el residuo Tyr756(C) con una distancia promedio de 6,54 Å. El bolsillo de interacción se conforma por los residuos Tyr756(A), Thr998(A), Gly999(B) y Phe970(B) los cuales mediante interacciones de van der Waals estabilizan la localización de la baicaleína en la región de trimerización (Figura 4.C).
El acoplamiento molecular realizado entre la glicoproteína de la espiga (S) en su conformación RBD cerrada, reveló que la baicaleína no presenta afinidades de interacción por la región de trimerización, sino que tiende a localizarse favorablemente en la región subyacente a este, cercana al dominio péptido de fusión (FP), con una energía libre de interacción promedio de -8,66±0,07 kcal.mol-1.
Sin embargo, la baicalina demostró afinidades de interacción favorables por el sitio de trimerización, la energía libre de unión registrada fue de -9,58±0,18 kcal.mol-1. Esta molécula posee una superficie de ⁓386 Å2.molécula-1 y un volumen molecular de ⁓353 Å3.molécula-1 (Figura 3.D,E,F, Figura 5.A,B).
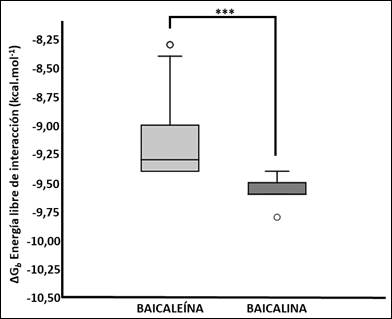
Figura 6 Valores de energía libre de interacción (∆G b ) de los compuestos en acoplamiento con el sitio de trimerización de la glicoproteína de la espiga (S). *** p<0,001.
Los residuos activos implicados en la formación de interacciones con la baicalina fueron la Ala1020(C), Leu1024(C), Pro728(C), Asp1041(C), Lys1028(C), Glu780(A) y Ser1030(A). Se registró la formación de puentes de hidrógeno entre la baicalina y los residuos Ser1030(A), Glu780(A), Asp1041(C) y Lys1028(C) con distancias promedio de 4,87, 3,19, 4,69 y 6,51 Å respectivamente entre la baicalina y estos residuos.
Se detectó la formación de interacciones hidrofóbicas entre los orbitales π de la baicalina y las cadenas de alquilo de los residuos Ala1020(C) y Pro728(C) con distancias de 6,74 y 6,82 Å, respectivamente. Con el residuo Leu1024(C) se registró la formación de interacciones entre orbitales π y orbitales σ del residuo, con una distancia de 5,60 Å entre ellos. Así también se registraron interacciones desfavorables del tipo aceptor-aceptor entre la baicalina y el residuo Thr1027(A), ocasionada posiblemente por repulsiones entre los átomos de la baicalina y del residuo33.
El bolsillo de interacción se conforma por los residuos Phe1042(C), Asn1023(A), Leu727(C), Ser1021(C), Lys947(C), Glu1017(C), Ala1026(A) y Gln784(A), los cuales favorecen a la localización de la baicalina en la región de trimerización mediante interacciones de van der Waals (Figura 5.C).
Los datos de energía libre de unión obtenidos en los complejos formados entre los ligandos (baicaleína y baicalina) y el sitio de trimerización de la glicoproteína de la espiga del SARS-CoV2, revelaron que las interacciones registradas con la baicalina demostraron ser significativamente favorables (p<0,001) en comparación a la energía ∆G b obtenida con la baicaleína, lo cual nos sugiere una mayor afinidad de interacción de la baicalina con el sitio de trimerización (Figura 6). Las diferencias en las energía libre de interacción nos permiten seleccionar u optar por moléculas que presentan un mejor modo o posicionamiento de unión en la proteínas dianas, ya que estas poseen los valores de energía de interacción más favorables (ΔG<0), y son las de mayor tiempo de interacción con las proteínas34-35.
Varias son las estrategias que la comunidad científica opta por investigar para combatir el avance de la Covid-19, una de ellas consiste en inhibir la actividad de la espiga (S) del SARS-CoV2 mediante la interacción de fármacos antivirales o de moléculas de origen vegetal con esta glicoproteína viral32,36-37.
La mayoría busca inhibir la acción del dominio RBD de la glicoproteína de la espiga (S), sin embargo, también se ha descrito la capacidad que poseen algunas moléculas de bloquear o desestabilizar la multimerización de proteínas virales38. Bongini y colaboradores8 sugieren que el sitio de trimerización de la espiga viral del SARS-CoV2 presentan regiones drogables, lo que hace que sea buen blanco para la búsqueda de moléculas que sean capaces de intervenir la formación de la estructura cuaternaria funcional de la glicoproteína, favorecido con la posibilidad de que la trimerización sea un proceso lento e indirecto, lo cual da la posibilidad de bloquear este evento y probablemente así interferir también con el ciclo replicativo del SARS-CoV2.
La baicaleína (5,6,7-trihidroxiflavona) y la baicalina (5,6-dihidroxi-7-O-glucuronido flavona) son flavonoides presentes en las raíces de la especie S. baicalensis9-10. Varios estudios en mamíferos evidenciaron que luego de la administración oral la baicaleína es metabolizado ~90%, convirtiéndose en la molécula 7-O-β-glucopiranoronósido que es un metabolito precursor de la baicalina, catalizada por la UDP-glucuronosiltransferasa en las paredes intestinales y en el hígado13,39-42. En sangre también se ha detectado la presencia de baicalina inmediatamente después de la presencia de la baicaleína en el tejido, por lo que prácticamente ambos flavonoides coexisten en el organismo animal y humano una vez ingerido o administrado, favoreciendo sus propiedades medicinales y biológicas13,39-41,42,44.
Estudios computacionales y ensayos in vitro han demostrado la eficacia de la baicaleína y baicalina actuando como inhibidores de la proteasa NS2B-NS3 del virus del Dengue (DENV), también se ha reportado como inhibidores de la replicación del DENV-245-46. Así como también se las ha reportado como potenciales inhibidores de las proteínas de superficie hemaglutininas y neuraminidasas del virus Sendai47.
Recientes estudios computacionales han demostrado que la baicalina presenta afinidades de interacción con la proteasa 3CLpro del SARS-CoV2, la estabilidad de la unión ha sido demostrada mediante análisis de dinámica molecular, teniéndose como candidata por su potencial acción inhibidora de la proteína viral22,48).
También se ha registrado la actividad de la baicaleína inhibiendo la replicación del SARS-CoV2 al igual que del virus de la estomatitis vesicular (VSV), esto mediante la inhibición de la fosforilación oxidativa afectando directamente a la replicación del virus49. Investigadores de China, han reportado que la mezcla líquida de baicalina, ácido clorogénico y forsitina puede reducir los efectos ocasionados por el SARS-CoV2, probablemente por su capacidad de disminuir la respuesta inflamatoria del organismo afectado50.
En un estudio in vitro llevado a cabo con la baicaleína y el extracto etanólico de S. baicalensis ante la infección del SARS-CoV2 a células Vero ha evidenciado que esta molécula presenta mayor eficacia de acción luego de la internalización viral siendo el principal inhibidor de la proteasa 3CLpro. Por otra parte, también se ha observado que el extracto etanólico de S. baicalensis actúa también inhibiendo la entrada viral a un bajo porcentaje de actividad (<30%)23.
Recientemente, Vankadari en el año 202051 llevando a cabo un análisis estructural evidenció que moléculas de arbidol, un fármaco antiviral de amplio espectro52-57, también presenta potenciales actividades como bloqueantes de la trimerización de la espiga viral del SARS-CoV2, donde los residuos implicados en la interacción con este fármaco fueron la Lys776, Glu780, Lys947, Glu1017, Arg1019, Ser1021, Asn1023, Leu1024 y Thr1027.
En este estudio se logró identificar que el potencial sitio de acoplamiento del flavonoide baicalina es la región de trimerización de la glicoproteína de la espiga, la cual también presenta el mismo residuo activo (Leu1024) encontrado en interacción con el arbidol, además de encontrarse los mismos tipos de residuos Glu, Ser y Lys51. Esto nos indicaría que la baicalina probablemente presente afinidades de unión en un sitio de acoplamiento semejantes al de la molécula de arbidol. La afinidad de acoplamiento encontradas con la baicalina por el sitio de trimerización, nos sugiere que esta molécula presenta aptitudes de interactuar durante el ensamblaje de los protómeros (protómeros A, B, C) para la conformación de la estructura cuaternaria funcional de la espiga viral.
La posible lentitud del proceso de ensamblaje de esta proteína viral sugerido por Bongini y colaboradores8 durante el proceso replicativo viral, podría favorecer a que ocurran estas interacciones con las moléculas analizadas, desencadenándose en un posible bloqueo del ensamblaje de la glicoproteína de la espiga (S) lo cual podría interferir con el avance del ciclo replicativo del SARS-CoV2 en las células infectadas8.
A pesar de registrarse afinidades de acoplamiento de la baicaleína con la glicoproteína de la espiga viral, este no resultó unirse en localizaciones semejantes a las descritas por Bongini y colaboradores8, sin embargo dicha región concuerda con el rango de aminoácidos que interaccionan con las moléculas de umifenovir (aa947-aa1027) descrito por Xiu y colaboradores32. La presencia de la baicaleína en estas regiones podría contribuir o favorecer la acción de la baicalina en el bloqueo del proceso de ensamblaje de la estructura cuaternaria de la espiga viral. Estos resultados sugieren que ambas moléculas (baicaleína y baicalina) podrían actuar como potenciales bloqueantes de la trimerización de la proteína S del SARS-CoV2 durante su maduración, siendo candidatas para estudios experimentales posteriores para corroborar estas predicciones.
4. CONCLUSIÓN
Los resultados de este estudio computacional sugieren que el flavonoide baicalina presenta afinidades de acoplamiento por el sitio de trimerización de la glicoproteína de la espiga (S) con valores de energía libre de interacción significativamente favorables. También se detectó que el flavonoide baicaleína presentó afinidades de interacción con valores de energía favorables en regiones proximales al sitio de trimerización de la espiga viral. La presencia de ambos flavonoides en interacción con los protómeros de esta glicoproteína durante su ensamblaje podría generar el bloqueo de su maduración en una estructura cuaternaria funcional pudiendo intervenir con el ciclo replicativo del virus.
Estos resultados contribuyen enormemente en la búsqueda de estrategias para combatir al SARS-CoV2, específicamente en la detección de moléculas de origen vegetal con perfiles de interacción con la proteína de la espiga del virus durante el ensamblaje y maduración estructural del mismo. Estas simulaciones a pesar de presentar resultados altamente predictivos, desestiman el modo y tiempo de acción de las moléculas, así como las concentraciones óptimas de actividad lo cual depende exclusivamente de ensayos experimentales.












 uBio
uBio 


