INTRODUCCIÓN
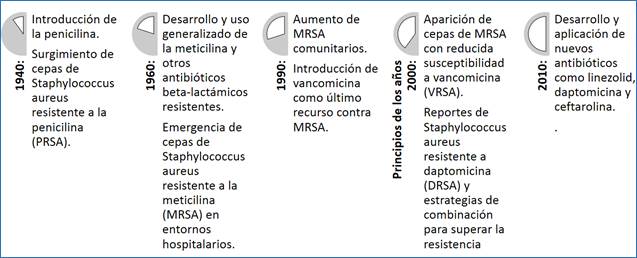
A lo largo de la historia de la medicina, S. aureus ha demostrado una notable capacidad para desarrollar resistencia a los antibióticos en respuesta a la introducción de estos fármacos 1,2. El punto de partida fue el descubrimiento de la penicilina por Alexander Fleming en 1928, marcando el comienzo de la era de los antibióticos 3-5. En la década de 1940, la penicilina se introdujo en la práctica médica y S. aureus mostró inicialmente sensibilidad a este antibiótico 6. Sin embargo, en la década de 1960 surgieron las primeras cepas resistentes a la penicilina (PRSA), lo que llevó a la introducción de la meticilina en 1961 como alternativa. Posteriormente, en las décadas de 1960 y 1970, aparecieron cepas de S. aureus resistentes a la meticilina (MRSA) 1,6.
En la década de 1980 se introdujo la vancomicina como último recurso para tratar infecciones por MRSA. Sin embargo, a medida que avanzaba el siglo XXI, surgieron cepas de S. aureus resistentes a la vancomicina (VISA) a finales de la década de 1990 y a la daptomicina (DRSA) en la década de 2000, planteando nuevos desafíos clínicos 7,8. La resistencia a estos antibióticos críticos ha evolucionado con el tiempo (VISA - finales de la década de 1990; DRSA - década de 2000) y la necesidad de estrategias terapéuticas innovadoras se ha vuelto más urgente (figura 1) 8,9. Este panorama en constante cambio subraya la complejidad y la adaptabilidad de S. aureus frente a los tratamientos antimicrobianos 10.
S. aureus desempeña un papel esencial en las enfermedades de origen bacteriano en los seres humanos, siendo esta bacteria un agente patógeno versátil capaz de causar un amplio espectro de infecciones, desde patologías cutáneas superficiales hasta enfermedades sistémicas potencialmente mortales 11,12. La resistencia de S. aureus a múltiples antibióticos, incluido la meticilina, es el resultado de una combinación de factores genéticos y adaptativos que han permitido a esta bacteria desafiar la acción de los antimicrobianos 1,13. La resistencia a la meticilina, en particular, se debe a la presencia de un gen llamado mecA, que codifica una proteína de unión a la penicilina modificada llamada PBP2a. Esta proteína tiene una afinidad reducida por los antibióticos betalactámicos, como la meticilina, lo que le permite a la bacteria sobrevivir y replicarse incluso en presencia de estos fármacos 14,15. La transferencia horizontal de genes, a menudo mediada por elementos genéticos móviles como plásmidos y transposones, ha contribuido a la diseminación de la resistencia a la meticilina entre diferentes cepas de S. aureus y a otras especies bacterianas, lo que complica aún más su control 11. Además de los factores de virulencia, S. aureus ha evolucionado para expresar una variedad de proteínas y toxinas que le permite colonizar, invadir y causar daño a los tejidos del huésped 4,16. Entre los factores de virulencia específicos se encuentran las adhesinas, que son proteínas de superficie que facilitan la unión de la bacteria a las células del huésped y a las superficies biológicas 17,18. Estas adhesinas pueden incluir la fibronectina, el fibrinógeno, la colágena y otras moléculas de la matriz extracelular 19. Al adherirse eficazmente, S. aureus puede establecer una infección y evitar ser eliminado por los mecanismos de defensa del huésped 20. Las toxinas producidas por S. aureus también son fundamentales para su capacidad patogénica 21. Las enterotoxinas, por ejemplo, pueden causar intoxicación alimentaria cuando se consumen alimentos contaminados, las leucocidinas y la toxina alfa hemolítica son capaces de dañar las membranas celulares y los glóbulos blancos, debilitando así la respuesta inmunológica del huésped y permitiendo que la infección progrese 7. Además, S. aureus produce exoenzimas, como la coagulasa y la estafilocinasa, que desempeñan un papel en la invasión y el daño de los tejidos 22. En consecuencia, puede inducir patologías que varían desde infecciones cutáneas, como furúnculos y celulitis, hasta enfermedades más graves, como neumonía, osteomielitis, endocarditis, sepsis y abscesos en órganos vitales 23-25.
El objetivo fue documentar los cambios en los patrones de resistencia de S. aureus a lo largo del tiempo, además de identificar las etapas críticas en el desarrollo de la resistencia a diferentes antibióticos
METODOLOGÍA
Se realizó una búsqueda en la literatura médica de artículos. Se comenzó con la definición del período de estudio, abarcando publicaciones desde 2010 hasta 2023 para capturar la evolución reciente en la resistencia de S. aureus. Se llevaron a cabo búsquedas exhaustivas en bases de datos clave como PubMed, Scopus, Embase, Cochrane Library y Scielo. Los criterios de inclusión fueron estudios observacionales que investigaran la resistencia antimicrobiana de S. aureus, artículos de revisión que sintetizaran el conocimiento actual sobre los mecanismos de resistencia, y guías clínicas relevantes para el manejo de infecciones por S. aureus resistente. Por otro lado, se excluyeron estudios no relacionados con S. aureus o la resistencia antimicrobiana, estudios con datos incompletos o métodos poco claros que afectaran la calidad de la información, así como artículos en idiomas distintos al inglés, español. Las estrategias de búsqueda específicas se diseñaron utilizando combinaciones de términos como "Staphylococcus aureus", "antibiotic resistance", "mechanisms", y "clinical guidelines", adaptadas a las especificidades de cada base de datos. Los artículos fueron preseleccionados mediante la revisión de títulos y resúmenes para identificar aquellos que potencialmente cumplían con los criterios de inclusión, y posteriormente se evaluaron en su totalidad para confirmar su relevancia y coherencia con los objetivos de la revisión. Se extrajeron datos pertinentes sobre patrones de resistencia, mecanismos específicos, impacto clínico y recomendaciones terapéuticas de los artículos seleccionados. Los datos recopilados fueron analizados y sintetizados para identificar tendencias temporales en la resistencia de S. aureus y las etapas críticas en el desarrollo de la resistencia a diferentes antibióticos.
RESULTADOS
Se incluyeron un total de 100 artículos originales que exploran la evolución temporal de la resistencia de S. aureus, desde las primeras cepas resistentes a la penicilina hasta las cepas actuales que muestran resistencia a la vancomicina y la daptomicina. Estos estudios proporcionaron un análisis detallado de los mecanismos moleculares clave que impulsan la resistencia antimicrobiana, tales como la producción de beta-lactamasas, las alteraciones en las proteínas de unión a penicilina y las modificaciones en la membrana celular. Los hallazgos destacan una evolución significativa en la capacidad de S. aureus para adaptarse a diferentes antibióticos a lo largo del tiempo, subrayando la complejidad y la diversidad de los mecanismos de resistencia desarrollados por esta bacteria. Esta revisión enfatiza la importancia de comprender estos mecanismos para orientar el desarrollo de estrategias terapéuticas efectivas, así como la necesidad continua de vigilancia y control de la resistencia antimicrobiana.
La exclusión de estudios no originales garantizó fiabilidad de la evidencia examinada, proporcionando una base sólida para las conclusiones y recomendaciones derivadas del estudio.
S. aureus sensible a la penicilina (PSSA)
Las cepas de S. aureus sensibles a la penicilina (PSSA) representan una parte importante de la historia epidemiológica de esta bacteria patógena 26. En un pasado no tan lejano, la penicilina era el antibiótico de elección para el tratamiento de infecciones causadas por S. aureus, las cepas eran sensibles a este fármaco y respondían favorablemente 27. No obstante, la rápida evolución de S. aureus hacia la resistencia a la penicilina, a través de la producción de una enzima llamada penicilinasa, ha reducido drásticamente la eficacia de este antibiótico en el tratamiento de las infecciones estafilocócicas 26,28. A lo largo de la década de 1950 y la década de 1960, la resistencia a la penicilina se volvió prevalente en las cepas de S. aureus, lo que llevó a la búsqueda de alternativas terapéuticas 23. Hagstrand Aldman A et al29 en un estudio retrospectivo que se enfocó en bacteriemias causadas por PSSA, comparó la eficacia de la penicilina G y la cloxacilina como tratamientos. Se evidenció que las tasas de mortalidad a los 90 días no diferían significativamente entre los grupos de tratamiento (cloxacilina: 19% vs. penicilina G: 13%). Sin embargo, los pacientes tratados con cloxacilina presentaron una mayor probabilidad de experimentar complicaciones relacionadas con el tratamiento en comparación con aquellos tratados con penicilina G, sugiriendo que, aunque las tasas de mortalidad en la bacteriemia por PSSA son comparables entre los tratamientos, la cloxacilina conlleva un mayor riesgo de complicaciones relacionadas con el tratamiento en general. Reynolds G et al27 en un estudio de cohorte retrospectivo con 90 casos de un total de 140 infecciones PSSA, del total de pacientes incluidos, 69% tenía infecciones PSSA adquiridas en la comunidad y 82% presentaba infecciones PSSA complejas, 66 pacientes recibieron tratamiento definitivo con penicilina y 24 pacientes recibieron tratamiento definitivo con beta-lactámicos antiestafilocóccicos. No se observaron diferencias significativas en la mortalidad a los 30 días. Los datos respaldan el uso de penicilina como una alternativa adecuada y bien tolerada a los beta-lactámicos antiestafilocóccicos en el manejo de infecciones complejas por PSSA en el torrente sanguíneo (tabla 1) 1,8,10.
Tabla 1 Ejemplo de antibiograma de S. aureus sensible a la penicilina (PSSA)
| Antibióticos | Concentración (ug/mL) | Sensibilidad |
|---|---|---|
| Penicilina | < 0,03 | Sensible |
| Ceftriaxona | < 0,5 | Sensible |
| Azitromicina | < 0,5 | Sensible |
| Ciprofloxacina | < 1,0 | Sensible |
| Vancomicina | < 2,0 | Sensible |
| Gentamicina | < 4,0 | Sensible |
| Trimetoprim/sulfametoxazol | < 1,0/19,0 | Sensible |
| Meropenem | < 0,5 | Sensible |
| Tetraciclina | < 4,0 | Sensible |
Elaborado por los autores
S. aureus sensible a la meticilina (MSSA)
S. aureus sensible a la meticilina (MSSA) representa una cepa bacteriana susceptible a los efectos de la meticilina y otros antibióticos de la clase de las penicilinas. A diferencia de las cepas resistentes a la meticilina (MRSA), MSSA exhibe sensibilidad a estos tratamientos, facilitando opciones terapéuticas más amplias. A pesar de esta susceptibilidad, MSSA sigue siendo una consideración significativa en el panorama de las infecciones bacterianas, ya que puede causar una variedad de enfermedades. Comprender la dinámica y la gestión de MSSA es esencial para abordar eficazmente las infecciones asociadas y garantizar la elección adecuada de antibióticos para un tratamiento exitoso.
Penicilinasas de espectro estrecho BLAz
El MSSA es una cepa de esta bacteria que se caracteriza por su susceptibilidad a ciertos antibióticos, en particular la oxacilina y la cefazolina 30,31. Posee unas β-lactamasas de espectro estrecho (BLAz), las cuales constituyen una clase de enzimas cruciales en el mecanismo de resistencia de S. aureus a los antibióticos β-lactámicos de espectro estrecho, como la penicilina G y la ampicilina 1,23. Estas enzimas presentan una estructura molecular que les permite hidrolizar de manera específica el anillo β-lactámico de los antibióticos, lo que resulta en la inactivación de estos 11. El mecanismo molecular subyacente a su acción implica la escisión de la unión β-lactámica, llevando a la desactivación de estos antibióticos 32. La relevancia clínica de las BLAz radica en su capacidad para conferir resistencia a antibióticos de espectro estrecho, disminuyendo así la eficacia de estos fármacos en el tratamiento de infecciones por S. aureus33,34. En consecuencia, las cepas bacterianas que expresan estas enzimas son resistentes a antibióticos como la penicilina G y la ampicilina, lo que limita las opciones terapéuticas para controlar las infecciones 32,35. La adquisición de genes que codifican BLAz a través de transferencia horizontal de material genético ha contribuido al aumento de la resistencia a estos antibióticos en S. aureus. Las BLAz representan una pieza fundamental en la resistencia de S. aureus a los antibióticos β-lactámicos de espectro estrecho, actuando a nivel molecular mediante la hidrólisis del anillo β-lactámico 36,37.
En el enfoque clínico de MSSA, el tratamiento de las infecciones generalmente consiste en el uso de un agente β-lactámico como nafcilina, oxacilina o cefazolina 38-40. En el tratamiento ambulatorio, cefazolina puede ser una opción más práctica y mejor tolerada que las penicilinas antiestafilocóccicas 40,41. Aunque no hay evidencia definitiva de superioridad entre cefazolina y las penicilinas antiestafilocóccicas, algunos estudios sugieren que cefazolina podría tener un perfil de riesgo-beneficio favorable. Li J et al40 realizaron un estudio retrospectivo que compara cefazolina con oxacilina en el tratamiento de bacteriemias complicadas por MSSA. Se observó que las tasas de curación clínica al final del tratamiento fueron similares entre ambos grupos (95% versus 88%). Sin embargo, la tasa de fracaso general a los 90 días fue mayor en el grupo de oxacilina (47% versus 24%), se observó tasas más altas de eventos adversos con oxacilina, lo que llevó a la interrupción del tratamiento en más casos en comparación con cefazolina. Bidell M et al42) en un metaanálisis, incluyeron 1589 pacientes que recibieron cefazolina y 2802 con una penicilina antiestafilocóccica. La tasa de mortalidad a los 90 días por cualquier causa fue significativamente menor en los pacientes que recibieron cefazolina (OR 0,63 ; IC 95% 0,41-0,99; I2 = 58%). La probabilidad de interrupción del tratamiento debido a eventos adversos fue significativamente menor en los pacientes que recibieron cefazolina (OR 0,25, IC 95% 0,11-0,56; I2 = 13%). No se observaron diferencias en los fallos clínicos (OR 0,85 ; IC 95% 0,41-1,76; I2 = 74), encontrándose una disminución significativa en la mortalidad asociada con la terapia de cefazolina para bacteriemias por MSSA en comparación con las penicilinas antiestafilocóccicas, aunque no se observaron diferencias en los fallos clínicos.
En casos de alergia a los β-lactámicos, la vancomicina se utiliza como alternativa. Sin embargo, la vancomicina es menos efectiva que los agentes β-lactámicos para tratar la bacteriemia por MSSA y se reserva para casos en los que no se pueden usar β-lactámicos debido a intolerancia. Los pacientes con bacteriemia por MSSA y alergia a la penicilina deben recibir tratamiento basado en la historia de alergia o, si es posible, en una evaluación completa de alergia con pruebas cutáneas o desafío oral (tabla 2).
Tabla 2 Ejemplo de antibiograma de S. aureus sensible a la meticilina (MSSA)
| Antibióticos | Concentración (ug/mL) | Sensibilidad |
|---|---|---|
| Penicilina | < 32 | Resistente |
| Clindamicina | < 0,25 | Sensible |
| Eritromicina | < 0,25 | Sensible |
| Ciprofloxacina | < 0,25 | Sensible |
| Oxacilina | < 0,25 | Sensible |
| Gentamicina | < 2,0 | Sensible |
| Trimetoprim/sulfametoxazol | < 1,0/19,0 | Sensible |
| Cefoxitina | < 0,5 | Sensible |
| Tetraciclina | < 2,0 | Sensible |
Elaborado por los autores
Efecto inóculo en MSSA
El efecto inóculo en MSSA se refiere a un fenómeno en el que la respuesta a ciertos antibióticos, en particular la cefazolina, puede variar según la cantidad de bacterias presentes en una muestra 43. En otras palabras, a medida que aumenta la concentración de MSSA en una infección, la eficacia de la cefazolina puede verse comprometida, lo que significa que se necesita una mayor cantidad de este antibiótico para inhibir el crecimiento bacteriano 44. Este fenómeno se ha asociado con la expresión de genes específicos en MSSA, como los que codifican para proteínas de unión a la penicilina (PBPs), que son esenciales para la síntesis de la pared celular bacteriana 7,45. Los niveles más altos de MSSA en una infección pueden llevar a una mayor expresión de PBPs, lo que puede requerir una concentración más alta de cefazolina para bloquear eficazmente estas proteínas y, por lo tanto, inhibir el crecimiento de las bacterias 9,46. La importancia de comprender este efecto inóculo radica en la necesidad de ajustar adecuadamente las dosis de antibióticos, especialmente en infecciones graves o de alto inóculo por MSSA 47,48. No tener en cuenta este efecto podría resultar en una terapia ineficaz y contribuir al desarrollo de resistencia antibiótica 49,50. El gen blaZ es aquel que codifica para la producción de una enzima llamada beta-lactamasa, que es capaz de desactivar ciertos antibióticos beta-lactámicos como las penicilinas 43. En el contexto de MSSA, la variabilidad en los tipos de genes blaZ es importante, ya que algunos tipos pueden influir en la resistencia a ciertos antibióticos 7. En los estudios se examinaron los tipos de genes blaZ presentes en aislados clínicos de MSSA. Los resultados mostraron diferentes tipos de genes blaZ, identificados como tipos A, B, C y D. Cada uno de estos tipos puede tener implicaciones en la resistencia a los antibióticos beta-lactámicos, especialmente aquellos que son susceptibles a la acción de las beta-lactamasas. Se observó que el tipo C de blaZ estaba presente en una proporción significativa de aislados de MSSA y que estos aislados mostraron un efecto inóculo más marcado en respuesta a ciertos antibióticos, como ampicilina/sulbactam y piperacilina/tazobactam 44. Esto sugiere que la presencia de ciertos tipos de genes blaZ en MSSA puede estar asociada con un mayor riesgo de resistencia a ciertos antibióticos beta-lactámicos, lo que tiene implicaciones importantes para la elección de tratamientos efectivos en infecciones por MSSA 7.
S. aureus resistente a la meticilina (MRSA)
S. aureus resistente a la meticilina (MRSA) es una cepa que ha desarrollado resistencia a los antibióticos de la familia de las penicilinas, incluyendo la meticilina y la oxacilina. Esta resistencia a los antibióticos hace que MRSA sea una amenaza significativa en el campo de la salud, ya que es más difícil de tratar y puede causar infecciones graves y potencialmente mortales 51,52.
La resistencia a la meticilina en MRSA es un fenómeno que se debe a una serie de mecanismos moleculares sofisticados que S. aureus ha desarrollado a lo largo del tiempo y que contribuyen a la resistencia a la meticilina en MRSA 53,54.
Producción de PBP2a: la resistencia a la meticilina
La resistencia a la meticilina en MRSA se debe principalmente a la producción de una proteína llamada PBP2a (proteína fijadora de penicilina 2a), que es una proteína de unión a la penicilina alterada 53,55,56. A diferencia de las proteínas de unión a la penicilina normales, PBP2a tiene una baja afinidad por los antibióticos betalactámicos como la meticilina, lo que le permite continuar construyendo la pared celular bacteriana incluso en presencia de estos antibióticos 23,57. La producción de PBP2a en MRSA se debe a la presencia del gen mecA, que codifica la síntesis de PBP2a 56,58. El gen mecA se encuentra en un elemento genético móvil conocido como el cassette cromosómico estafilocócico (SCCmec) 59-61. Este elemento genético puede transferirse entre diferentes cepas de S. aureus, lo que contribuye a la propagación de la resistencia a la meticilina 59.
Regulación de la expresión genética
La expresión del gen mecA está regulada por varios factores, incluidos reguladores de dos componentes como GraRS y WalKR 19,62,63. Estos reguladores controlan la producción de PBP2a y, por lo tanto, influyen en la resistencia a la meticilina 59. Cualquier alteración en estos sistemas de regulación puede llevar a un aumento en la resistencia, además del gen mecA, pueden ocurrir mutaciones en otros genes que también contribuyen a la resistencia a la meticilina 64. Estas mutaciones pueden afectar la expresión de las proteínas de unión a la penicilina normales y aumentar la capacidad de MRSA para resistir los antibióticos 63. La comprensión de estos mecanismos moleculares es esencial para desarrollar estrategias de tratamiento y control eficaces contra MRSA. Dado que la resistencia a la meticilina es un problema de salud pública significativo, la investigación continua se centra en identificar nuevas dianas terapéuticas y en el desarrollo de antibióticos alternativos que puedan combatir con éxito esta cepa de S. aureus51.
MRSA tiene una susceptibilidad a la vancomicina o la daptomicina siendo los agentes de elección para el tratamiento de infecciones invasivas por MRSA 52. Los agentes alternativos que se pueden usar para terapia de segunda línea o de rescate incluyen la telavancina, la ceftarolina y la linezolid 65.
Linezolid, daptomicina, telavancina y ceftarolina son medicamentos que han recibido aprobación regulatoria en las últimas décadas para el tratamiento de infecciones causadas por MRSA 24. Aunque estos medicamentos tienen ciertos atributos diferenciadores y pueden ofrecer algunas ventajas sobre la vancomicina, también tienen limitaciones significativas. Más importante aún, los datos de ensayos clínicos aleatorizados que respalden una mayor eficacia terapéutica de los nuevos agentes en comparación con la vancomicina en el tratamiento de infecciones graves por MRSA son limitados 52.
Cefoxitin como marcador de resistencia de MRSA
La resistencia a la meticilina en S. aureus está principalmente mediada por la presencia de la proteína fijadora de penicilina 2a, codificada por el gen mecA 64. En ciertas cepas de MRSA, el gen mecA se expresa de manera heterogénea in vitro. Históricamente, estas cepas han sido difíciles de detectar y se han utilizado medios selectivos para facilitar la recuperación de la subpoblación resistente en cultivos 66,67. Para aumentar la sensibilidad en la detección de cepas de MRSA con resistencia heterogénea se ha utilizado a cefoxitin como antibiótico de marcador de resistencia 66. Fernandes CJ et al67) evaluó el uso de cefoxitin como marcador sustituto para la detección de resistencia a la meticilina en cepas de S. aureus, la cual resultó altamente preciso, con una sensibilidad y especificidad del 100% tanto en las pruebas de difusión en disco como en la dilución en agar, permitiendo una clara distinción entre cepas resistentes y susceptibles a la meticilina, demostrando su utilidad en la detección de resistencia a la meticilina. Otro estudio comparó cuatro métodos fenotípicos convencionales con la PCR basada en el gen mec-A para la identificación de MRSA. Se encontró que el método de difusión con cefoxitin mostró una especificidad del 100%, lo que convierte en un método rápido, simple y económico, lo que sugiere que puede utilizarse rutinariamente como alternativa a la PCR para la detección de MRSA en laboratorios con limitaciones de recursos 3 (tabla 3).
Tabla 3 Ejemplo de antibiograma de S. aureus resistente a la meticilina (MSSA)
| Antibiótico | Concentración (ug/mL) | Sensibilidad |
|---|---|---|
| Clindamicina | < 0,25 | Sensible |
| Eritromicina | < 0,25 | Sensible |
| Ciprofloxacina | < 0,25 | Sensible |
| Oxacilina | > 4 | Resistente |
| Gentamicina | < 2,0 | Sensible |
| Trimetoprim/sulfametoxazol | < 1,0/19,0 | Sensible |
| Cefoxitin | > 4 | Resistente |
| Tetraciclina | < 2,0 | Sensible |
Elaborado por los autores
S. aureus intermedio a la vancomicina (VISA)
S. aureus intermedio a la vancomicina (VISA) destaca por su resistencia intermedia a la vancomicina. Esta resistencia es un fenómeno que plantea serias preocupaciones en la comunidad médica y científica, ya que limita aún más las opciones de tratamiento para las infecciones por S. aureus68-70. VISA presenta un nivel de resistencia significativo, lo que implica que se requieren dosis más altas de vancomicina para combatir la infección de manera efectiva 69,71. La identificación temprana de VISA es esencial para adaptar el tratamiento y evitar que las infecciones se vuelvan más graves y difíciles de tratar 68,72.
Una característica importante de VISA es su capacidad para modificar la densidad del peptidoglicano en la pared celular bacteriana, lo que dificulta que la vancomicina penetre y cumpla su función de inhibir la síntesis de la pared celular 71,73. Además, algunas cepas de VISA han adquirido genes específicos de resistencia, como vanA y vanB, que les permiten modificar los precursores del peptidoglicano, disminuyendo su susceptibilidad a la vancomicina 74,75.
La patogenia de VISA se basa en gran medida en su capacidad para evadir la acción de la vancomicina. Para comprender cómo VISA ha desarrollado esta resistencia, es fundamental examinar los mecanismos moleculares involucrados.
Mecanismos moleculares involucrados en el desarrollo de resistencia en VISA.
Modificación de las paredes celulares: VISA presenta cambios en la estructura de su pared celular, las bacterias han desarrollado una mayor densidad de peptidoglicano, que es una parte esencial de la pared celular 76. Esta densidad adicional dificulta que la vancomicina penetre y se una a sus objetivos en la pared celular 75.
Adquisición de genes de resistencia: algunas cepas aisladas de VISA han adquirido genes específicos de resistencia a la vancomicina. Los genes vanA, vanB, vanC, vanD y vanE son ejemplos de genes que codifican enzimas que modifican los precursores del peptidoglicano y hacen que sean menos susceptibles a la vancomicina 68,74,75.
Mutaciones en Dlt: Las mutaciones en el gen Dlt pueden llevar a la modificación de la carga superficial de las bacterias, disminuyendo la afinidad de la vancomicina por la membrana bacteriana 77-79.
Mutaciones en Agr: la regulación de la Agr (regulón de la respuesta a la Agr) se ha asociado con la virulencia de S. aureus80. Algunas mutaciones en el regulón Agr han sido implicadas en la resistencia a la vancomicina, aunque la relación exacta entre la Agr y la resistencia a la vancomicina aún se comprende parcialmente 81,82. Dai Y et al83 analizaron cepas clínicas de S. aureus con resistencia heterogénea a la vancomicina (hVISA), junto con cepas de referencia hVISA y VISA, y encontraron que el aumento en la concentración inhibitoria mínima (MIC) de la vancomicina se asoció con una mayor expresión del gen regulador asociado a la resistencia a la vancomicina (vraR) y una disminución en la expresión de genes de virulencia. Los resultados sugieren que VraR puede tener un efecto directo o indirecto en la virulencia de S. aureus, al inhibir la función del sistema de percepción de quórum Agr.
Adaptación a la presión de selección: la resistencia a la vancomicina puede surgir gradualmente cuando las bacterias son sometidas a una presión de selección, como la exposición repetida al antibiótico 80,84. Durante este proceso de adaptación, las bacterias con mutaciones favorables que les confieren resistencia a la vancomicina tienen ventaja en la supervivencia y proliferación 18,85. La adaptación a la vancomicina ha sido estudiada tanto en experimentos de laboratorio adaptativos como en entornos clínicos, con cepas recuperadas de pacientes antes y después del tratamiento 60. Los resultados muestran una superposición significativa en los cambios genéticos observados in vivo e in vitro, afectando procesos celulares importantes como la transcripción, el metabolismo y la biosíntesis de la pared celular 83. Se destacan sistemas de transducción de señales de dos y tres componentes, incluyendo VraT/VraSR, GraSR, y el esencial sistema WalKR, como especialmente afectados por las mutaciones 84.
Es importante destacar que VISA no es completamente resistente a la vancomicina, pero tiene una susceptibilidad reducida. Esto significa que, a menudo, se requieren dosis más altas de este antibiótico para tratar con éxito las infecciones por VISA. También puede requerir combinaciones de antibióticos u otras terapias 83.
S. aureus resistente a la vancomicina (VRSA)
La aparición de cepas de VRSA ha generado preocupación significativa en el ámbito de la salud pública. La vancomicina, un antibiótico de referencia para tratar infecciones por MRSA, ha sido desafiada por la evolución de cepas que han adquirido resistencia a este. El VRSA exhibe una capacidad única para resistir los efectos de la vancomicina 86. Los mecanismos de resistencia implican modificaciones en el peptidoglicano de la pared celular, específicamente a través de genes como vanA, vanB o vanC, que facilitan la resistencia a la acción antibiótica de la vancomicina 87. La emergencia de VRSA destaca la necesidad crítica de estrategias de control de infecciones, desarrollo de nuevos agentes antimicrobianos y prácticas de uso prudente de antibióticos para prevenir la propagación y evolución de estas cepas resistentes 88. La comprensión profunda de estos mecanismos de resistencia es esencial para abordar este desafío en constante evolución en la lucha contra las infecciones bacterianas 89.
El desarrollo de VRSA está vinculado a la transferencia horizontal de genes de resistencia. A menudo, este proceso involucra la adquisición del elemento genético vanA, vanB o vanC, que codifican para la síntesis de precursores de peptidoglicano con baja afinidad por la vancomicina 90.
Mecanismos moleculares de resistencia
Genes vanA, vanB, vanC: estos genes codifican enzimas que modifican la estructura del peptidoglicano, confiriendo resistencia a la vancomicina. El gen vanA es el más comúnmente asociado con la resistencia a la vancomicina en estafilococos 91,92.
Transferencia horizontal: el intercambio de material genético entre bacterias facilita la propagación de los genes de resistencia. La transferencia de plásmidos o elementos genéticos móviles juega un papel clave en la diseminación de resistencia entre cepas bacterianas 93.
Adaptación evolutiva: la exposición continua a la presión selectiva de la vancomicina favorece la evolución de cepas resistentes 80. La variabilidad genética intrínseca de S. aureus permite la selección de mutantes con resistencia mejorada 14,80.
Adquisición de resistencia de S. aureus a la vancomicina
La adquisición de resistencia a la vancomicina suele ocurrir en un contexto clínico, donde los pacientes con infecciones persistentes por MRSA son tratados repetidamente con vancomicina. La presión constante del antibiótico favorece la selección de cepas resistentes. Además, la coexistencia de Enterococcus resistentes a la vancomicina en el mismo entorno clínico puede facilitar la transferencia de genes de resistencia a S. aureus88,94.
La emergencia de VRSA subraya la importancia de estrategias rigurosas de control de infecciones y el desarrollo constante de nuevos agentes antimicrobianos.
S. aureus resistente a la daptomicina (DRSA)
El DRSA está delineado por su capacidad para resistir los efectos del vital antibiótico daptomicina y se erige como un desafío particularmente complejo 95. A nivel molecular, las mutaciones en genes esenciales como mprF y cls orquestan alteraciones en la membrana celular, erigiendo una barrera que obstaculiza la acción de la daptomicina 96. Este fenómeno despierta preocupación en entornos clínicos globales. La presencia del DRSA tanto en ambientes hospitalarios como en la comunidad en general ha desatado la necesidad crítica de estrategias terapéuticas innovadoras debido a la complejidad de S. aureus resistente a daptomicina, incitando a una inmersión profunda en su genética, su impacto clínico y la investigación para encontrar soluciones eficaces frente a esta entidad patológica desafiante 20,97.
Genes involucrados en la resistencia de DRSA
La resistencia a daptomicina implica genes diversos, como mprF, que codifica una proteína de transporte de fosfatidilglicerol, y cls, que orquesta la síntesis de cardiolipina, una clase crucial de lípidos de membrana 12. Mutaciones en estos genes alteran la carga eléctrica y la composición lipídica, obstaculizando la acción de la daptomicina, además de las mutaciones en los genes yycG y yycH, componentes de los sistemas que regulan la biosíntesis de la membrana y contribuyen a la resistencia 98. Estas alteraciones desencadenan cambios en la estructura y carga de la membrana, dificultando la penetración de la daptomicina 96. En un estudio de mutantes de S. aureus con mayor resistencia se identificaron inserciones de transposones en S. aureus HG003. Dos genes, Dsp1 y Asp23, se destacaron por su papel en la resistencia. Mutantes y cepas de pérdida de función mostraron resistencia incrementada a daptomicina y péptidos antimicrobianos 99. Estos hallazgos revelan la contribución de genes fundamentales en la resistencia y tolerancia de S. aureus, ofreciendo perspectivas cruciales para abordar estas infecciones 98,100.
El DRSA ha emergido en diversos contextos, subrayando la necesidad de estrategias terapéuticas innovadoras. Combinaciones de antibióticos, como la daptomicina con el antibiótico β-lactámico ceftarolina, han mostrado eficacia 50. Barber KE et al16) evaluó en un estudio in vitro la eficacia de ceftarolina sola o combinada con daptomicina o rifampicina contra cepas de S. aureus resistentes a meticilina y daptomicina. Ceftarolina mostró actividad constante, y la combinación de ceftarolina más daptomicina exhibió una potente actividad, ofreciendo una opción prometedora para infecciones asociadas a dispositivos médicos difíciles de tratar (tabla 4).
Tabla 4 Clasificación de S. aureus según su patrón de resistencia con sus principales genes implicados.
| S. aureus | Resistencia y mecanismos | Genes asociados |
| PSSA (sensible a la penicilina) | Sensible a penicilina y betalactámicos | Ausencia de genes de resistencia |
| SAMS (sensible a la meticilina) | Sensible a meticilina y betalactámicos | Ausencia de genes de resistencia |
| MRSA (resistente a la meticilina) | Resistencia a betalactámicos, cambio de proteína PBP | mecA, mecB, mecC |
| VISA (intermedio a la vancomicina) | Resistencia intermedia a vancomicina, modificación de peptidoglicano | Genes de baja especificidad |
| VRSA (resistente a la vancomicina) | Resistencia a la vancomicina, engrosamiento de pared celular | vanA, vanB, vanC |
| DRSA (resistente a la daptomicina) | Resistencia a la daptomicina | Genes de resistencia específicos |
Elaborado por los autores
Esta revisión aborda de manera exhaustiva el desarrollo histórico y los complejos mecanismos de resistencia de S. aureus a lo largo de la era de los antibióticos, desde las primeras cepas resistentes a la penicilina hasta las actuales que desafían antibióticos de último recurso como la vancomicina y la daptomicina. Esto permite identificar patrones evolutivos y emergentes en la resistencia bacteriana, proporcionando una comprensión profunda de los mecanismos moleculares y genéticos involucrados. Sin embargo, las limitaciones potenciales incluyen la variabilidad en la calidad y metodología de los estudios revisados, así como posibles sesgos de publicación que podrían sesgar los resultados.
La importancia de esta revisión radica en su capacidad para informar políticas de salud pública y guiar prácticas clínicas efectivas en el manejo de infecciones por S. aureus resistente a antibióticos. Destaca la urgencia de desarrollar estrategias de control de infecciones más efectivas y de promover la investigación continua para abordar esta persistente amenaza en la era moderna de los antibióticos.
Basado en los hallazgos de la revisión, se recomienda enfocarse en varias áreas clave para abordar la resistencia de S. aureus de manera efectiva. Es fundamental fortalecer la vigilancia epidemiológica para detectar tempranamente nuevas cepas resistentes y patrones emergentes. Además, es crucial implementar políticas y programas que promuevan el uso racional de antibióticos tanto en entornos clínicos como comunitarios, reduciendo así la presión selectiva que impulsa el desarrollo de resistencia bacteriana. Además, fomentar la investigación y desarrollo de nuevos agentes antimicrobianos y terapias alternativas para combatir eficazmente las cepas resistentes de S. aureus. Asimismo, se deben implementar y reforzar medidas efectivas de control de infecciones en hospitales y centros de atención sanitaria para reducir la transmisión de bacterias resistentes entre pacientes y personal de salud, todo lo anterior juega un papel decisivo en mitigar la amenaza que representa S. aureus resistente para la salud pública global.
En conclusión, la evolución de la resistencia de S. aureus ha seguido un patrón marcado por etapas críticas, desde la aparición de cepas productoras de penicilinasa tras la introducción de la penicilina, hasta el surgimiento de MRSA con la meticilina y de VISA y VRSA con la vancomicina. Estos cambios destacan la capacidad de adaptación de S. aureus a nuevas presiones antibióticas. La revisión subraya la necesidad urgente de desarrollar estrategias antimicrobianas innovadoras y sostenibles para controlar esta creciente amenaza. Comprender los mecanismos de resistencia es crucial para desarrollar enfoques más efectivos y personalizados en el tratamiento de las infecciones por este germen.