Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Investigación Agraria
On-line version ISSN 2305-0683
Investig. Agrar. vol.17 no.2 San Lorenzo Dec. 2015
https://doi.org/10.18004/investig.agrar.2015.diciembre.108-115
ARTÍCULO CIENTÍFICO
Perfil de expressão do gene fla 14 em tegumento de sementes de soja
Differential Expression of the gene fla 14 in soybean seed tegument
Daisy Ramirez Monzón1*, Caroline Borges Bevilacqua2, Eduardo Venske1, Luciano Carlos Da Maia1 e Paulo Dejalma Zimmer1*
1 Universidade Federal de Pelotas UFPel, Faculdade de Agronomia Eliseu Maciel, Pelotas, Brasil.
2 Universidade Federal de Santa Maria, UFSM, Brasil.
* Autor para correspondencia (daisyrami@gmail.com)
Recibido: 25/08/2015; Aceptado: 20/11/2015.
RESUMO
O tegumento apresenta uma função importante quanto à resistência das sementes e a sua deterioração. Sendo assim, torna-se necessário entender os mecanismos de genes responsáveis por caracteres que possam auxiliar no desenvolvimento de variedades com melhor qualidade. Nesse sentido, a análise da expressão do gene fla 14 que codifica para a proteína Arabinogalactana que esta relacionada diretamente com a síntese da parede celular no tegumento da semente de soja pode contribuir para o avanço dos estudos. Sendo assim, o objetivo do trabalho foi validar primers e analisar o perfil da expressão do gene fla 14 em quatro genótipos de soja, dois desses com tegumento preto (TP e IAC 222) e maior conteúdo de lignina, e outros dois com tegumento amarelo (CD 202 e Potência) e menor conteúdo de lignina. O tecido vegetal (tegumento) foi coletado aos 50 dias após a antese. Para a análise da expressão gênica dos genótipos, utilizou-se a técnica de qRT-PCR, onde foram testadas quatro combinações de primers construídos a partir de modelos de sequência de ESTs da soja. Os resultados da validação demostraram a eficiência dos primers FLA 14 p1 e FLA 14 p2para serem utilizados em estudos de expressão gênica. Em relação ao gene fla 14, pode-se observar queo perfil de expressão deste gene é quantitativamente superior nos genótipos de soja com tegumento preto e conteúdo superior de lignina quando relacionado com os genótipos com tegumento amarelo e menor teor de lignina.
Palavras-chave: Glycine max L. Marr; qRT-PCR; tegumento preto; Arabinogalactan.
ABSTRACT
The seed coat has an important role related to seed resistance and deterioration. Therefore, it becomes necessary to understand the mechanisms of genes responsible for traits which can assist developing cultivars with better seed quality. In this sense, the analysis of the expression of fla 14 gene, encoding for the Arabinogalactan protein, which is directly related to cell wall synthesis in soybean seed coat, can contribute to the advancement of this study. Thus, the aim of this work was to validate primers and analyze fla 14 gene expression profile in four soybean genotypes, two of these with black seed coat (TP and IAC 222) and higher lignin content, and two others with yellow seed coat (CD 202 and Potência) and lower lignin content. The plant tissue (seed coat) was collected 50 days after anthesis. qRT-PCR technique was used for genotypes gene expression analysis, through which four combinations of primersconstructed from soybean EST sequence models were tested. The validation results demonstrated the efficiency of FLA 14 p1 and FLA 14 p2 primers in gene expression studies. Regarding fla 14 gene, it was possible to notice that the expression profile of this gene is quantitatively superior in soybean genotypes with black coat and higher lignin content when comparing it with genotypes with yellow coat and lower lignin content.
Key words: Glycine max L. Marr; qRT-PCR; black seed coat; Arabinogalactan.
INTRODUÇÃO
A soja é uma das principais culturas produzidas no Brasil, apresentando um aumento na produção de 10,7% (30.135 milhões de hectares) no último ano (CONAB 2014), motivo pelo qual, esta cultura tem sido alvo de muitos estudos ao longo do tempo, com a finalidade de entender os processos que interferem na sua produção e cultivo, principalmente em ambientes desfavoráveis (Popp et al. 2003).
O aumento nos ganhos produtivos, proporcionados pelo alto desenvolvimento tecnológico, é dado pelo melhoramento genético além de ciências associadas, aumentando o avanço da tecnologia e proporcionando melhoras tanto fenotípicamente como genotípicamente (Muniz 2007). Com a estabilização da área de cultivo verificou-se um aumento na produtividade devido à utilização dessas novas tecnologias e ao desenvolvimento de cultivares adaptadas às diversas regiões produtoras (Mertz 2010).
As sementes possuem um rol fundamental como agente transferidor de tecnologia, sendo que entre os fatores para a obtenção de altas produtividades de soja encontra-se a utilização de sementes de qualidade. Para que os esforços realizados na obtenção de uma variedade sejam traduzidos em benefícios para a produção agrícola é fundamental a utilização de sementes de alta qualidade (Shuster 2003). Sendo que a qualidade da semente é caracterizada pela somatória dos atributos genéticos, físicos, fisiológicos e sanitários, características que irão determinar o desempenho da semente quando semeada ou armazenada (Botelho 2012).
Portanto, é fundamental a utilização de sementes de alta qualidade fisiológica, pois ela será responsável pela transferência de inovações tecnológicas e dos ganhos genéticos resultantes dos trabalhos de melhoramento, principais fatores na determinação do sucesso de uma cultura (Vieira et al. 1998). Na soja, esses atributos são, em grande parte, reflexos das funções do tegumento da semente (Silva 2003).
O tegumento apresenta funções importantes que estão relacionadas a resistência e deterioração das sementes (Dassou e Kueneman 1974), exercendo funções de proteção do eixo embrionário e do tecido de reserva (Carvalho e Nakagawa 2000); além de protegê-los de rupturas celulares e perda de substâncias intracelulares durante a embebição (Duke e Kakefuda 1981). Desta forma, as características do tegumento tais como as que definem o nível de permeabilidade à água, assim como a cor e o teor de lignina, podem contribuir para a geração de genótipos de sementes com elevado potencial fisiológico.
As sementes de soja com maior conteúdo de lignina apresentam menores índices de danificação mecânica (Capeleti et al. 2005). Sendo que, o teor porcentual da lignina nos tegumentos de sementes de coloração escura é de 12,18%, no entanto que o teor percentual nos tegumentos amarelo apresenta 4,75% (França Neto et al. 1999).
A semente da soja apresenta variações de tonalidades na coloração do tegumento, característica esta que é controlada por vários genes (Carvalho e Nakagawa 2000, Mertz et al. 2009). As sementes com tegumento preto são linhagens não melhoradas, apesar de apresentarem o tegumento semi-permeável, não são utilizadas comercialmente, pois estas características não estão presentes nas cultivares modernas (Mertz et al. 2009).
Às sementes que apresentam tegumento amarelo podem atribuir-se características de permeabilidade e são considerados mais suscetíveis a deterioração. Danos causados principalmente pelas flutuações na umidade, maior taxa de danos mecânicos, desenvolvendo rachaduras durante a embebição, são uns dos problemas observados na soja com tegumento amarelo, quando comparados com sementes de soja com tegumento semi-permeável (França Neto e Potts 1979, Ma et al. 2004, Mertz et al. 2009).
Sequenciando fragmentos de cDNA para comparar a expressão diferencial de genes nos genótipos de soja com tegumento preto, foi possível identificar uma proteína correspondente a Arabinogalactana (FLA 14), proteína que, segundo Yang et al. (2008), estaria envolvida na regeneração da parede celular.
Igualmente, Friedmann et al. (2007), sugere que esta proteína tem um papel fundamental durante a formação da parede celular secundária, por ser parte do complexo da celulose sintase, além de estar envolvida na modificação das microfibras de células, processo este desenvolvido tanto nas angiospermas quanto nas gimnospermas.
Sendo assim, o objetivo deste estudo foi validar primers e analisar o perfil da expressão do gene fla 14 em quatro genótipos de soja, dois desses com tegumento preto (TP e IAC 222) e maior conteúdo de lignina, e outros dois com tegumento amarelo (CD 202 e Potência) e menor conteúdo de lignina.
MATERIAL E MÉTODOS
O experimento foi conduzido na casa de vegetação da Estação Terras Baixas da Embrapa Clima Temperado (CPACT) em Pelotas/RS, durante o ano agrícola 2010/12. Os genótipos utilizados foram CD 202 e BMX Potência RR, caracterizados por apresentarem tegumento amarelo, permeável e considerados suscetíveis à deterioração, e os genótipos IAC 222 e Tegumento Preto (TP), ambos de tegumento preto, semi-permeável e mais resistentes à deterioração (Mertz 2007).
A semeadura foi feita em condições homogêneas em baldes plásticos de 20 L contendo solo, conforme a recomendação da análise de solo foi adicionado adubo químico, sendo a dose indicada para a cultura na formulação 5:20:20. Foram utilizadas cinco sementes por balde e, após a emergência das plântulas, foi realizado o raleio, deixando apenas duas plantas por balde.
A partir da antese iniciou-se a marcação das flores, para que todas as sementes coletadas estivessem no mesmo estádio de desenvolvimento. Foram realizadas quatro coletas aos 50 dias após a antese, o material coletado foi imediatamente armazenado à temperatura de -80°C até o momento da extração do RNA total.
Para a obtenção do RNA total das sementes, foi utilizado o protocolo do kit Pure Link Plant RNA (InvitrogenTM), de acordo com as instruções do fabricante (Protocolo Pure Link RNA Reagent(InvitrogenTM). A avaliação da qualidade do RNA foi realizado em gel de agarose a 1% corado com brometo de etídeo, enquanto que a pureza do RNA extraído foram mensuradas no equipamento espectrofotômetro NanoVueTM Plus por meio da análise de absorbância (260/280 nm).
As amostras de RNA foram tratadas com a enzima DNAse I (kit Deoxyribonuclease I, Amplification Grade, InvitrogenTM) para remoção de possíveis resíduos de DNA genômico. A seguir, realizou-se a síntese da primeira fita de cDNA utilizando o kit SuperScriptTM III First-Strand Synthesis System for RT-PCR (InvitrogenTM) empregando-se oligo (dT) 50 µM.
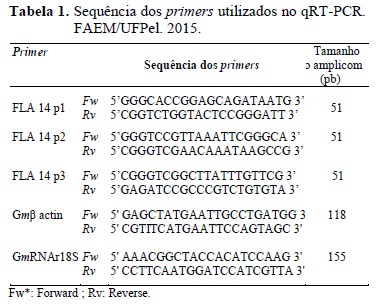
As sequências do gene alvo foram obtidas do banco de dados do NCBI – National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov/) pelas sequências de ESTs correspondentes a Arabidogalactana, e exportadas no banco de dados SoyBase and The Soybean Breeder´s Toolbox (http://www.soybase.org/). Os conjuntos de primers foram desenhados com auxílio do programa BioEdit Sequence Alignmente Editor, observando-se que as região mais conservadas foram obtidas no cromossomo 13, logo após, procedeu-se o alinhamento comparando com o do Arabidogalactana (FLA14). Os pares de primers foram desenhados com auxílio do programa Primer Express 2.0 (Applied Biosystems) (Tabela 1).
As reações de qRT-PCR (Quantitative Real Time Polymerase Chain Reaction) foram realizadas no termociclador 7500 Fast Real-Time PCR Products, (Applied Biosystems, Foster, CA, USA). As condições de ciclagem utilizadas para amplificação foram: desnaturação inicial 50°C durante 2 min, 95°C durante 10 min, 40 ciclos a 95°C durante 15 s, 60°C durante 1 min, 72°C durante 1 min, sendo que os dados foram coletados no segundo ciclo e na última fase.
Para avaliar a eficiência da reação de amplificação de cada gene alvo, foram realizadas diluições seriadas de cDNA as quais foram submetidas à amplificação nas mesmas condições citadas acima. O gráfico gerado pela concentração de cDNA e os respectivos valores de Ct foram empregados para o cálculo da equação de regressão segundo Zhao e Fernald (2005), eficiência da amplificação E=[10(-1/slope)]-1.
A relação da expressão relativa (R) do gene alvo, foi calculada baseando-se na eficiência da amplificação (E) e no ciclo do PCR onde foi detectado o aumento da fluorescência acima do sinal basal (Ct) (Pfaffl 2001).
Os resultados das amplificações das amostras (Ct) obtidas pela qRT-PCR foram analisados considerando os pontos dos Ct gerados no PCR das amostras de um genótipo de expressão, tegumento com coloração preta, relacionados com os Ct da curva padrão dos genótipos com tegumento amarelo, calculado por meio da equação da reta pela análise de regressão linear.
RESULTADOS E DISCUSSÃO
A técnica de qRT-PCR permite a quantificação da expressão dos genes em tempo real, apresentando um maior grau de sensibilidade que o PCR convencional. A quantificação da expressão dos genes é feita de forma relativa, isto é, pela comparação da expressão em um grupo exposto a um estímulo a outro não exposto, denominado de grupo controle (Zarlenga e Higgins 2001).
De acordo com os resultados obtidos no experimento referente a validação dos oligonucleotídeos (primers), para o gene fla 14, foi possível identificar a qualidade da curva padrão que foi aferida a partir do slope da equação de regressão, utilizado para o cálculo da eficiência de amplificação, assim como do coeficiente de determinação (R2) (Deprez et al. 2002).
As eficiências mais ajustadas, entre 1,7 e 1,9, foram evidenciadas para os primers FLA 14 p1 (1,7), FLA 14 p2 (1,7) e ß actina (1,9). No entanto, o FLA 14 p3(4,5) e o GmRNAr18s (5,9) obtiveram valores de eficiência fora dos valores recomendados (Applied Biosystems®), evidenciando possíveis problemas na qualidade destes primers, indicando que os mesmos são ineficientes para estudos posteriores de expressão relativa (Tabela 2).
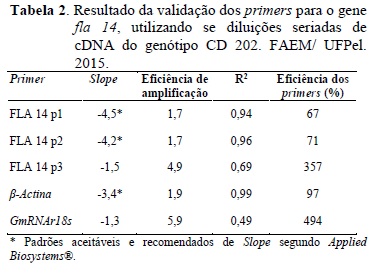
Em relação aos valores dos coeficientes de determinação (R2), estes foram superiores a 67% para os primers, FLA 14 p1, FLA 14 p2 eß actina, indicando que os resultados obtidos, com exceção do gene endógeno GmRNAr18s, apresentaram confiabilidade. Coeficientes de determinação superiores indicam que a quantidade de amplicons que cada primer produz nas diferentes diluições do cDNA sofre redução conforme a redução da concentração inicial de cDNA (diluições) (Tabela 2).
Estudos utilizando o fluorocromo SYBR Green I para análise de qRT-PCR, obteve um coeficiente de regressão de 1, e quase todos os resultados para eficiência encontraram-se entre 70 e 110% (Deprez et al. 2002). Já Pfaffl (2001), afirma que a eficiência deve ser E= 100± 20%.
Apesar da diferença nos valores da eficiência entre os genes endógenos testados, GmRNAr18s (5,9) e ß actina(1,9), a inclusão do gene GmRNAr18s em trabalhos utilizando qRT-PCR, em outros estádios de desenvolvimento do tecido vegetal, pode ser recomendada. Isso pode ser sugerido devido a seus altos valores de expressão, o que é importante quando genes são testados (Stolf Moreira et al. 2011).
No entanto, trabalhos realizados com soja submetida à seca, para observar a expressão gênica da subfamília HD-Zip I, utilizando como controle endógenos os genesß-Actina e GmRNAr18s, validados no programa GeNorm (GeNorm 2002), demonstraram que o gene ß-Actina apresentou uma maior estabilidade em relação ao GmRNAr18s (Pereira et al. 2011), sendo que estes resultados corroboram os encontrados no presente estudo.
Para ser considerado como um gene endógeno o gene deve ser expresso em níveis relativamente constantes nos diferentes tecidos, em todas as etapas de desenvolvimento, não sendo afetado por condições experimentais (Bustin 2002). Portanto, neste estudo, o primer do gene ß actina parece não ter relação direta com o metabolismo do “polissacarídeo”, permitindo assim, sua escolha e utilização como controle endógeno.
A especificidade dos primers foi analisada pela curva de dissociação gerada após a reação de qRT-PCR, as quais foram visualizadas nas temperaturas de 78°C; 83°C e 82°C.
Observa-se que não apresentaram outros picos, indicando que os primers produzem um único fragmento de PCR, ou seja, a amplificação é específica.
Os níveis de expressão do gene fla 14 foram analisados relacionando diferenças existentes entre os quatro genótipos com características contrastantes para a coloração do tegumento (tegumento preto e amarelo) e conteúdo de lignina. Ou seja, a expressão relativa (QR) foi calculada com base nas combinações das características expressas nos genótipos, sendo que as diferenças de expressão são ilustradas na Figura 1.

A análise foi conduzida visando identificar os genes com maior e/ou menor expressão nos diferentes genótipos, obtidos a partir da comparação entre o Ct da cultivar Potência (tegumento amarelo) e o Ct das cultivares IAC e TP (ambos de tegumento preto). Os resultados obtidos para a diferença da expressão foram de 12,21% e 19,42% para IAC 222/Potência e TP/Potência, respectivamente, sendo que nas combinações TP/CD 202 e o IAC 222/CD 202, apresentaram valores de 0,21% e 0,13% respectivamente (Figura 2), quando analisada a expressão relativa para o primer 1.
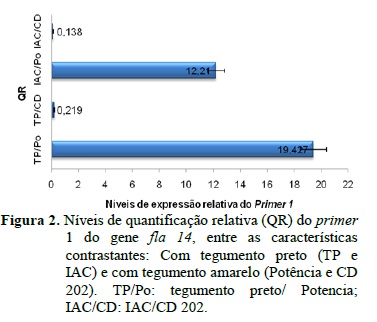
A expressão relativa do primer 1 dos genótipos com tegumento preto (TP e IAC 222), foi maior em relação ao genótipo Potência, sendo que o mesmo resultado foi observado no primer 2.
Entretanto, no primer 2 a expressão diferencial do gene para os genótipos TP e IAC 222, permitiram a melhor diferenciação do genótipo CD 202, apresentando QR superiores aos encontrados com o primer 1.
Nas combinações entre IAC 222/Potência e o TP/Potência foram observados valores de expressão entre 11,31% e 18,63%, respectivamente. No entanto, TP/CD 202 e o IAC 222/CD 202, apresentaram valores de 0,45 e 0,27%, respectivamente, as quais são superiores aos apresentados pelo primer 1. Evidenciando que ambos primers foram eficientes para detectar diferentes níveis de expressão gênica neste genótipo (Figura 3).
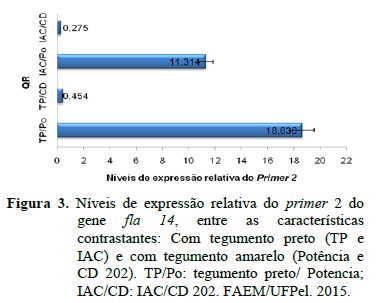
Os resultados obtidos demonstram que ocorreu expressão diferencial do gene fla 14 aos 50 dias após a antese no tegumento da soja, apesar de ainda, não estar completamente elucidada a exata relação entre coloração do tegumento e o teor de lignina na expressão desta proteína. A diferença de expressão encontrada entre as cultivares (TP/Potência, TP/CD 202 e IAC 222/Potência, IAC 222/CD 202) pode ter ocorrido devido aos diferentes teores de lignina no tegumento das sementes. Além disso, de acordo com Menezes et al. (2009), essa variação na expressão pode ser devido a diferenças existentes entres as próprias cultivares, variando entre híbridos, independente dos genitores apresentarem valores altos ou baixos de lignina.
Assim como no presente estudo, Mertz (2010) utilizando a técnica de cDNA-AFLP para identificar genes diferencialmente expressos em tegumentos de sementes de soja, ressaltou que as proteínas FLAs podem explicar o maior conteúdo de lignina presente em sementes de soja com coloração preta. Do mesmo modo, em estudo realizado com algodão (Gossypium hirsutum), os genes fla 6/14/15/18, ressaltam a sua importância sobre os níveis de expressão da fibra do algodoeiro (Huang et al. 2008), sendo a mesma relacionada com a proteção física proporcionada pelo tegumento das sementes. Trabalhos realizados com análise de expressão dos FLAs em trigo (Triticum aestivum) e arroz (Oryza sativa), Faik et al. (2006), sugerem que a proteína, mesmo que fracamente expressa, é encontrada principalmente na semente e na raiz destes genótipos. No entanto, foram realizadas buscas nas bibliotecas de cDNAs do banco de dados TIGR, onde não foi possível verificar expressão desses genes na semente, no endosperma, no pistilo e coroa; contudo, foram encontrados cDNAs medianamente expressos nas anteras e folhas, e altamente expressos na raiz do trigo. Apesar dessa proteína não estar sendo expressa na semente, foi verificado que ela pode estar relacionada à resposta ao estresse por desidratação.
Neste sentido, os resultados obtidos nesse estudo sugerem que os primers desenhados para proteína FLA 14 são eficientes para expressão gênica em tegumentos de sementes de soja de coloração preta, apresentando diferencia de expressão entre tegumentos de sementes com caraterísticas contrastantes. Entretanto, a conformação desses resultados depende de estudos mais detalhados, relacionados com a expressão gênica em tecidos específicos de interesse, com a intenção de aprimorar e ampliar a compreensão de rotas metabólicas ainda não completamente elucidadas como é o caso da proteína FLA 14.
CONCLUSÕES
Os primers FLA 14 p1 e FLA14 p2são eficientes para sua utilização em estudos de expressão gênica em genótipos de soja.
O gene fla 14 apresenta expressão relativa superior na soja com tegumento preto e caracterizado por maior conteúdo de lignina, em relação aos genótipos com tegumento amarelo e menor conteúdo de lignina.
AGRADECIMENTOS
Ao INBIO pela concessão de bolsa à primeira autora.
REFERÊNCIAS BIBLIOGRÁFICAS
Applied Biosystems. Disponível em: suporte@appliedbiosystems.com [ Links ]
Bustin, SA. 2002. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology 29:23-39. [ Links ]
Botelho, FJE. 2012. Qualidade de sementes de soja com diferentes teores de lignina obtidos de plantas submetidas à dessecação. 90f. Tese (Doutorado), Departamento de Fitotecnia, Universidade Federal de Lavras. [ Links ]
Carvalho, NM; Nakagawa, J. 2000. Sementes: ciência, tecnologia e produção. 4a.ed. Jaboticabal, FUNEP. 588 p. [ Links ]
Capeleti, I; Ferrarese, MLL; Krzyzanowski, FC; Ferrarese Filho, O. 2005. A new procedure for quantification of lignin in soybean (Glycine max (L.) Merrill) seed coat and their relationship with resistance to mechanical damage. Seed Science and Technology, 33(2): 511-515. [ Links ]
CONAB (Campanha Nacional de Abastecimento). 2014. Disponível em: http://www.conab.gov.br/OlalaCMS/uploads/arquivos/13_09_10_16_05_53_boletim_portugues_setembro_2014.pdf [ Links ]
Dassou, S; Kueneman, EA. 1974. Screening methodology for resistance to field weathering of soybean seed. Crop science. Madison 24:774-779. Disponível em: http://crop.scijournals.org/cgi/content/abstract/24/4/774 [ Links ]
Deprez R, HL; Fijnvandraat AC; Ruijter, JM; Moorman, A, FM. 2002. Sensitivity and accuracy of quantitative real-time polymerase chain reaction using SYBR green I depends on cDNA synthesis conditions. Analytucal Biochemistry 307:63-69. Disponível em: http://www.sciencedirect.com/science/article/pii/S0003269702000210 [ Links ]
Duke S, H; Kakefuda, G. 1981. Role of the testa in preventing cellular rupture during imbibition of the legume seeds. Plant Physiology 67(2):449-456. Disponível em: http://www.plantphysiol.org/cgi/reprint/67/3/449.pdf [ Links ]
França Neto, JB; Potts, HC. 1979. Efeitos da colheita mecânica e da secagem artificial sobre a qualidade da semente dura em soja. Revista Brasileira de Sementes, 01(2):64-77. [ Links ]
França Neto, JB; Krzyzanowski, FC; Costa, NP. 1999. Metodologia do teste de tetrazólio em sementes de soja. In: Krzyzanowski, FC; Vieira, RD; França Neto, JB (Ed.). Vigor de sementes: conceitos e testes. Londrina: ABRATES. 218 p. [ Links ]
Faik, A; Abouzouhair, J; Sarhan, F. 2006. Putative fasciclin-like arabinogalactan-proteins (FLA) in wheat (Triticum aestivum) and rice (Oryza sativa): identiWcation and bioinformatic analyses. Molecular And Geral Genetic 276:478-494. Disponível em: http://www.ohio.edu/people/faik/Papers/Faik%20et%20al-MGG.pdf [ Links ]
Friedmann, M; Ralph, SG; Aeschliman, D; Zhuang, J; Ritland, K; Ellis, BE; Bohlmann, J; Douglas, C. 2007. Microarray gene expression profiling of developmental transitions in Sitka spruce (Picea sitchensis) apical shoots. Jornal of Experimental Botany 58(3):593-614. Disponível em: http://jxb.oxfordjournals.org/content/58/3/593.full.pdf+html [ Links ]
GeNorm. 2002. Normalization of real-time PCR expression data. Available at: http://medgen.ugent.be/~jvdesomp/genorm [ Links ]
Huang G, Q; Xu W, L; Gong S, Y; Li B; Wang X, L; Xu D; Bao L, X. 2008. Characterization of 19 novel cotton FLA genes and their expression profiling in fiber development and in response to phytohormones and salt stress.Physiologia Plantarum 134:348-359. Disponível em: http://onlinelibrary.wiley.com/doi/10.1111/j.1399-3054.2008.01139.x/pdf [ Links ]
Ma, E; Cholewa, E; Mohamed, T; Peterson, CA; Gijzen, M. 2004. Cracks in the palisade cuticle of soybean seed coats correlate with their permeability to water. Annals of Botany Company 94(2): 213-228. [ Links ]
Menezes M, De; Von Pinho E, V De R; Roveri S, CBJ; Baldoni A; Mendes F, F. 2009. Aspectos químicos e estruturais da qualidade fisiológica de semente de soja. Pesquisa Agropecuária Brasileira 44(12):1716-1723. Disponível em: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-204X2009001200022 [ Links ]
Mertz L, M; Henning F, A; Zimmer P, D. 2009. cDNA-AFLP na identificação de genes relacionados a qualidade fisiológica. Revista Brasileira de Sementes 31(2): 048-053. Disponível em: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0101-31222009000200005 [ Links ]
Mertz L, M; Henning F, A; Cruz H, L da; Meneghello G, E; Ferrari C, dos S; Zimmer P, D. 2009. Diferenças estruturais entre tegumentos de sementes de soja com permeabilidade contrastante. Revista Brasileira de Sementes 31(1):23-29. [ Links ]
Mertz L, M. 2007. Caracterização morfo-fisiologica e identificação de fragmentos de cDNA diferencialmente expressos em tegumentos de sementes de soja com permeabilidade contrastante. Dissertação, Faculdade de Agronomia, Universidade Federal de Pelotas, Pelotas. 60 p. Disponível em: http://www.dominiopublico.gov.br/download/texto/cp074538.pdf [ Links ]
Mertz L, M. 2010. Identificação de genes diferencialmente expressos em tegumento de sementes de soja com caracteres contrastantes. Tese, Faculdade de Agronomia, Universidade Federal de Pelotas, Pelotas. 74 p. Disponível em: http://www.dominiopublico.gov.br/download/texto/cp143544.pdf [ Links ]
Muniz F, RS. 2007. Analise da variabilidade genética em população segregantes de soja. Tese, Faculdade de Ciências Agrárias e Veterinárias do Campus de Jaboticabal. 94 p. [ Links ]
NCBI (National Center for Biotechnology Information). Disponível em: http://www.ncbi.nlm.nih.gov [ Links ]
Pereira A, A; Aguida Morales M, AP; Borém A; Loureiro M, E. 2011. Expressão de genes da subfamília HD-Zip I em soja submetida à seca. Pesquisa Agropecuária Brasileira 46(8):884-889. Disponível em: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-204X2011000800014&lng=pt&nrm=iso&tlng=pt [ Links ]
Pfaffl M, W. 2001. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research 29(9):45. Disponível em: http://www.ncbi.nlm.nih.gov/pmc/articles/PMC55695 [ Links ]
Popp M, P; Dillon C, R; Keisling T, C. 2003. Economic and weather influences on soybean planting strategies on heavy soils. Agricultiral Systems (76):969-984. Disponível em: http://www.sciencedirect.com/science/article/pii/S0308521X02000173 [ Links ]
Shuster I. 2003. O DNA da semente. Consultado el 25 de set 2015. Disponible en: http:// www.coodetec.com.br/artigos. [ Links ]
Silva M, AD. 2003. Morfologia da testa e potencial fisiológico de sementes de soja. Tese,- Faculdade de Ciências Agrárias e Veterinárias, Universidade Estadual Paulista, Jaboticabal. 84 p. Disponível em: http://www.athena.biblioteca.unesp.br/exlibris/bd/bja/33004102050P5/2003/silva_mad_dr_jabo.pdf [ Links ]
SoyBase and The Soybean Breeder´s Toolbox. Disponível em: http://www.soybase.org. [ Links ]
Stolf-Moreira R; Medri M, E; Neumaier N; Lemos N, G; Pimenta J, A; Tobita S; Brogin R, L; Marcelino-Guimarães F, C; Oliveira M, CN; Farias J, RB; Abdelnoor R, V; Nepomuceno A, L. 2011. Soybean Physiology and gene espression during drought. Genetics and Molecular Research 9:1946-1956. Disponível em: http://sumarios.org/sites/default/files/pdfs/gmr851.pdf [ Links ]
Vieira R, D; Minohara L; Panobianco M; Bergamaschi M, CM; Mauri A, O. 1998. Comportamento de cultivares de soja quanto à qualidade fisiológica de semente. Pesquisa Agropecuária Brasileira. Brasília 33(2):123-130. Disponível em: http://orton.catie.ac.cr/cgi-bin/wxis.exe/?IsisScript=ACERVO.xis&method=post&formato=2&cantidad=1&expresion=mfn=014403 [ Links ]
Yang X; Tu L; Zhu L; Fu L; Min L; Zhang X. 2008. Expression profile analysis of genes involved in cell wall regeneration during protoplast culture in cotton by suppression subtractive hybridization and macroarray. Journal of Experimental Botany 59(13):3661-3674. [ Links ]
Zarlenga D, S; Higgins J. 2001. PCR as diagnostic and quantitative technique in veterinary parasitology. Veterinary Parasitology. 101(3-4):215-230. Disponível em: http://www.sciencedirect.com/science/article/pii/S0304401701005684 [ Links ]
Zhao, S; Fernald, RD. 2005. Comprehensive alogarithm for quantitative real-time polymerase chain reaction. Journal of Computational Biology 12(8):1047-1064. [ Links ]














