INTRODUCCIÓN
Las nucleasas guiadas por RNA (RGNs) proveen regulación génica con especificidad de secuencia a través de interacciones de apareamiento de bases entre un pequeño RNA guía y un DNA o RNA blanco. Los sistemas RGN, que incluyen a CRISPR/Cas, son extremadamente prometedores como herramientas programables para la ingeniería genética1,2.
La tecnología CRISPR/Cas se originó a partir del sistema CRISPR/Cas tipo II que provee a las bacterias de una inmunidad adaptativa a virus y plásmidos3. La proteína Cas9 asociada a CRISPR es una endonucleasa que utiliza una secuencia guía (gRNA) dentro de un RNA dúplex (tracrRNA:crRNA) que, al formar pares de bases con la secuencia blanco en el DNA, le permite introducir un sitio específico de rotura en la doble hebra de DNA4. Con el objetivo de optimizar su uso en el laboratorio, el dúplex tracrRNA:crRNA se ha fusionado en una sola molécula de gRNA, llamada RNA guía simple (sgRNA). CRISPR/Cas9 está teniendo un importante impacto en modelos experimentales de genómica funcional5,6. El sistema CRISPR/Cas9 realiza una edición precisa y dirigida del genoma de células vivas. La especificidad es la mayor preocupación en el sistema CRISPR/Cas9, debido a que Cas9 puede cortar sitios que no son totalmente complementarios a la secuencia del sgRNA7. Con el objeto de optimizar la predicción de los sgRNAs con alta especificidad y reducir la frecuencia relativa de corte de sitios no blanco (off-target), se han desarrollado diversos programas informáticos que sirven como herramientas de apoyo8. Su aplicación a estudios de genomas completos permitirá un cribado a gran escala de blancos de drogas9,10 y otros fenotipos11,12, ayudará a generar modelos animales modificados genéticamente que podrán beneficiar a estudios farmacológicos y a la comprensión de enfermedades humanas. Las aplicaciones de CRISPR/Cas9 en plantas y hongos también prometen un cambio en el curso de las investigaciones en el área de la agricultura13-16.
CRISPR/Cas es una novedosa técnica de edición de genes y genomas llamada comúnmente CRISPR. En esta revisión nos centraremos en su función original en procariotas y en cómo los investigadores la han modificado para proporcionar nuevas técnicas de edición de genomas. Discutiremos sobre las ventajas de esta nueva técnica, las formas en que podemos utilizarla y algunas de las limitaciones que aún encontramos en su aplicación.
FUNCIÓN DEL LOCUS CRISPR
Las bacterias y arqueas han desarrollado un sistema inmune de defensa adaptativa llamado “Repeticiones Palindrómicas Cortas Agrupadas y Regularmente Interespaciadas” (“Clustered Regularly Interspaced Short Palindromic Repeats”, CRISPR)17. Los sistemas CRISPR/Cas están compuestos de genes organizados en operones18 y arreglos consistentes en secuencias llamadas espaciadores, interespaciadas por repeticiones idénticas19. CRISPR protege a los procariotas de la invasión de virus y plásmidos20. Este sistema de defensa recae sobre pequeños RNAs que detectan pequeñas secuencias específicas de ácidos nucleicos foráneos. La inmunidad mediada por CRISPR/Cas ocurre en dos fases: adaptación e inhibición. En la fase de adaptación21, las bacterias y arqueas que albergan uno o más loci CRISPR responden a la introducción de material genético, viral o plasmídico, integrando fragmentos cortos de las secuencias foráneas (protoespaciadores) dentro de sus cromosomas en el extremo proximal del arreglo CRISPR22 (Figura 1).
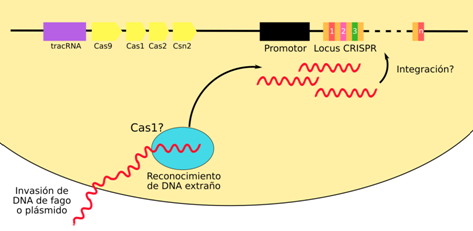
Figura 1: Fase de adaptación. La entrada de un DNA foráneo a una célula a través de la transformación, conjugación o traducción puede llevar a la adquisición de nuevos espaciadores de DNA por la adaptación del complejo Cas (ensamblaje de proteína desconocida). La imagen no representa exactamente la disposición de los loci.
En la fase de inhibición23, se transcribe el elemento repetitivo espaciador y el producto se denomina precursor de CRISPR RNA (pre-crRNA). Éste luego es cortado enzimáticamente para producir CRISPR RNA (crRNA) cortos, los cuales pueden aparearse con secuencias protoespaciadoras complementarias al invasor viral o plasmídico. El blanco de reconocimiento para los crRNAs dirige el corte de genes foráneos mediante el uso de nucleasas Cas que funcionan en complejos con los crRNAs (Figura 2).
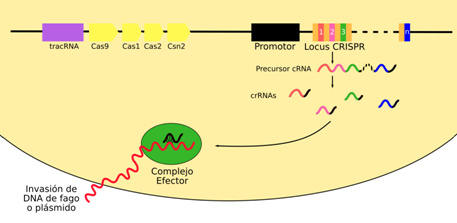
Figura 2: Fase de inhibición. El complejo efector está unido a un crRNA producido por la transcripción del locus CRISPR y el posterior procesamiento. El complejo efector se aparea con una región de ácido nucleico foráneo (por pareamiento perfecto) y allí inhibe al material genético invasivo y lo destruye mediante su corte. La imagen no representa exactamente la disposición de los loci.
El sistema CRISPR mejor caracterizado y utilizado en edición genómica es el de tipo II24. Está compuesto por la nucleasa Cas9, que utiliza como guía a un crRNA que se aparea con el llamado RNA trans activador (tracrRNA), el cual cumple una función estructural25. Estos RNAs son considerados el RNA guía (gRNA) y forman un complejo con la proteína Cas9. Cada unidad de crRNA contiene una secuencia guía de 20 nucleótidos y una repetición parcial directa, donde la primera dirige a Cas9 a un blanco de 20 nucleótidos a través del apareamiento de bases de Watson-Crick26. Cas9 posee los dominios catalíticos RuvC y HNH, además del dominio de interacción con el motivo protoespaciador adyacente (PAM). Cas9 realiza cortes de doble hebra (DSB) utilizando los dominios RuvC y HNH (Figura 3 y Figura 4). Como se ha dicho al inicio, el crRNA y tracRNA se han fusionado para su uso en el laboratorio, dando origen al sgRNA27.
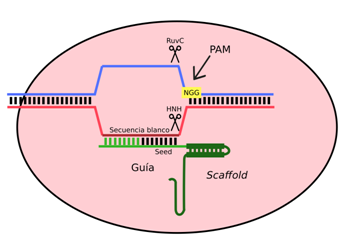
Figura 3: Esquema de la nucleasa Cas9 guiada por RNA. Cas9 (en rosado) es apuntada al DNA genómico a través de un sgRNA que consiste en una secuencia guía de 20 nucleótidos y una secuencia scaffold. La secuencia guía se aparea con el DNA blanco, directamente aguas arriba de un motivo adyacente 5’-NGG requisito (PAM). Cas9 media una ruptura de doble hebra alrededor de 3 pb aguas arriba del PAM.
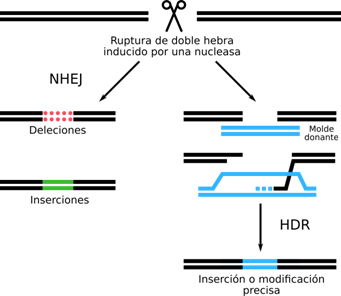
Figura 4: Edición génica inducida por una nucleasa. Las rupturas de doble hebra (DSB) inducidas por una nucleasa pueden ser reparadas por vías de unión de extremos no homólogos (NHEJ) o de reparación dirigida por homología (HDR). Una reparación imprecisa mediada por NHEJ puede producir mutaciones de inserción y/o deleción de longitud variable en el sitio de la ruptura de doble hebra28.
Consideraciones prácticas para la implementación de la tecnología CRISPR/Cas
Debido al rápido progreso en el campo, los usuarios potenciales se encontrarán con una variedad de opciones acerca de cómo implementar la tecnología CRISPR/Cas. Discutiremos algunos parámetros a considerar cuando se desea implementar esta tecnología:
Elección de sitios blanco de sgRNA
Una de las ventajas principales del sistema CRISPR/Cas9 es su simplicidad, ya que es posible desarrollar la técnica en pasos simples. Primeramente, se debe caracterizar el problema de investigación y a partir de allí seleccionar el gen blanco. Una vez elegido el gen con el que se trabajará, se pueden seguir secuencialmente los pasos detallados en la Figura 5.
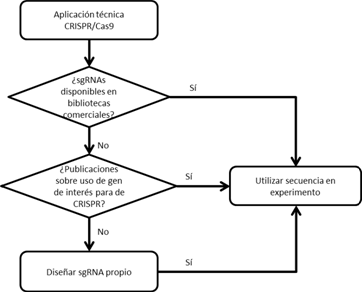
Figura 5: Pasos para la aplicación de la técnica CRISPR/Cas9. Primeramente, es aconsejable buscar secuencias sgRNA diseñadas anteriormente en bibliotecas comerciales. Si esta opción no está disponible, se procede a publicaciones donde se ha utilizado el gen de interés para un ensayo con CRISPR; allí los autores habrán publicado el sgRNA que han empleado en el trabajo y los resultados obtenidos. Finalmente, si no se encuentra una secuencia sgRNA a través de estas opciones, se debe diseñar un sgRNA propio.
En los pasos iniciales del diseño de un sgRNA propio, se deben tener en cuenta cuestiones básicas respecto al gen blanco, como la selección de secciones sin SNPs (polimorfismos de un solo nucleótido), la elección de segmentos específicos del gen de interés en el caso de que ese gen sea parte de una familia génica, y finalmente la utilización de secuencias de genes que especifiquen exactamente la localización de cada exón. Es posible que al utilizar secuencias de mRNA accidentalmente se diseñe el sgRNA en una región de unión exón-exón, la cual no existe en la secuencia de DNA. Se recomienda elegir las secuencias entre los primeros exones, después del codón de inicio29.
Luego de seleccionar el gen blanco, se procede a diseñar el sgRNA. Actualmente existen numerosos sitios web que lo diseñan30 y evalúan su especificidad mediante análisis de BLAST. Debido a que estos recursos utilizan en sus bases de datos genomas de sus organismos de interés, es importante verificar si el organismo de interés del investigador se encuentra dentro de su base de datos.
Si no es posible diseñar el sgRNA con alguno de estos recursos, se debe realizar el diseño de novo. Para ello, hay que seguir una serie de pasos generales, detallados en el Cuadro 1.
Cuadro 1: Pasos a seguir para el diseño de un sgRNA.
Seleccionar una secuencia N20NGG inmediatamente después del codón de inicio y entre los primeros exones.
Tener en cuenta que cualquiera de las dos cadenas puede ser un sitio blanco del sgRNA.
Evaluar las estructuras secundarias y el contenido de “GC”. Este paso puede ser realizado con programas como “Generunner”.
Evaluar la especificidad del sgRNA mediante BLAST.
Entrega de los componentes de CRISPR/Cas
Las RGNs han sido utilizadas en un amplio rango de tipos celulares y organismos utilizando una variedad de métodos de entrega. En cultivos de células de mamíferos los investigadores han usado electroporación32, nucleofección33, inyección de virus recombinante asociado a adenovirus34, y transfección mediada por lipofectamina de plásmidos no replicativos que expresan Cas9 en forma transiente, y sgRNAs28.
También han sido empleados vectores lentivirales para expresar Cas9 y/o sgRNAs en forma constitutiva en cultivos celulares de humanos35 y ratones36. Moléculas de RNA37 y/o plásmidos de DNA transcriptos in vitro han sido inyectados directamente en embriones de peces cebra38, moscas de la fruta39, ratones40 y ratas41. Se ha utilizado con éxito Cas9 en múltiples especies de plantas como trigo, arroz, sorgo, tabaco y Arabidopsis con un amplio rango de métodos de entrega; incluyendo transformación de protoplastos mediada por polietilenglicol, transferencia en embriones, y tejido de hoja mediada por Agrobacterium tumefaciens o bombardeo de células del callo con plásmidos de DNA42.
Los componentes de CRISPR/Cas también pueden ser entregados mediante complejos de ribonucleoproteínas (Cas9 RNPs), consistentes en la proteína Cas9 purificada en conjunto con el sgRNA. Son ensambladas in vitro y pueden ser entregadas directamente43 a la célula mediante electroporación u otras técnicas estándar de transfección (Figura 6).

Figura 6: Métodos de entrega de los componentes del sistema CRISPR/Cas9. a. El sgRNA y la nucleasa Cas9 pueden ser entregadas mediante un plásmido, el cual utiliza la maquinaria celular para producir los componentes y luego realizar la edición génica. b. El sgRNA y la nucleasa Cas9 son sintetizadas in vitro y pueden ser entregados directamente a la célula mediante electroporación u otras técnicas de transfección14.
Aplicaciones de CRISPR/Cas
CRISPR/Cas ha revolucionado los laboratorios de todo el mundo por sus innovadoras aplicaciones: permite realizar análisis sistemáticos de funciones de genes de células de mamíferos, estudios de rearreglos y progresión de cánceres y otras enfermedades, potencialmente permite corregir mutaciones responsables de desórdenes hereditarios44, e inclusive puede ser utilizado para diagnósticos en biomedicina45. Además, ha sido exitosamente empleado para corregir las mutaciones causantes de la fibrosis quística y la β-talasemia en líneas celulares humanas46,47. También puede utilizarse para eliminar la malaria creando mosquitos genéticamente modificados. Sin embargo, su implementación para eliminar ciertas especies invasivas o para alterar ciertas poblaciones salvajes (gene drive)48 ha generado mucha controversia con respecto a las consecuencias ambientales de la eliminación de especies49-51. También es prometedora en el desarrollo de la investigación en el área de la agricultura; en particular con CRISPR/Cas9, se han editado exitosamente los genomas de Arabidopsis, tabaco, arroz, trigo, maíz, y tomate52. La identificación de CRISPR/Cas subraya la forma en que muchas invenciones han avanzado en la biología molecular a partir de la investigación básica de los mecanismos naturales de la replicación del DNA, la reparación, y la defensa contra los virus. Una vez que fue comprendido el mecanismo subyacente a su funcionamiento, se pudieron visualizar sus posibles aplicaciones en biología molecular y genética.
Desafíos actuales en el uso de la tecnología CRISPR/Cas
Debido a su versatilidad, CRISPR/Cas9 se ha convertido en el método más popular de edición génica, triplicando en el 2015 la cantidad de publicaciones relacionadas en comparación a otras técnicas de edición génica como TALEN y ZNF53. Aun así, se presentan aspectos que siguen siendo explorados y representan desafíos54.
Se avanza hacia la comprensión de los diferentes subtipos del sistema CRISPR/Cas y el uso de complejos efectores más pequeños (como el Cpf1)55-57. Por otro lado, la entrega de los componentes de CRISPR/Cas aún sigue siendo un desafío. En el caso específico de los vectores virales, preferidos debido a su baja inmunogenicidad, el gran tamaño de los efectores Cas dificulta su empaquetamiento dentro del vector58. Existe un gran interés en desarrollar estrategias de entrega no virales para introducir los componentes de CRISPR/Cas en las células in vivo. Se han probado compuestos de nanopartículas lipídicas sólidas, lipofectamina, nanopartículas de oro y polímeros catiónicos como vehículos de entrega no viral de Cas9, y el sgRNA ha sido capaz de inactivar genes en forma eficiente in vivo en una variedad de tejidos. Las nanopartículas permiten tratamientos acumulativos repetitivos, por lo que con un pequeño porcentaje de genes editados en cada tratamiento, el efecto positivo total puede incrementar con el número de aplicaciones. Si bien esto parece promisorio, los vehículos de entrega de CRISPR/Cas9 por medio de nanopartículas necesitan ser optimizados con respecto a la biodegradabilidad y el acceso desde la corriente sanguínea hasta el tejido blanco59,60. Se han observado avances significativos en los últimos años para mitigar los defectos asociados con los abordajes de entrega por virus, basados en mRNA, plásmidos y proteínas para el sistema CRISPR/Cas9. A pesar de ello, cada plataforma aún enfrenta diferentes barreras en la traducción. Sin embargo, los avances actuales han llevado al desarrollo de vehículos de entrega con capacidades sin precedentes superando muchos obstáculos que alguna vez estuvieron escondidos en la capacidad traduccional de este sistema61.
En resumen, esta tecnología puede utilizarse para la edición genética en microorganismos y plantas, y en células humanas para la erradicación de enfermedades. Debido a su gran alcance, tanto en investigaciones y en desarrollo de nuevos productos en varios sectores como la industria agro-ganadera y la salud humana, se produjo una atención particular a nivel mundial sobre las consecuencias que puede tener en el ambiente62, la política, la economía63 y la sociedad64-67. Si bien es de gran importancia conocer cómo funciona y sus posibles aplicaciones, no se debe dejar de lado uno de los factores que determina su viabilidad: la regulación de la tecnología68-70. Para poder seguir impulsando la biotecnología mediante la investigación básica y el desarrollo de nuevas tecnologías, es necesario definir y diferenciar las técnicas de edición genética de otras, como la transgénesis, dentro del ámbito regulatorio. Con esto se lograría evitar inconvenientes innecesarios dentro del área regulatoria-legal71 para así poder resolver problemas actuales o desarrollar soluciones más eficientes, sin dejar de lado la ética en la investigación y la bioseguridad que conlleva una tecnología de alto impacto. Si bien los mecanismos más adecuados para regular esta tecnología están aún en discusión, es importante abordar este problema desde varias perspectivas como la social, política, económica y ambiental para lograr aprovecharla al máximo, de manera integral y sustentable.














