1. INTRODUCCIÓN
El aislamiento clínico de las micobacterias atípicas (MA) especies diferentes de Mycobacterium tuberculosis (MT), se ha incrementado considerablemente en los últimos años principalmente con la aparición del virus del SIDA, y con los tratamientos estéticos invasivos. Su identificación y tratamiento han representado un gran desafío(1).
Definir la epidemiología de las MA ha sido más difícil que con MT. La enfermedad producida por estas micobacterias no es de declaración obligatoria a las autoridades de salud pública; por lo tanto, los datos epidemiológicos y de vigilancia no están fácilmente disponibles. Sin embargo, la prevalencia de la enfermedad pulmonar se ha incrementado dramáticamente en todo el mundo durante las últimas tres décadas(2) por los motivos mencionados, además de identificarse con precisión gracias a las técnicas moleculares y al poder discriminatorio de la secuenciación de genes, que los métodos fenotípicos no pueden lograr(3).
El diagnóstico preciso a nivel de especie tiene un impacto directo, debido a los diferentes patrones de sensibilidad de las especies a los medicamentos, especialmente importante en individuos inmunocomprometidos(4), caracterizándose estas micobacterias por una relativa resistencia a un amplio espectro de fármacos antituberculosos, debido, fundamentalmente, a la poca permeabilidad y alta hidrofobia que presenta su superficie celular(4).
Se encuentran ampliamente distribuidas en la naturaleza, fundamentalmente en agua y suelo y la transmisión persona a persona no ha sido demostrada(2). Debido a que estos organismos son ubicuos, la exposición a ellos es universal. En los seres humanos, la enfermedad por micobacterias atípicas se cree que se adquiere por la exposición a factores ambientales, aunque una fuente específica aún no se ha identificado(3). Además, pueden colonizar las vías respiratorias sin causar enfermedad(2).
Aunque en general de baja patogenicidad para las personas, las MA pueden causar una amplia gama de enfermedades clínicas oportunistas llamadas micobacteriosis: la enfermedad pulmonar es la más frecuente, seguida de linfadenitis en niños, enfermedad de la piel y otras infecciones extrapulmonares o diseminadas en pacientes gravemente inmunocomprometidos(5).
El mecanismo de infección más aceptado es por inhalación de aerosoles conteniendo microorganismos del medio ambiente, la ingestión por vía digestiva en los casos de linfadenitis en niños y en las formas diseminadas de los pacientes con SIDA. La inoculación directa de microorganismos del agua y otros vehículos también puede ser responsable de infección en tejido conectivo(2).
Para el diagnóstico de la enfermedad extrapulmonar, es necesaria una combinación de criterios clínicos y bacteriológicos (como el aislamiento de sitios normalmente estériles), a veces ayudados por la confirmación histopatológica. Para el diagnóstico de enfermedad pulmonar, ha sido difícil determinar si un cultivo positivo representa solo contaminación de laboratorio, colonización del tracto respiratorio o verdadera enfermedad. Una combinación de las características clínicas, radiológicas y bacteriológicas también se ha sugerido para el diagnóstico de verdadera micobacteriosis pulmonar(6).
En Brasil de acuerdo a algunas publicaciones la frecuencia de aislamiento de micobacterias atípicas es de alrededor del 10%, aisladas mayoritariamente de muestras pulmonares y en menor medida de muestras extrapulmonares como ganglio y biopsia de piel, con las especies del complejo CAM como principales agentes etiológicoa, además de M. abscesus, M. fortuitum y M. kansasii, con presencia de coinfección en pacientes VIH, y con prevaelncia en el sexo masculino(24,25,26).
En Chile encontramos mayor aislamiento en muestras pulmonares (alrededor del 90%), con identificación mayoritaria de especies del Complejo CAM (M. intracellulare, M. avium), además de M. kansasii, M. abscessus y M. fortuitum(27,17), con frecuencia de aislamiento de micobacterias No Mt del 8%.
En la Argentina se describen publicaciones con aislamiento de micobacterias atípicas en alrededor del 5% de los casos, mayormente aisladas en muestras de origen pulmonar (esputo especialmente), con las especies más aisladas: complejo CAM, M. abscessus, M. fortuitum, M. kansasii y M. chelonae. En muestras extrapulmonares fueron aisladas de sangre, médula ósea, biopsia, LCR, con coinfección con VIH(19,20,21).
El objetivo principal de este protocolo de estudio consistió en determinar la frecuencia de aislamiento de las micobacterias atípicas o no MT en los cultivos estudiados.
2. MATERIALES Y MÉTODO
El trabajo es de tipo observacional descriptivo, de corte transverso y temporalmente retrospectivo. El intervalo temporal de estudio se sitúa entre los años 2010 y 2013.
2.1. Muestreo y recolección de datos
El muestreo fue no probabilístico de conveniencia por casos consecutivos, realizado en el Departamento de Tuberculosis, perteneciente al Laboratorio Central de Salud Pública (LCSP), ubicado en Asunción, Paraguay.
El LCSP es el laboratorio de referencia nacional perteneciente al Ministerio de Salud Pública, y lidera la Red Nacional de Laboratorios de diagnóstico de tuberculosis, constituido a su vez, durante el momento del estudio, por la Red de Baciloscopía (131 servicios), la Red de cultivo (11 laboratorios), y la Red de GeneXpert MTB/RIF (14 laboratorios).
De un total de 2765 cultivos positivos, 171 aislamientos habían sido identificados como micobacterias atípicas (MA) o Micobacterias no Mycobacterium tuberculosis (MNT); el resto de los aislamientos fueron identificados como MT. Las 171 cepas de micobacterias atípicas fueron recuperadas del cepario del Departamento de Tuberculosis donde se encontraban registradas con un código y año de aislamiento. En la mayoría de los casos no se había llegado a especie y fueron clasificadas solamente en uno de los 4 grupos Runyon.
Origen de las cepas: Los aislamientos provienen de los laboratorios de la Red Nacional de Cultivo y de otros servicios de salud no pertenecientes al Ministerio de Salud, por lo cual la población en estudio proviene de todo el país.
2.2. Técnicas laboratoriales realizadas
El reaislamiento fue realizado en Löwenstein Jensen, con incubación a 37 ± 1 oC hasta el crecimiento de colonias. La identificación de los aislamientos fue realizada con las siguientes técnicas:
a) PRA-hsp65:
Esta técnica molecular permite identificar la mayoría de las especies de micobacterias atípicas.
El gen hsp65 que consta de 441 pb, y que codifica para una proteína de shock térmico, fue amplificado por PCR con el par de primers Tb11 (5’-ACC AAC GAT GGT GTG TCC AT) y Tb12 (5’-CTT GTC GAA CCG CAT ACC CT) (7) (Tabla 1), y el producto obtenido fue digerido con dos enzimas de restricción, BstE II y Hae III. Los patrones de restricción obtenidos luego de la electroforesis permitieron definir la especie según el número, tamaño y migración de los patrones de restricción al confrontarlo con un algoritmo disponible en el sitio electrónico PRASITE disponible en http://app.chuv.ch/prasite/index.html
b) Secuenciación de genes 16S rRNA, hsp65 y rpoβ:
El genoma de Mycobacterium tuberculosis, especie tipo del género Mycobacterium ya se ha secuenciado completamente; contiene 4,4 millones de pares de bases con 4.000 genes que codifican proteínas y 50 que codifican RNA(8). Las micobacterias contienen regiones bien conservadas de su DNA específicas de género y regiones hipervariables específicas de especie. Estas últimas son las que se utilizan como dianas, siendo muy bien estudiadas: el gen hsp65 (heat shock protein), que codifica la proteína de 65 KDa, el gen 16S RNAr que codifica la subunidad ribosómica 16S y el gen rpoβ, que codifica la subunidad β de la RNA polimerasa, entre otros. Aunque la secuenciación es muy específica en la discriminación de especie entre las micobacterias, se utiliza por lo general en los laboratorios especializados, ya que debido a su alto costo no puede realizarse de rutina (4).
Para el desarrollo de este protocolo, se realizó amplificación por PCR de cada uno de los genes con los primers correspondientes, para las cepas seleccionadas. Los amplicones obtenidos fueron enviados a Macrogen Inc de Korea para ser secuenciados. Esta técnica permite la identificación de muchas micobacterias en las que no es posible hacerlo por métodos fenotípicos y constituye el patrón de referencia para la identificación genotípica(9) (Tabla 1).
Tabla 1 Primers utilizados para las técnicas moleculares para la identificación de micobacterias atípicas
| Target/ Técnica | Primer | Secuencia | Producto de PCR | Referencias |
|---|---|---|---|---|
| Hsp65/ PCR | Tb11 | ACC AAC GAT GGT GTG TCC AT | 441 pb | 3 |
| Tb12 | CTT GTC GAA CCG CAT ACC CT | 441 pb | 3 | |
| 16S rRNA gene/Sec | pA | AGAGTTTGATCCTGGCTCAG | 1535 pb | 14, 48, 49 |
| pH | AAGGAGGTGATCCAGCCGCA | |||
| hsp65gene/ | HSPF3 | ATCGCCAAGGAGATCGAGCT | 644 pb | 28, 46, 47 |
| Sec | HSPR4 | AAGGTGCCGCGGATCTTGTT | ||
| rpoβ gene/ Secuenciación | Myco F | GGCAAGGTCACCCCGAAGGG | 764 pb | 14, 32, 45 |
| Myco R | AGCGGCTGCTGGGTGATCATC |
El gen 16S rRNA contiene regiones altamente conservadas, que permiten establecer la relación taxonómica profunda entre familias, clases y filos, así como regiones variables que posibilitan discriminar entre especies dentro del mismo género(10). Estas características permiten utilizar el gen como marcador filogenético y herramienta de identificación(11). El grado de conservación es resultado de la importancia de este gen como un componente crítico de la función celular(12).
El gen hsp65 codifica como producto a la proteína de choque térmico (heat shock protein) sintetizada por la bacteria en presencia de factores adversos como temperaturas extremas, cambios de pH, de presión, presencia de toxinas, etc. El análisis de la secuencia del gen hsp65 muestra un mayor grado de divergencia que el análisis del gen 16S rRNA(13). Debido a sus características, fue un buen blanco para desarrollar la técnica de polimorfismo de restricción PRA-hsp 65 para la identificación de las micobacterias(13), y es una opción para reemplazar a esta técnica como herramienta de identificación en la mayoría de los casos secuenciando el gen.
El gen rpoβ codifica la producción de la subunidad β de la enzima RNA polimerasa. Es un buen complemento del gen 16S rRNA para la identificación. Es uno de los blancos mejor caracterizados para el género Mycobacterium para identificación. La presencia de polimorfismo nucleotídico múltiple a lo largo de la secuencia del gen rpoB entre las micobacterias que permiten la identificación está bien establecido(14-16).
3. RESULTADOS
3.1. Según características demográficas y epidemiológicas
Según la edad, la mayor frecuencia fue obtenida en la Adultez de 33 a 59 años: 55 casos (32,2%), y en la Tercera edad, con mayores de 60 años 46 (26,9%); según sexo: hombres 107 (62,6%) y mujeres 64 (37,4%) (Tabla 2).
Tabla 2 Frecuencia de aislamiento según categoría de edades y sexo de la población en estudio
| Variables Demográficas | Frecuencia | Porcentaje (%) |
|---|---|---|
| Categoría de edades | ||
| Infancia (menor de 10 años) | 6 | 3,5 |
| Juventud (11 a 32 años) | 28 | 16,4 |
| Adultez (33 a 59 años) | 55 | 32,2 |
| Tercera edad (mayor a 60 años) | 46 | 26,9 |
| Sin datos | 36 | 21 |
| Sexo | ||
| Hombre | 107 | 62,6 |
| Mujer | 64 | 37,4 |
En cuanto a los pacientes con co-infección con VIH (PVVS Persona que Vive con Virus del SIDA) se identificaron 21 casos positivos (12,3%) (Tabla 3).
Tabla 3 Pacientes con co-infección VIH
| PVVS | FRECUENCIA | PORCENTAJE (%) |
|---|---|---|
| SI | 21 | 12,3 |
| SD/NO? | 150 | 87,7 |
| Total | 171 | 100 |
3.2. Según características de las muestras
Las muestras según su origen fueron distribuidas en: pulmonar 152 casos (89,1%) y extrapulmonar 19 casos (10,9%), obteniéndose en esputo la mayoría de los aislamientos (Tabla 4).
Tabla 4 Frecuencia de aislamiento según el tipo de muestra
| TIPO DE MUESTRA | FRECUENCIA | PORCENTAJE (%) |
|---|---|---|
| Esputo | 147 | 86 |
| Heces | 5 | 2,9 |
| Hemocultivo | 4 | 2,4 |
| Lavado Broncoalveolar (BAL) | 3 | 1,9 |
| Secreción purulenta | 2 | 1,1 |
| Biopsia | 2 | 1,1 |
| Lesión de piel | 2 | 1,1 |
| PAMO | 2 | 1,1 |
| Líquido gástrico | 1 | 0,6 |
| Líquido duodenal | 1 | 0,6 |
| Secreción traqueal | 1 | 0,6 |
| Absceso | 1 | 0,6 |
| Total | 171 | 100 |
3.3. Identificación de los aislamientos
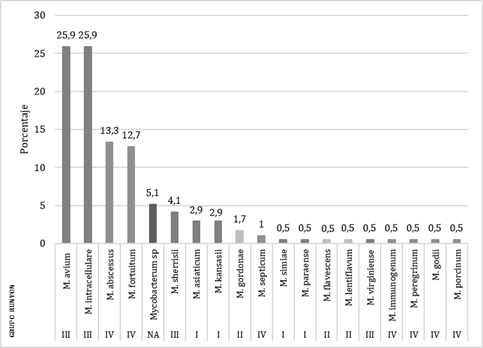
De los 171 aislamientos analizados, no se llegó a especie en 9 casos al no verificarse concordancia entre los resultados obtenidos entre los genes, o debido a que la homología obtenida no correspondía a similitudes ≥ 99.7% para 16S rRNA, ≥ 99% para hsp65, o ≥ 97% para rpoβ, que se requieren para establecer especie. Los resultados obtenidos fueron los siguientes: Mycobacterium intracellulare 44 aislamientos (25,9%), Mycobacterium avium 44 aislamientos (25,9%), Mycobacterium abscessus 23 aislamientos (13,3%), Mycobacterium fortuitum 22 (12,7%); el 17,1% restante correspondía a otras 14 especies y en el 5,1% no se llegó a identificación de especie (Figura 1).
4. DISCUSIÓN
En cuanto a la frecuencia de aislamiento general de micobacterias atípicas encontrada en las muestras que fue del 6,2%, se ha observado en publicaciones con períodos de tiempo cercanos al realizado en este estudio (que también presentan coincidencia mayoritariamente con las especies identificadas), que el Instituto de Salud Pública de Chile informa un 8,4% de aislamientos en el año 2008(17), el Departamento de Neumología del Hospital Público de Teresiña, localizado en el estado de Piauí, Brasil, en el año 2007 reporta un 9%(24); el Hospital de Santa María, Río Grande do Sul, Brasil entre los años 2008 y 2010 informa 33% de aislamiento de MA(18); en otro estudio en Buenos Aires, entre los años 2003 y 2004, la frecuencia de MA fue de 5,1%(19); también en Buenos Aires otro estudio realizado en el norte de la provincia entre 2004 y 2010 reporta 6,1% de aislamientos de MA(20); y en el Hospital Regional de Tuberculosis de la Provincia de Córdoba en un estudio entre los años 1991 y 2000 reporta 9% de aislamientos(21), mientras que Uruguay en un estudio realizado entre 1990 y 1999 informa una frecuencia del 6,3%(22). En este estudio la frecuencia encontrada fue de 6,2% el cual, teniendo en cuenta que fue realizado en años posteriores a los estudios citados, aparenta una cifra cercana. En Paraguay existe un trabajo de investigación sobre la frecuencia de aislamiento de estas micobacterias realizado en el Instituto de Investigaciones en Ciencias de la Salud, entre el 2006 y el 2010, con las mismas especies identificadas(23).
Según los hallazgos, se observaron mayor cantidad de aislamientos en muestras de origen pulmonar. Sin embargo, de acuerdo a los estudios publicados, algunos coinciden y otros mencionan que es más frecuente aislar micobacterias atípicas en muestras extrapulmonares, adjudicando los aislamientos pulmonares a contaminaciones o colonizaciones en su mayoría.
El factor de riesgo de infección por VIH presente en el 12,3% de los pacientes confirma la mayor predisposición a sufrir infecciones por micobacterias oportunistas en estos casos, lo que ya está demostrado ampliamente.
Con respecto al grupo etario también se esperaría mayor proporción de casos en los extremos de la vida en los cuales el sistema inmunológico se encuentra más debilitado.
Los resultados obtenidos arrojan como especies más frecuentemente aisladas a M. intracellulare (25,9%) y M. avium (25,9%), seguidos del grupo de crecimiento rápido con M. abscessus (13,3%) y M. fortuitum (12,7%), lo que concuerda con la mayoría de los datos regionales y mundiales publicados(17,18,19) sobre aislamiento de micobacterias atípicas en muestras clínicas. La frecuencia de aislamiento de M. kansassi, frecuentemente mencionado como agente causal de micobacteriosis, junto con las especies mencionadas anteriormente, en este estudio fue muy baja, solamente del 2,9%.
La combinación de las técnicas PRA-hsp65 y secuenciación de los genes 16S rRNA, hsp65 y rpoB ha permitido llegar a la identificación de la mayoría de los casos y hoy en día podrían complementarse con secuenciación de otros blancos como sodA, recA, ITS 16S-23S entre otros para lograr la identificación total de los aislamientos.
Teniendo en cuenta que este estudio fue realizado hace algunos años, hoy en día podría lograrse la identificación de todos los aislamientos sumando otros genes de identificación como sodA, ITS 16S-23S, gyrA/B, secA1 y recA.
Como posible limitación del estudio, podemos mencionar la calidad de la ficha clínico-epidemiológica remitidas con las muestras. Esto se debió a que muchas muestras no estuvieron acompañadas por una ficha clínico-epidemiológica que declare los datos epidemiológicos, o fueron remitidas de manera incompleta. Esto dificultaba descartar posible contaminación o colonización transitoria que pudieron contribuir a la sobreestimación de la frecuencia en muestras no estériles como esputo que representaba la mayor cantidad de muestras estudiadas, donde un único aislamiento no diagnostica la enfermedad. El aislamiento único en una muestra estéril, en cambio, tiene mayor relevancia. La coinfección en pacientes VIH podría también estar subestimada por la no declaración de esta condición en la ficha epidemiológica de los pacientes, por lo que esto podría representar un sesgo en los resultados obtenidos.
El aislamiento de estas micobacterias en casi todos los casos fue fortuito, ya que ingresaron al Departamento de Tuberculosis con solicitud de estudio para Mycobacterium tuberculosis como agente etiológico sospechoso, y no para confirmar o descartar una micobacteriosis. Incluso hay casos de pacientes que ingresaron ya tratados por tuberculosis con sospecha de resistencia a las drogas antibacilares, en los que se terminó confirmando micobacteriosis.
5. CONCLUSIONES
La frecuencia de aislamiento de micobacterias atípicas en las muestras positivas fue del 6,2%.
Los resultados obtenidos muestran que las especies más frecuentemente aisladas corresponden a M. intracellulare (25,9%) y M. avium (25,9%), micobacterias de crecimiento lento, seguidos de M. abscessus (13,3%) y M. fortuitum (12,7%), pertenecientes al grupo de micobacterias de crecimiento rápido.
El factor de riesgo de infección por VIH presente fue del 12,3 %.
Con respecto a los aislamientos según origen de la muestra (pulmonar y extrapulmonar), se encontró que fue mayor en las muestras de origen pulmonar (89,1%).
Con respecto al grupo etario, en este estudio se encontró que fue mayor en la etapa adulta (32,2%) seguida de la tercera edad (26,9%).












 uBio
uBio