INTRODUCCIÓN
En la actualidad, la Organización Panamericana de la Salud (OPS) considera que la Resistencia Bacteriana representa un grave problema para la Salud Pública. Las infecciones generadas por bacterias resistentes a la terapia antibiótica convencional, ocasionan 700.000 muertes por año y se estima que para el año 2050 la Resistencia Bacteriana ascenderá su mortalidad anual a 10.000.000 de muertes 1.
Por otro lado, acorde a la Guía para el control de Infecciones Asociadas a la Atención a la Salud (IAAS), la contaminación microbiológica de las aguas de los Hospitales puede estar asociada a la aparición de brotes de infecciones y dicha contaminación se relaciona a factores como las manos del personal y/o usuarios de los Hospitales, colonizadas como consecuencia de la alta carga bacteriana del agua y la acumulación de bio-películas en los tanques de suministro de agua, debido a la cloración deficiente y al mal diseño del tanque 2.
Para determinar la existencia de contaminación microbiológica en el agua, se tienen en cuenta distintas normativas en cada país. En Paraguay, el Instituto Nacional de Tecnología, Normalización y Metrología (INTN), en la norma NP 24-001-80-2001 (INTN 2001), establece que para calificar como potable, el agua debe tener recuento de heterótrofos totales inferior a 500 UFC/mL y ausencia de coliformes totales, coliformes fecales y Pseudomonas aeruginosa 3.
Aunque las normativas abarcan el control de la presencia de bacterias con potencial patógeno, las mismas no contemplan si se trata de bacterias resistentes, capaces de transferir sus genes de resistencia y propiciar la aparición de brotes de infecciones nosocomiales.
Las bacterias multi-drogo resistentes (MDR) son bacterias resistentes a la acción de 3 clases de antibióticos o más. Las bacterias Gram (-) MDR a menudo resisten penicilinas, cefalosporinas e incluso carbapenémicos, dejando como única alternativa la terapia con fármacos nefrotóxicos como la colistina 4.
Existen estudios que han aislado Escherichia coli productora de Betalactamasas de Espectro Extendido (BLEE) en muestras de agua potable, Escherichia coli MDR en muestras de agua efluente de Hospitales, y Pseudomonas aeruginosa formadora de biopelícula en muestras de agua potable 5,6.
La detección de bacterias con multi-resistencia en agua de hospital, requiere la aplicación de medidas con carácter urgente; debido a que las bacterias MDR son capaces de presentar una combinación de mecanismos de resistencia como BLEE con sobreproducción de bombas de eflujo, y por ende, son las que tienen mayor capacidad de actuar como vector en la propagación de genes de resistencia, de una cepa ambiental a otra, a través de las conexiones de agua, como los alcantarillados municipales 6,7.
En consecuencia, el objeto de este estudio fue evaluar el perfil de resistencia de las cepas bacterianas aisladas en muestras de aguas de Establecimientos de Salud.
MATERIAL Y MÉTODOS
Este es un estudio observacional descriptivo, transversal. El muestreo se realizó en un estudio preliminar 8, que se efectuó durante los meses de octubre hasta diciembre del año 2020, acorde a las directrices de la Norma NP 24 005 81 9.
Se incluyeron las muestras que provenían de Establecimientos de Salud urbano y rural, de los 22 distritos del Departamento de Caaguazú, de Paraguay. En dicho estudio preliminar, se analizaron un total de 104 muestras de agua (80 de agua tratada de pozos artesianos y 24 de agua potable de tanques), y se identificaron 55 aislados bacterianos con potencial patógeno.
Los aislamientos con potencial patógeno fueron de las especies Enterobacter cloacae, Escherichia coli, Citrobacter brakii, Citrobacter freundii, Klebsiella pneumoniae, Citrobacter koseri, Klebsiella oxytoca y Pseudomonas aerugionosa.
En el Procesamiento de muestras, en el estudio preliminar, se utilizaron sistema miniaturizado API 20 E para la identificación de las especies de Enterobacterales y sistema miniaturizado API 20 NE para la identificación de la especie Pseudomonas aeruginosa.
En este estudio que inicio su procesamiento el mes de julio del año 2021, se empezó con el descongelamiento de las cepas aisladas, las cuales fueron almacenadas en medio Skim milk y conservadas a -80°C. El descongelado se realizó en estufas a 37°C y luego se re-aislaron las colonias de Enterobacterales en medio MacConkey y las colonias de Pseudomonas aeruginosa en medio Cetrimide.
Una vez que se obtuvieron cultivos frescos y puros, se efectuaron antibiogramas a las 55 cepas identificadas, utilizando placas con agar Mueller hinton y discos de antibióticos: amoxicilina con ácido clavulánico (AMC), aztreonam (ATM), amikacina (AK), ceftazidime (CAZ), ciprofloxacina (CIP), colistina (CT), ceftriaxona (CTX), ácido etilendiaminotetraacético con mercaptoacetato de sodio (EDTA/SMA), cefoxitina (FOX), cefepime (FEP), gentamicina (GEN), imipenen (IMP), cefalotina (KF), levofloxacina (LEV), meropenem (MEM), trimetroprima sulfametoxazol (SXT), piperacilina tazobactam (TZP), con el método de difusión de Kirby-Bauer 10.
También se observó si había producción de: AmpC, BLEE y Carbapenemasas, a través de técnicas fenotípicas. Para AmpC se usó la técnica de aproximación de discos de CAZ, IMP y TZP, para BLEE se utilizó la técnica de sinergia con los discos de AMC, ATM, CAZ, CTX, FEP y para Carbapenemasas se realizó prueba enzimática, con test rápido (Rapidec carba NP) 11-13.
Para la interpretación de los antibiogramas, se tuvieron en cuenta los puntos de corte de los lineamientos del Clinical & Laboratory Standards Institute (CLSI) 14.
Análisis de datos obtenidos.
Los datos fueron cargados en planilla electrónica Microsoft Office Excel 2017© y posteriormente se exportaron al paquete de software estadístico STATA 14.0® (Stata Corporation, College Station, Texas, USA), para su procesamiento estadístico.
Aspectos éticos
El protocolo de este trabajo cuenta con la aprobación del Comité de Ética de la Facultad de Ciencias de la Salud, de la Universidad Santa Clara de Asís (USCA). Resolución Nº002/2020.
Además, este estudio cuenta con los permisos, de los directivos de cada uno de los Centros de Salud involucrados.
RESULTADOS
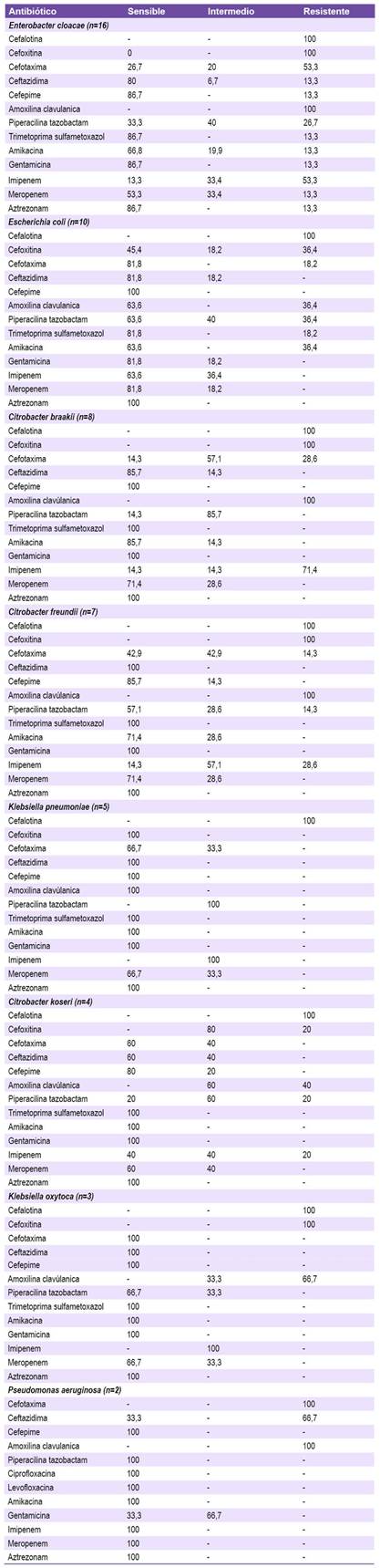
De las 104 muestras de agua analizadas, se aislaron 55 cepas con potencial patógeno en las cuales se efectuaron pruebas de sensibilidad antibiótica. En las cepas de complejo Enterobacter cloacae (n= 16), se observó 100% de resistencia a cefalosporinas de primera y segunda generación (KF, FOX) y a AMC, 53,3% de resistencia a cefalosporina de tercera generación (CTX) e IMP, 26,7% de resistencia a SXT y 13,3% de resistencia a cefalosporinas de tercera y cuarta generación (CAZ, FEP), aminoglucósidos (AK, GEN) y al resto de los carbapenémicos (MEM, ATM). En las cepas de Escherichia coli (n= 10), se detectó 100% resistencia cefalosporina de primera (KF), 36,4% de resistencia a cefalosporinas de segunda generación (FOX), AMC, TZP y AK y 18,2% de resistencia a cefalosporinas de tercera (CTX) y SXT. En las cepas de Citrobacter braakii (n=8) se constató 100% de resistencia a cefalosporinas de primera y segunda generación (KF, FOX), y a AMC, 71,4% de resistencia a IMP y 28,6% de resistencia a cefalosporina de tercera (CTX). En las cepas de Citrobacter freundii (n=7) se visualizó 100% de resistencia a cefalosporinas de primera y segunda generación (KF, FOX) y a AMC, 28,6% de resistencia a IMP, 14,3% de resistencia a TZP y cefalosporinas de tercera generación (CTX). En las cepas de Klebsiella pneumoniae (n=5) se visualizó 100% de resistencia a cefalosporina de primera generación (KF). En las cepas de Citrobacter koseri (n=4) se observó 100% de resistencia a cefalosporina de primera generación (KF), 40% de resistencia a AMC y 20% de resistencia a cefalosporina de segunda generación (FOX), IMP y TZP. En las cepas de Klebsiella oxytoca (n=3) se constató 100% de resistencia a cefalosporinas de primera y segunda generación (KF, FOX) y 66,7% a AMC. En las cepas de Pseudomonas aerugionosa (n=2) se constató 100% de resistencia a cefalosporina de tercera generación (CTX) y AMC y 66,7% de resistencia a otra cefalosporina de tercera generación (CAZ). Tabla 1.
Tabla 1 Perfil de resistencia de los aislamientos bacterianos de las muestras de agua de los Establecimientos de Salud (%). N=55
En cuanto a los mecanismos de resistencia, se detectó Betalactamasas tipo AmpC en 100% de las cepas de Pseudomonas aeruginosa, en 85,7% de las cepas de Citrobacter braakii, en 66,7% de las cepas de complejo Enterobacter cloacae, en 42,8% de las cepas de Citrobacter freundii y en 40% de las cepas de Citrobacter koseri. Se constató presencia de BLEE en 60% de las cepas de Citrobacter koseri, en 42,8% de las cepas de Citrobacter braakii, en 36,4% de las cepas de Escherichia coli, en 33,3% de las cepas de Klebsiella pneumoniae, y en 28,6% de las cepas de Citrobacter freundii. También se observó producción de Carbapenemasas en 13,3% de las cepas de complejo Enterobacter cloacae y se confirmó multi-drogo resistencia en 13,3% de las cepas de complejo Enterobacter cloacae. Figura 1.
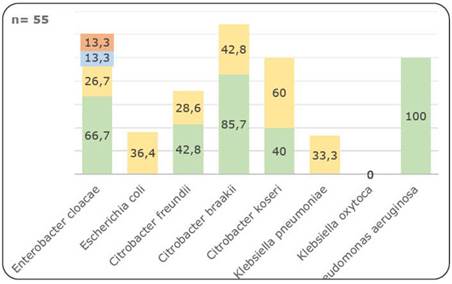
Figura 1 Detección AmpC, BLEE, Carbapenemasas y MDR en los aislamientos bacterianos de muestras de agua de Establecimientos de Salud (%)
Además, se observó la aparición en simultáneo, de Betalactamasas tipo AmpC y BLEE en 6 (10%) de los aislamientos, de las especies Enterobacter cloacae (5%), Citrobacter Braakii (3,3%) y Citrobacter freundii (1,7%). También la conjunción de Betalactamasas tipo AmpC con Carbapenemasas en 2 (3,3%) de las cepas de la especie complejo Enterobacter clocae, las cuales resultaron ser MDR.
DISCUSIÓN
En esta investigación se detectaron aislados bacterianos resistentes en muestras de agua de establecimientos de salud (agua potable de tanques y agua tratada de pozos artesianos) y aunque en la mayoría se pudo observar el comportamiento intrínseco de cada especie, se destacan los siguientes hechos:
- En los aislados de complejo Enterobacter cloacae se pudo observar resistencias propias de la especie, resistencia a cefalosporinas de las cuatro generaciones, resistencia, disociación de resistencia a carbapenémicos y producción de AmpC, BLEE y Carbapenemasas, hallazgo semejante a estudios como el de Lépesová et al, que aisló Enterobacter cloacae BLEE en muestras de agua residual de Hospitales y el de Martins et al, que halló Enterobacter spp. resistente a dos grupos de antimicrobianos en muestras de agua para abastecimiento comunitario 7,15.
Las resistencias a cefalotina, cefoxitina y amoxilina clavúlanica son intrínsecas de la especie, cuando se genera Betalactamasas tipo AmpC de clase C, la resistencia a ceftazidima, cefotaxima y aztreonam se presenta cuando hay producción de Betalactamasas tipo AmpC derreprimida y se amplía la hidrólisis. En los casos donde hubo resistencia a imipenem con sensibilidad a meropenem, se podría tratar de un mecanismo de resistencia adquirida, que otorga impermeabilidad (AmpC + déficit de porina Opr D2), mecanismo que es con frecuencia observado en Pseudomonas, pero que pudo haber sido adquirido por la coexistencia en los tanques o pozos de agua de ambas especies 11,16-19.
En cuanto a la aparición de resistencia a cefalosporina de cuarta generación, aminoglúcidos y carbapenémicos se debió a la generación de carbapenemasas. Respecto a la presencia de AmpC y BLEE, ambas son consideradas betalactamasas propias de la especie, pero en el caso de la producción de carbapenemasas, esto podría atribuirse a la mutación cromosómica y/o adquisición de mecanismos plásmidicos, mutación que pudo estar propiciada por las técnicas de saneamiento deficientes de los tanques y pozos de suministro de agua, puesto que la cloración no es un método efectivo para eliminar microrganismos y solo favorece la generación de mayor resistencia bacteriana (17,18,20,21,22.
- En parte de los aislados de Escherichia coli se pudo observar resistencia a cefalosporinas, betalactámicos y aminoglúcidos, con una resistencia disociada a carbapenémicos. Además, en los aislados de Escherichia coli y Klebsiella pneumoniae se constató la producción de BLEE (36,4% y 33,3%). Este resultado es comparable con investigaciones como la de Mahmud et al, que halló 17,2% de E. coli productora de BLEE en muestras de agua potable comunitaria (22) y como la de Lépesová et al, que encontró 70% de E. coli productora de BLEE en muestras de agua residual de Hospitales 7.
En los aislados de Escherichia coli, la resistencia intermedia a imipenem y meropenem acompañada de sensibilidad a aztreonam, podría deberse a la generación de metalobetalactamasas, lo cual también explicaría la resistencia que los aislados presentaron a piperacilina tazobactam, cefalosporinas de tercera generación y aminoglucósidos. En cuanto al hallazgo de BLEE, en las dos especies citadas, es considerado un comportamiento previsible, puesto que son las dos especies que generan con mayor frecuencia este tipo de betalactamasas. Se destaca que la aparición de metalobelactamasas en la especie E. coli, podría deberse a transferencia horizontal de genes resistencia de especies del género Pseudomonas dentro de los tanques y pozos de agua con un mantenimiento irregular 18,21,23,24.
- En los aislados de Citrobacter (braakii, freundii y koseri) se hallaron en su mayoría resistencias y mecanismos intrínsecos del género, como producción de AmpC, pero también se observó disociación de resistencia a carbapenemicos y producción de BLEE, resultado que podría indicar transferencia de mecanismos de resistencia.
Los hallazgos son similares al estudio de Lépesová et al, que halló Citrobacter freundii productora de BLEE en muestras de agua residual de Hospitales de República Checa y Eslovaquia 7; pero difiere del estudio de Martins et al, que aisló Citrobacter spp. sensible a todos los grupos de antimicrobianos en muestras de aguas de fuentes de abastecimiento público de la ciudad de San Paulo 15.
Respecto a las tres especies del género Citrobacter, la detección de Betalactamasas de tipo AmpC, es atribuible a la naturaleza de la especie y en el caso de la presencia de BLEE y la resistencia a imipenem con sensibilidad a meropenem, estos mecanismos pudieron darse debido a la coexistencia de especies dentro de los tanques y pozos de agua, en conjunción con un mal mantenimiento, favoreciéndose así la transferencia de resistencia 17,18,20,23.
- En los aislados de Pseudomonas aeruginosa se detectó resistencia a cefalosporinas de tercera y cuarta generación y producción de betalactamasas AmpC, este hallazgo se puede comparar con investigaciones como la de Guerrero et al, que detectó 80% de presencia de Pseudomonas aeruginosa en muestras de agua potable 25 y la de Suman et al, que halló Pseudomonas aeruginosa resistente a gentamicina en muestras de agua de servicios de hemodiálisis 26.
Referente a la resistencia de los aislados de Pseudomonas aeruginosa a cefalosporinas de tercera generación, dicha resistencia se debería a las betalactamasas AmpC cromosómica inductible y en cuanto a la producción de AmpC, la presencia de este mecanismo de resistencia es considerada una característica intrínseca de la especie 11.
- Otro hallazgo interesante, es que en algunos de los aislados de Complejo Enterobacter cloacae y Citrobater freundii se constató sensibilidad cefepime acompañada de resistencia a ceftazidima, hecho que podría indicar presencia de enzimas tipo OXA, lo cual se asemeja a los resultados de estudios como el Mahmoud et al, que halló Escherichia coli productora de carbapenemasas OXA-48 en muestras de agua potable 27 y como el de Lépesová et al, que aisló Citrobacter freundii productora de betalactamasas OXA en muestras de agua residual de hospitales 7.
La presunta presencia de OXA, podría deberse al traspaso de genes por mecanismos plásmidicos, dado a que este fenómeno es bastante frecuente entre las distintas especies de Enterobacterales 28.
- Adicionalmente, se hallaron dos aislados MDR, de las especies Complejo Enterobacter cloacae, hallazgo que coincide con otros trabajos que aislaron Enterobacter spp. resistente en muestras de agua potable y de agua de efluentes hospitalarios 7,15,29.
Con respecto a la detección de aislados MDR, esto podría haberse dado por transferencia genética, dado a que hay evidencia de que tanto los Gram (-) fermentadores como los no fermentadores, pueden transferir sus mecanismos de resistencia 23,28.
Los principales motivos por los cuales se habría dado el crecimiento de bacterias resistentes, radican en la fuente de suministro del agua. En las muestras cuya fuente fue pozos artesianos, el tratamiento para potabilizar el agua suele ser realizado por personal del establecimiento de salud, muchas veces sin un debido adiestramiento y/o actualización respecto a los productos y a la frecuencia con la cual se debería hacer el tratamiento.
En las muestras de agua cuya fuente fue tanques de agua potable, los tanques estaban abastecidos por empresas de servicio público, con una adecuada instrucción respecto a los tratamientos aplicados, pero se cometerían errores en la desinfección y en el mantenimiento del tanque, sobre todo porque se acostumbra utilizar únicamente productos a base de cloro y el desagüe con limpieza se estaría efectuando con una frecuencia inferior a la recomendada.
Cabe destacar, que los reservorios como tanques y pozos con agua con un pobre saneamiento se convierten en un medio ideal para la proliferación de microorganismos. Puesto que la formación de biofilm por parte de las especies del género Pseudomonas puede disminuir las concentraciones de cloro y puede propiciar el mal flujo del agua en las conexiones, facilitándose así la acumulación de microorganismos de varias especies en los ductos o cañerías de distribución del agua, dando como resultado una conjunción de factores que predisponen la transferencia horizontal de genes de resistencia 5,28,29.
Considerando los hallazgos de este estudio, aconsejamos que se optimicen las técnicas de tratamiento del agua y de mantenimiento de los reservorios que proveen de agua a los establecimientos sanitarios, para así lograr erradicar a las bacterias resistentes y prevenir posibles brotes infecciosos en los centros estudiados















